위부토신
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
위부토신은 tRNAPhe의 37번째 위치에서 발견되는 초변형된 뉴클레오시드이다. 빵 효모를 모델로 하여 위부토신의 생합성 경로가 밝혀졌으며, TRM5, TYW1, TYW2, TYW3, TYW4 효소들이 관여한다. 위부토신은 tRNA의 리딩 프레임 유지, 안티코돈 루프 구조 안정화에 중요한 역할을 하며, 고세균과 진핵생물에서 보존되지만 세균에서는 발견되지 않는다. 37번 위치의 위부토신 변형은 페닐알라닌 코돈의 특성과 리보솜 슬립과의 관련성을 시사하며, 프레임 시프트 잠재력과 관련하여 세포의 자체 조절 메커니즘으로 프레임 시프트가 사용될 수 있음을 보여준다.
더 읽어볼만한 페이지
- 뉴클레오사이드 - 아데노신
아데노신은 다양한 생리적 과정을 조절하는 내인성 퓨린 뉴클레오사이드로, 심실상 빈맥 치료, 심장 스트레스 검사 보조제, 혈관 확장, 항염증, 항바이러스 효과 연구 등에 사용되며, 뇌에서 수면을 유도하고 주름 개선 화장품 성분으로도 활용된다. - 뉴클레오사이드 - 구아노신
구아노신은 옅은 짠맛이 나는 흰색 결정성 분말로, mRNA 스플라이싱에 관여하고 항바이러스제 및 항 HIV 약물과 유사한 구조를 가지며, 이자, 클로버, 커피 등에서 발견된다. - 하이드록시메틸 화합물 - 아데노신
아데노신은 다양한 생리적 과정을 조절하는 내인성 퓨린 뉴클레오사이드로, 심실상 빈맥 치료, 심장 스트레스 검사 보조제, 혈관 확장, 항염증, 항바이러스 효과 연구 등에 사용되며, 뇌에서 수면을 유도하고 주름 개선 화장품 성분으로도 활용된다. - 하이드록시메틸 화합물 - 블레오마이신
블레오마이신은 DNA 가닥을 절단하여 암을 치료하는 항암제로, 다양한 암에 효과가 있으며 주사 또는 연고 형태로 투여되지만, 발열, 발진, 폐섬유증 등의 부작용이 있을 수 있다.
| 위부토신 - [화학 물질]에 관한 문서 | |
|---|---|
| 일반 정보 | |
 | |
| IUPAC 이름 | 메틸 (2S)-4-(3,4′′-디메틸-3H-이미다조[1′′,2′′:1,2]이노신-5′′-일)-2-[(메톡시카르보닐)아미노]부타노아트 |
| 계통 이름 | 메틸 (2S)-4-{3-[(2R,3R,4S,5R)-3,4-다이하이드록시-5-(하이드록시메틸)옥솔란-2-일]-4,6-다이메틸-9-옥소-4,9-다이하이드로-3H-이미다조[1,2-a]퓨린-7-일}-2-[(메톡시카르보닐)아미노]부타노아트 |
| 다른 이름 | yW |
| 약어 | yW |
| CAS 등록번호 | 55196-46-8 |
| UNII | 5PCY5AS87Q |
| ChEBI | 46574 |
| ChemSpider ID | 28184605 |
| InChI | 1S/C21H28N6O9/c1-9-11(6-5-10(19(32)34-3)24-21(33)35-4)27-17(31)13-16(25(2)20(27)23-9)26(8-22-13)18-15(30)14(29)12(7-28)36-18/h8,10,12,14-15,18,28-30H,5-7H2,1-4H3,(H,24,33)/t10-,12+,14+,15+,18+/m0/s1 |
| 표준 InChIKey | QAOHCFGKCWTBGC-QHOAOGIMSA-N |
| SMILES | Cc1c(n2c(=O)c3c(n(c2n1)C)n(cn3)[C@H]4[C@@H]([C@@H]([C@H](O4)CO)O)O)CC[C@@H](C(=O)OC)NC(=O)OC |
| PubChem | 14135916 |
| 속성 | |
| 분자식 | C21H28N6O9 |
| 위험성 | |
| 추가 정보 | |
| IUPAC 이름 (대안) | 7-{(3S)-4-메톡시-3-[(메톡시카르보닐)아미노]-4-옥소부틸}-4,6-다이메틸-3-(β-D-리보푸라노실)-3,4-다이하이드로-9H-이미다조[1,2-a]퓨린-9-온 |
2. 생합성 경로
''S. cerevisiae'' 모델을 이용하여 위부토신의 생합성 경로가 제안되었다.[4] 이 과정은 페닐알라닌 tRNA의 구아노신(G37) 위치에서 시작되는 다단계 효소 반응을 통해 진행된다.[4]
또한, 위부토신은 화학적으로도 합성이 가능하다.[5][6]
2. 1. 주요 효소 및 반응 단계
''S. cerevisiae'' 모델을 사용하여 위부토신의 생합성 경로가 제안되었다. 이 경로는 여러 단계의 효소 반응을 거친다.[4]생합성의 첫 번째 단계는 N1-메틸전달효소인 TRM5가 페닐알라닌 tRNA의 G37 위치를 메틸화하여 m1G37으로 전환시키는 것이다. 이후 m1G37은 효소 TYW1의 기질로 작용한다. TYW1은 플라빈 모노뉴클레오타이드(FMN)를 보조 인자로 사용하고, 피루브산을 C-3 공급원으로 하여 위부토신의 삼환식 코어를 형성한다. 다음으로 효소 TYW2는 S-아데노실메티오닌(Ado-Met)으로부터 α-아미노-α-카르복시프로필 그룹을 yW-187의 C-7 위치 측쇄로 전달하여 yW-86을 형성한다. 효소 TYW3는 yW-86의 N-4 위치를 메틸화하여 yW-72를 생성한다. TYW4는 Ado-Met 의존성 카르복시메틸전달효소로, yW-72의 α-카르복시 그룹을 메틸화하여 yW-57을 생성한다. 마지막 단계에서는 TYW4의 두 번째 기능 또는 아직 확인되지 않은 다른 인자에 의해 yW-58 측쇄의 α-아미노 그룹이 메톡시카르보닐화되어 최종적으로 위부토신이 생성된다.[4]
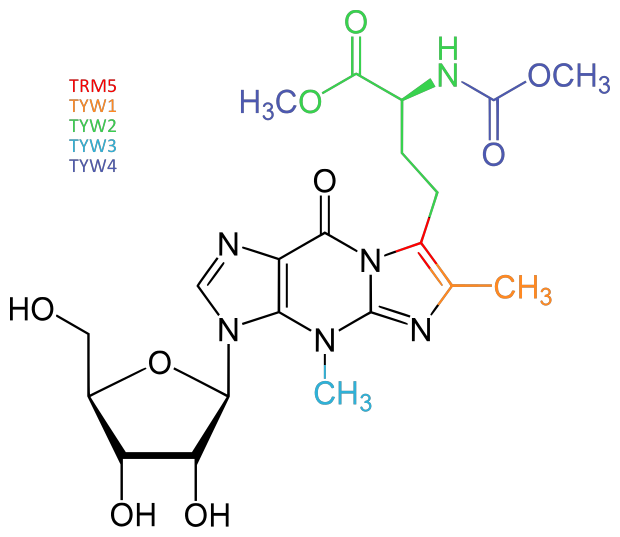
3. tRNA에서의 역할 및 초변형
위부토신(Wybutosine)과 같은 비자연적인 뉴클레오시드는 tRNA 내에서 초변형(hypermodification) 과정을 거쳐 중요한 기능을 수행한다. 특히 tRNAPhe의 37번 위치에서 일어나는 위부토신 초변형은 번역의 정확성을 유지하는 데 핵심적인 역할을 한다.[7] 이러한 변형은 해독틀 유지를 돕고[7] 안티코돈 루프 구조를 안정화하는 데 기여하며,[8] 위부토신이 없을 경우 틀이동 돌연변이가 증가하는 것으로 알려져 있다.
1960년대와 1970년대 연구를 통해 tRNA의 특정 위치, 특히 34번과 37번 위치의 변형이 번역 정확성에 중요하다는 사실이 밝혀졌다. 대부분의 생물 종에서 이 위치들은 변형된 뉴클레오타이드를 가지고 있다. 위부토신과 그 유도체들은 특이하게 37번 위치에서만 발견되는데, 이는 페닐알라닌 코돈(UUU, UUC)의 특성 및 리보솜 슬립 현상과의 관련성을 시사한다.[9] 또한, tRNAPhe의 위부토신 변형은 고세균과 진핵생물에서는 보존되어 나타나지만, 세균에서는 발견되지 않는다는 종 간의 차이점도 확인되었다.[10]
3. 1. 리딩 프레임 유지
위부토신과 같은 비자연적인 뉴클레오시드는 초변형(hypermodification)이라는 결과를 만드는 것으로 여겨진다. 특히 tRNAPhe의 37번 위치에서 일어나는 이러한 초변형은 염기 쌓임 상호작용을 통해 해독틀을 유지하는 데 중요한 역할을 할 수 있다.[7] 위부토신은 큰 방향족 구조를 가지고 있어, 인접한 염기인 A36과 A38과의 쌓임 상호작용을 강화시킨다. 이는 안티코돈의 유연성을 제한하여 정확한 코돈 해독을 돕는다.[8] 만약 tRNAPhe에 위부토신이 없으면 틀이동 돌연변이 현상이 증가하는 것으로 관찰되었다. 일반적으로 tRNA의 37번 위치에 있는 변형된 염기는 안티코돈 루프 구조를 안정적으로 유지하고 열린 상태로 만들어, 인접한 뉴클레오티드와의 원치 않는 염기쌍 형성을 막고 해독에 적합한 구조를 만드는 데 기여한다. tRNAPhe의 위부토신 변형은 고세균과 진핵생물에서는 보존되어 나타나지만, 세균에서는 발견되지 않는다.1960년대와 1970년대 연구에서는 여러 돌연변이가 번역 정확성에 문제를 일으킬 수 있음을 지적했다. 번역 정확성에 관여하는 메커니즘을 더 깊이 연구한 결과, tRNA의 34번과 37번 위치의 변형이 중요하다는 사실이 밝혀졌다. 생물 종에 관계없이 tRNA의 이 두 위치는 거의 항상 변형되어 있다. 위부토신과 그 유도체들이 오직 37번 위치에서만 발견된다는 점은, 이것이 페닐알라닌 코돈(UUU, UUC)의 특성 및 리보솜 슬립 현상에 대한 선호도와 관련 있을 수 있음을 시사한다.[9] 이는 37번 위치에서의 tRNAPhe 변형 정도가 게놈 내 다유리딘(polyuridine) 미끄러운 서열의 양과 상관관계가 있을 것이라는 가설로 이어졌다.[10]
3. 2. 안티코돈 루프 구조 안정화
위부토신(Wybutosine)과 같은 뉴클레오시드의 초변형은 tRNAPhe의 37번 위치에서 일어나며, 이는 안티코돈 루프의 구조를 안정화하는 데 중요한 역할을 한다. 구체적으로, 37번 위치의 변형은 큰 방향족기를 통해 인접한 염기인 A36 및 A38과의 염기-스태킹 상호작용을 강화한다.[8] 이 강화된 상호작용은 안티코돈의 유연성을 제한하여 정확한 코돈 인식을 돕고, 결과적으로 리딩 프레임 유지에 기여한다.[7] 만약 tRNAPhe에 위부토신이 없으면 프레임 시프트가 증가하는 것으로 관찰되었다.일반적으로 tRNA의 37번 위치 변형은 안티코돈 루프 구조를 안정적으로 유지하고 열린 형태로 만들어, 해독 과정에 필요한 안티코돈 루프를 형성하는 데 도움을 준다. 동시에 이는 인접한 뉴클레오타이드와의 비정상적인 염기쌍 형성을 방지하는 역할도 한다. 이러한 위부토신 변형은 고세균과 진핵생물에서는 보존되어 발견되지만, 세균에서는 발견되지 않는다.
37번 위치에서 발견되는 위부토신 및 그 유도체들은 페닐알라닌 코돈(UUU, UUC)의 특성과 리보솜 슬립 현상과의 연관성을 시사할 수 있다.[9] 이는 37번 위치에서의 tRNAPhe 변형이 게놈 내 다유리딘(polyuridine) 미끄러운 서열의 양과 관련이 있을 수 있다는 가설로 이어진다.[10]
3. 3. 종 간 차이
tRNAPhe의 37번 위치에서의 변형은 번역 정확성에 중요한 역할을 한다.[9] 종에 관계없이 tRNA의 34번과 37번 위치는 거의 항상 변형된다.[9] 특히 tRNAPhe의 37번 위치에서 발견되는 위부토신 변형은 고세균과 진핵생물에서는 보존되지만, 세균에서는 발견되지 않는다. 위부토신 및 그 다양한 유도체가 37번 위치에서만 발견된다는 사실은 페닐알라닌 코돈(UUU 및 UUC)의 특성과 리보솜 슬립에 대한 선호도를 나타낼 수 있다.[9]4. 번역 정확성 및 프레임 시프트
위부토신(Wybutosine영어)과 같은 초변형된 뉴클레오시드는 tRNAPhe의 37번 위치에서 발견된다. 이 위치에서의 초변형은 큰 방향족기를 통해 인접 염기 A36 및 A38과의 염기 쌓임(base-stacking) 상호작용을 강화시켜 안티코돈(anticodon)의 유연성을 제한하고 해독틀(reading frame) 유지에 중요한 역할을 하는 것으로 여겨진다.[7][8] 일반적으로 tRNA의 37번 위치 변형은 루프 구조를 안정화시키고 인접 뉴클레오티드와의 비정상적인 염기쌍 형성을 방지하여 정확한 해독을 돕는다. tRNAPhe의 위부토신 변형은 고세균과 진핵생물에서는 보존되어 있지만, 세균에서는 발견되지 않는다.
4. 1. 페닐알라닌 코돈 및 리보솜 슬립
1960년대와 1970년대의 연구에서는 많은 돌연변이가 번역 정확성 문제로 이어질 수 있음을 지적했다. 번역 정확성에 관여하는 메커니즘에 대한 추가 연구를 통해 tRNA의 34번과 37번 위치의 변형이 중요하다는 것이 밝혀졌다. 종에 관계없이 tRNA의 이러한 부위는 거의 항상 변형된다.위부토신 및 그 다양한 유도체가 37번 위치에서만 발견된다는 사실은 페닐알라닌 코돈(UUU 및 UUC)의 특성과 리보솜 슬립에 대한 선호도를 나타낼 수 있다.[9] 실제로 tRNAPhe에 위부토신이 없을 경우 프레임 시프트(frameshifting)가 증가하는 것으로 밝혀졌다. 이는 37번 위치에서 tRNAPhe 변형이 게놈에서 발견되는 다유리딘(polyuridine) 미끄러운 서열의 양과 상관관계가 있다는 가정으로 이어졌다.[10]
4. 2. 프레임 시프트 잠재력
위부토신이 프레임 시프트를 방지하는 역할을 하지만, 그 중요성에 대해서는 몇 가지 의문이 제기되어 왔다. 와이부토신(yW)을 이용한 변형 외에도 프레임 시프트를 방지하는 다른 전략들이 존재하기 때문이다. 예를 들어, 초파리에서는 tRNAPhe의 37번 위치에 변형이 없는 반면, 포유류에서는 yW가 변형된다.이러한 가변성을 설명하기 위해 프레임 시프트 잠재력이라는 아이디어가 생겨났다. 이는 세포가 항상 프레임 시프트를 피하려 하기보다는 자체 조절 메커니즘으로 프레임 시프트를 사용한다는 것을 의미한다.[11] 프레임 시프트는 프로그램된 방식으로, 아마도 코딩 다양성을 증가시키기 위해 사용될 수 있다는 점이 시사되었다.
참조
[1]
논문
Biosynthesis of wybutosine, a hyper-modified nucleoside in eukaryotic phenylalanine tRNA
[2]
논문
Wybutosine biosynthesis: Structural and mechanistic overview
2014-12-02
[3]
논문
Crystal Structure of the Radical SAM Enzyme Catalyzing Tricyclic Modified Base Formation in tRNA
2007-10
[4]
서적
Radical SAM Enzymes
[5]
논문
Practical synthesis of wybutosine, the hypermodified nucleoside of yeast phenylalanine transfer ribonucleic acid
[6]
논문
Total synthesis of the hypermodified RNA bases wybutosine and hydroxywybutosine and their quantification together with other modified RNA bases in plant materials
[7]
논문
Posttranscriptional RNA Modifications: playing metabolic games in a cell's chemical Legoland
[8]
논문
Naturally-occurring modification restricts the anticodon domain conformational space of tRNA(Phe)
[9]
논문
Control of catalytic cycle by a pair of analogous tRNA modification enzymes
[10]
논문
Transfer RNA modifications: nature's combinatorial chemistry playground
[11]
논문
Role of a tRNA Base Modification and Its Precursors in Frameshifting in Eukaryotes
2007-09
[12]
논문
Biosynthesis of wybutosine, a hyper-modified nucleoside in eukaryotic phenylalanine tRNA
null
[13]
논문
Crystal structure of the radical SAM enzyme catalyzing tricyclic modified base formation in tRNA
null
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com