세균분류학
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
세균분류학은 세균의 다양성, 역사, 분류, 분석 방법, 그리고 병리학과의 관계를 다루는 학문이다. 세균은 핵막이 없고 단세포이며 이분법으로 분열하는 등 고세균과 공통적인 특징을 공유하며, 계통발생, 대사, 환경, 형태 등의 특성을 기준으로 분류된다. 세균 분류는 안톤 판 레이우엔훅의 현미경 관찰 이후, 형태, 대사, 유전자 분석을 통해 발전해왔으며, 현재는 분자 계통 발생학을 기반으로 16S rRNA 유전자 분석 등이 활용된다. 세균 분류는 국제 원핵생물 명명 규약에 따라 이루어지며, 종 개념은 수평적 유전자 전달과 무성생식으로 인해 복잡하며, 표현형과 유전적 차이를 모두 고려하는 다상 종 정의가 사용된다. 병원성 균주의 경우, 유전적 차이로 인해 진화론적 관점에서 다계통으로 분류되더라도 의학적 중요성 때문에 별개의 종으로 유지되는 경우도 있다.
더 읽어볼만한 페이지
| 세균분류학 | |
|---|---|
| 세균 분류 | |
 | |
| 리네의 분류 | 1735년 |
| 현재의 3역 체계 | 1990년 |
| 분류 계급 | |
| 생물역 | 생물역 |
| 계 | 세균계 |
| 문 | 문 |
| 강 | 강 |
| 목 | 목 |
| 과 | 과 |
| 속 | 속 |
| 종 | 종 |
2. 다양성
세균(세균강, 원핵생물)은 고세균과 함께 많은 공통적인 특징을 공유한다. 이러한 공통점에는 핵막의 부재, 단세포성, 이분법에 의한 분열, 그리고 일반적으로 작은 크기가 포함된다. 다양한 종은 여러 특성을 비교하여 구별할 수 있으며, 이를 통해 식별하고 분류할 수 있다. 예시는 다음과 같다.
- '''계통발생''': 모든 세균은 공통 조상으로부터 유래되어 이후 다양해졌으며, 결과적으로 서로 다른 수준의 진화적 관련성을 갖는다. (세균 문 및 진화 연대표 참조)
- '''대사''': 서로 다른 세균은 서로 다른 대사 능력을 가질 수 있다. (미생물 대사 참조)
- '''환경''': 서로 다른 세균은 고온/저온 및 염분과 같은 서로 다른 환경에서 번성한다. (극한 환경 미생물 참조)
- '''형태''': 세균 사이에는 세포 모양, 그람 염색(지질 이중층 수) 또는 이중층 구성과 같은 많은 구조적 차이가 있다. (세균 세포 형태, 세균 세포 구조 참조)
2. 1. 계통발생
세균(세균강, 원핵생물)은 고세균과 핵막 부재, 단세포성, 이분법에 의한 분열, 작은 크기 등 여러 공통 특징을 공유한다. 모든 세균은 공통 조상으로부터 유래되어 다양하게 진화했으며, 서로 다른 수준의 진화적 관련성을 갖는다. (세균 문 및 진화 연대표 참조)2. 2. 대사
세균(원핵생물)은 고세균과 함께 핵막이 없고, 단세포이며, 이분법으로 분열하고, 일반적으로 크기가 작다는 공통적인 특징을 공유한다. 다양한 종은 여러 특성을 비교하여 구별할 수 있는데, 그중 하나는 대사이다. 서로 다른 세균은 서로 다른 대사 능력을 가질 수 있다. (미생물 대사 참조)2. 3. 환경
세균(원핵생물)은 고세균과 함께 핵막 부재, 단세포성, 이분법에 의한 분열, 그리고 일반적으로 작은 크기와 같은 많은 공통적인 특징을 공유한다. 다양한 종은 여러 특성을 비교하여 구별할 수 있으며, 이를 통해 식별하고 분류할 수 있다. 서로 다른 세균은 고온/저온 및 염분과 같은 서로 다른 환경에서 번성한다. (극한 환경 미생물 참조)2. 4. 형태
세균(원핵생물)은 고세균과 함께 핵막이 없고, 단세포이며, 이분법으로 분열하고, 일반적으로 크기가 작다는 공통적인 특징을 갖는다. 다양한 종은 여러 특성을 비교하여 식별하고 분류할 수 있는데, 그 예시는 다음과 같다.- '''계통발생''': 모든 세균은 공통 조상으로부터 유래되어 다양해졌으며, 서로 다른 수준의 진화적 관련성을 갖는다. (세균 문 및 진화 연대표 참조)
- '''대사''': 서로 다른 세균은 서로 다른 대사 능력을 가질 수 있다. (미생물 대사 참조)
- '''환경''': 서로 다른 세균은 고온/저온 및 염분과 같은 서로 다른 환경에서 번성한다. (극한 환경 미생물 참조)
- '''형태''': 세균 사이에는 세포 모양, 그람 염색(지질 이중층 수) 또는 이중층 구성과 같은 많은 구조적 차이가 있다. (세균 세포 형태, 세균 세포 구조 참조)
3. 역사
3. 1. 최초의 관찰
안톤 판 레이우엔훅은 1676년 자신이 직접 설계한 단렌즈 현미경을 사용하여 세균을 처음 관찰했다.[3] 그는 세균을 "미생물"이라고 불렀으며, 관찰 결과를 왕립 학회에 보낸 편지에 게재했다.[4][5][6] 세균의 초기 기술에는 O. F. 뮐러가 명명한 ''Vibrio'' 와 ''Monas''(1773, 1786)가 있으며, 당시에는 섬모충류로 분류되었다. H. F. 링크가 명명한 ''Polyangium''(1809)은 현재까지 인정받는 최초의 세균이며, 비지오가 명명한 ''Serratia''(1823), 에렌베르크가 명명한 ''Spirillum'', ''Spirochaeta'', ''Bacterium''(1838) 등이 있다.[7][8] 1838년 에렌베르크가 속으로 도입한 ''Bacterium''[9]은 막대 모양의 세포를 포괄하는 용어가 되었다.[7]3. 2. 초기 분류
세균은 처음에는 식물로 분류되어 ''Schizomycetes'' 강을 구성했으며, 이는 ''Schizophyceae'' (남조류/''Cyanobacteria'')와 함께 ''Schizophyta'' 문을 형성했다.[10]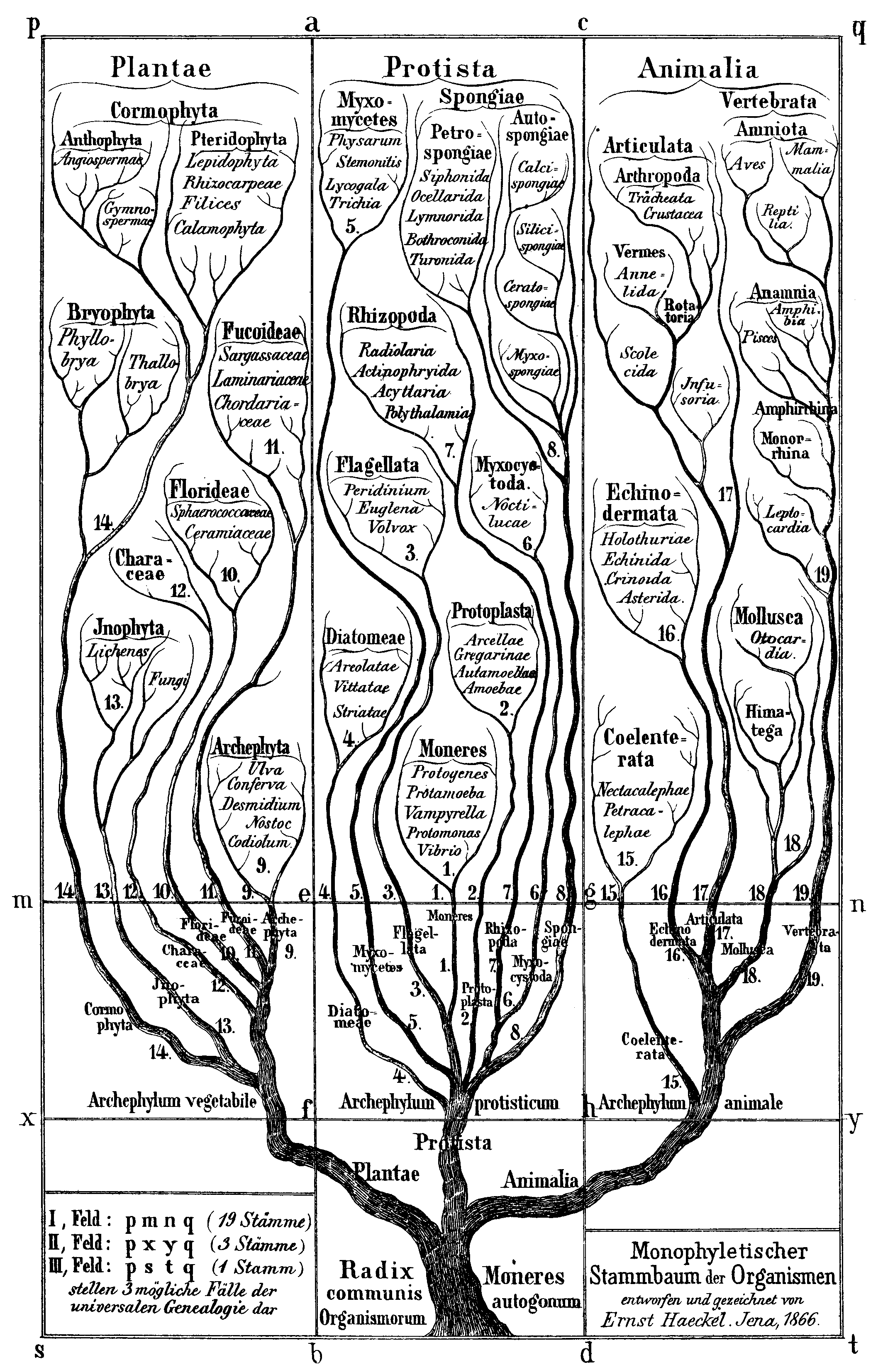
에른스트 헤켈은 1866년에 이 그룹을 ''Moneres'' 문(μονήρης에서 유래: 단순한)과 ''Protista''계에 배치하고 이를 완전히 구조가 없고 균질한 유기체로 정의했으며, 이는 플라스마 덩어리만으로 구성되었다.[11] 그는 이 문을 die Gymnomonerende (피막 없음)과 die Lepomonerende (피막 있음)의 두 그룹으로 나누었다.[11] Gymnomoneren에는 ''Vibrio'' – 1854년에 처음 기술된 콤마 모양 세균 속[12], ''Bacterium'' – 1828년에 처음 기술된 막대 모양 세균 속, ''Bacillus'' – 1835년에 처음 기술된 포자 형성 막대 모양 세균 속[13], ''Spirochaeta'' – 얇은 나선형 세균으로 1835년에 처음 기술[13], ''Spirillum'' – 나선형 세균으로 1832년에 처음 기술[14] 등이 포함되었다.
페르디난트 콘의 분류(1872)는 19세기에 영향력이 있었으며, 여섯 개의 속(''Micrococcus'', ''Bacterium'', ''Bacillus'', ''Vibrio'', ''Spirillum'', ''Spirochaeta'')을 인정했다.[7]
이 그룹은 나중에 에두아르 샤톤에 의해 ''원핵생물''로 재분류되었다.[16]
''Cyanobacteria''(남조류)의 분류는 조류인지 세균인지에 대해 논쟁이 있었다.[11] 1905년, 에르빈 F. 스미스는 33개의 유효한 세균 속 이름과 150개 이상의 무효한 이름을 인정했고,[17] 1913년 부이민의 연구에서는[18] ''Bacteria''의 모든 종이 특정 속에 속해야 한다고 결론지었다.
콘[19]은 네 개의 족(Spherobacteria, Microbacteria, Desmobacteria, Spirobacteria)을 인정했다. 스타니에르와 반 닐[20]은 Myxophyta와 Schizomycetae의 두 문을 가진 Monera계를 인정했으며, 후자는 여러 강을 포함했다. 비세트[21]는 1강과 4목을 구별했다. 발터 미굴라의 시스템[22]은 당시 가장 널리 받아들여진 시스템이었으며, 형태학만을 기반으로 했다. 오를라-옌센[23]은 두 목을 설정했다. 베르기 등[24]은 미국 세균학회 위원회의 1920년 최종 보고서를 따르는 분류법을 제시했다.
그러나 보이는 특징이 부족하여 속의 재분류가 빈번했고, 이는 1915년 로버트 얼 부캐넌에 의해 요약되었다.[25] 1947년의 세균학적 규약 제1판은 몇 가지 문제를 해결했다.[30]
A. R. 프레보의 시스템[31][32]은 4개의 아문과 8개의 강을 가지고 있었다. 그람 염색 결과는 가장 일반적으로 분류 도구로 사용되었으며, 분자 계통 발생학이 등장하기 전까지 원핵생물계(''Kingdom ''Prokaryota'')는 4개의 문(''Gracilicutes'', ''Firmacutes'', ''Mollicutes'', ''Mendocutes'')으로 나뉘었다.[33] 이 분류 체계는 베르기 세균 분류 매뉴얼에서 공식적으로 사용된다.[34]
3. 3. 분자 시대
칼 우즈(Carl Woese)는 세균, 고세균, 진핵생물이 조상 생물 집단에서 초기에 분화된 별개의 계통을 나타낸다고 주장했다.[37][38] 그러나 소수의 생물학자들은 고세균과 진핵생물이 세균 집단에서 유래했다고 주장한다.[39] 바이러스와 고세균은 약 20억 년 전에 관계를 시작했으며, 이들 집단 간에 공진화가 일어났을 가능성이 있다고 여겨진다.[40] 세균과 고세균의 마지막 공통 조상이 고온성일 가능성이 있으며, 이는 더 낮은 온도가 고세균의 관점에서 "극한 환경"이며, 더 시원한 환경에서 사는 생물은 나중에 나타났을 가능성을 제기한다.[41] 고세균과 세균은 진핵생물만큼 서로 관련이 없기 때문에, ''원핵생물'''s의 유일하게 남은 의미는 "진핵생물이 아님"이며, 그 가치를 제한한다.[42]방법론이 개선되면서, 메탄 생성 세균이 매우 다르다는 것이 분명해졌다.[43] 칼 우즈는 세 개의 주요 계통, 즉 ''고세균'', ''진정세균'', 그리고 현재 ''진핵생물''의 핵-세포질 구성 요소로 대표되는 ''원핵생물''을 확인했다.[44] 이 계통들은 생명을 3개의 도메인, 즉 ''진핵생물'', ''고세균'', 그리고 ''세균''으로 나누는 도메인(라틴어로 ''regio'')의 지위로 공식화되었다.[2]
2023년, ''원핵생물 코드''는 원핵생물 명명법에 도메인과 계의 지위를 추가했다. ''세균''과 ''고세균''의 이름은 이러한 새로운 규칙을 사용하는 Oren과 Goker의 출판물을 따른 유효하게 출판된 분류군이다.[45] 1987년 칼 우즈는 ''진정세균''을 16S 리보솜 RNA (SSU) 염기서열을 기반으로 11개의 문으로 나누었으며, 여기에 몇 가지가 더해져 오늘날까지 사용되고 있다.[46][47]
오렌과 괴커는 문/계보다 상위 분류로 여러 강을 유효하게 발표했다:[45]
- 도메인 세균
- * 강 바실라티 (문 Firmicutes와 'Tenericutes', 'Terrabacteria', 'Terrabacterida', 일부 단층 세균, 아강 'Unibacteria' 일부)
- * 강 푸소박테리아티 ('Fusobacterida')
- * 강 슈도모나다티 (문 Gracilicutes, 'Hydrobacteria', 'Hydrobacterida' 및 'Aquificida', 이중 세균, 아강 'Negibacteria')
- * 강 테르모토가티 ('Thermotogida')
- 도메인 고세균
- * 강 메탄박테리아티 (문 'Euryarchaeota' sensu lato, 'Euryarchaeida')
- * 강 나노브델라티 (DPANN 상문)
- * 강 테르모프로테아티 (TACK 상문, 'Crenarchaeida')
- * 강 프로메테아르케아티 (아스가르드, 이마치 등이 나중에 제안)
3역 체계가 널리 받아들여지고 있지만,[48] 일부 저자는 다양한 이유로 이에 반대해 왔다.
3역 체계에 반대하는 저명한 과학자 중 한 명은 토마스 카발리에-스미스로, 그는 ''고세균''과 ''진핵생물''(''네오무라'')이 그람 양성 세균(''포시박테리아'')에서 유래하며, 이는 다시 그람 음성 세균(''네기박테리아'')에서 유래한다고 제안했다.[49][50]
제임스 레이크는 리보솜 구조를 기반으로 5개의 주요 왕국 체계(진핵생물, 에오사이트, 메탄세균, 할로박테리아, 유세균) 및 4개의 주요 왕국 체계(진핵생물, 에오사이트, 메탄세균, 광세균)를 제안했는데, 세균은 광합성(광세균), 메탄 생성 (메탄세균), 황 호흡 (에오사이트)의 3가지 주요 생화학적 혁신에 따라 분류되었다.[61][62][63] 그는 또한 그람 음성 세균이 2개의 그람 양성 세균 간의 공생에서 발생했다는 증거를 발견했다.[64]
4. 분류
분류는 계통 발생 및 표현형을 기반으로 유기체를 점진적으로 더 포괄적인 그룹으로 묶는 것이며, 명명법은 유기체 명명에 대한 공식적인 규칙을 적용하는 것이다.[106]
4. 1. 명명법
원핵생물에 대한 공식적이고 완전한 분류는 없지만, 원핵생물에 부여된 명칭(학명)은 국제 원핵생물 명명 규약(Prokaryotic Code)에 의해 규제된다.[65] 분류학적 명칭은 종과 아종의 형용사를 제외하고 첫 글자를 대문자로 하여 이탤릭체로 표기하거나 손으로 쓸 때는 밑줄을 긋는다. 동물학에서 흔히 사용되는 동형이명은 허용되지 않으며, 동물학, 식물학 또는 균학에 사용된 분류군명은 세균에 재사용될 수 없다.명명법은 분류군의 명칭을 규율하는 규칙과 관례의 집합이다.[103] 세균의 경우, 유효한 명칭은 라틴어 또는 신라틴어 명칭을 가져야 하며 기본 라틴 문자(w와 j 포함)만 사용할 수 있다.[104] 하이픈, 악센트 및 기타 문자는 허용되지 않으며 올바르게 음역해야 한다.[104] 고대 그리스어는 그리스 문자로 작성되었으므로 라틴 문자로 음역해야 한다.
복합어를 만들 때, 후자가 모음으로 시작하는 경우 연결 모음이 추가되지 않는다는 점을 제외하고는, 뒤따르는 단어에 관계없이 앞선 단어의 기원에 따라 연결 모음이 필요하다. 첫 번째 복합어가 라틴어인 경우 연결 모음은 -i-이고, 첫 번째 복합어가 그리스어인 경우 연결 모음은 -o-이다.[114]
''원핵생물''(세균과 고세균)의 경우, 2024년까지는 계급이 사용되지 않았다. 계급 범주는 2023년 11월 세균학 규약에 포함되었으며, 처음 4개의 제안(''바실라티'', ''푸소박테리아티'', ''슈도모나다티'', ''테르모토가티'')는 2024년 1월에 유효하게 발표되었다. 새로운 종이나 수정된 종이 새로운 계급에 배치되면, 세균학 규약의 규칙 9에 따라 이름은 기준 속의 이름에 적절한 접미사를 추가하여 형성된다.
| 계급 | 접미사 | 예시 |
|---|---|---|
| 속 | 엘루시미크로비움(Elusimicrobium) | |
| 아족 (사용 안 함) | (엘루시미크로비이나이) | |
| 족 (사용 안 함) | (엘루시미크로비이에) | |
| 아과 | (엘루시미크로비오이데) | |
| 과 | 엘루시미크로비아케아이 | |
| 아목 | (엘루시미크로비네) | |
| 목 | 엘루시미크로비알레스 | |
| 아강 | (엘루시미크로비데) | |
| 강 | 엘루시미크로비아 | |
| 문 | 엘루시미크로비오타 | |
| 계 | 엘루시미크로비아티 | |
2021년까지 문은 세균 명명 규약의 적용을 받지 않아 비공식적으로 명명되었다.[110] 2021년에 세균 명명 규약에 이름을 포함하기로 결정했다.[111] 새로운 규칙에 따라 문의 이름은 기준 속에서 파생된다.
- ''산성세균문''(Acidobacteriota)(''산성세균''(Acidobacterium)에서 유래)
- ''방선균문''(Actinomycetota)(''방선균''(Actinomyces)에서 유래)
- ''온천세균문''(Aquificota)(''온천세균''(Aquifex)에서 유래)
- ''아르마티모나스문''(Armatimonadota)(''아르마티모나스''(Armatimonas)에서 유래)
- ''아트리박테르문''(Atribacterota)(''아트리박테르''(Atribacter)에서 유래)
- ''바실러스문''(Bacillota)(''바실러스''(Bacillus)에서 유래)
- ''세균문''(Bacteroidota)(''세균''(Bacteroides)에서 유래)
- ''발네올라문''(Balneolota)(''발네올라''(Balneola)에서 유래)
- ''와동균문''(Bdellovibrionota)(''와동균''(Bdellovibrio)에서 유래)
- ''칼디세리쿰문''(Caldisericota)(''칼디세리쿰''(Caldisericum)에서 유래)
- ''칼디트릭스문''(Calditrichota)(''칼디트릭스''(Caldithrix)에서 유래)
- ''캄필로박터문''(Campylobacterota)(''캄필로박터''(Campylobacter)에서 유래)
- ''클라미디아문''(Chlamydiota)(''클라미디아''(Chlamydia)에서 유래)
- ''녹세균문''(Chlorobiota)(''녹세균''(Chlorobium)에서 유래)
- ''녹유연세균문''(Chloroflexota)(''녹유연세균''(Chloroflexus)에서 유래)
- ''크리시게네스문''(Chrysiogenota)(''크리시게네스''(Chrysiogenes)에서 유래)
- ''코프로써모박테르문''(Coprothermobacterota)(''코프로써모박테르''(Coprothermobacter)에서 유래)
- ''철환원세균문''(Deferribacterota)(''철환원세균''(Deferribacter)에서 유래)
- ''데아노코쿠스문''(Deinococcota)(''데아노코쿠스''(Deinococcus)에서 유래)
- ''딕티오글로무스문''(Dictyoglomota)(''딕티오글로무스''(Dictyoglomus)에서 유래)
- ''엘루시미크로비움문''(Elusimicrobiota)(''엘루시미크로비움''(Elusimicrobium)에서 유래)
- ''섬유세균문''(Fibrobacterota)(''섬유세균''(Fibrobacter)에서 유래)
- ''푸소박테리움문''(Fusobacteriota)(''푸소박테리움''(Fusobacterium)에서 유래)
- ''제마티모나스문''(Gemmatimonadota)(''제마티모나스''(Gemmatimonas)에서 유래)
- ''이그나비박테리움문''(Ignavibacteriota)(''이그나비박테리움''(Ignavibacterium)에서 유래)
- ''키리티마티엘라문''(Kiritimatiellota)(''키리티마티엘라''(Kiritimatiella)에서 유래)
- ''렌티스파에라문''(Lentisphaerota)(''렌티스파에라''(Lentisphaera)에서 유래)
- ''미코플라스마문''(Mycoplasmatota)(''미코플라스마''(Mycoplasma)에서 유래)
- ''점액세균문''(Myxococcota)(''점액세균''(Myxococcus)에서 유래)
- ''니트로스피나문''(Nitrospinota)(''니트로스피나''(Nitrospina)에서 유래)
- ''니트로스피라문''(Nitrospirota)(''니트로스피라''(Nitrospira)에서 유래)
- ''플랑크토마이세스문''(Planctomycetota)(''플랑크토마이세스''(Planctomyces)에서 유래)
- ''슈도모나스문''(Pseudomonadota)(''슈도모나스''(Pseudomonas)에서 유래)
- ''로도써머스문''(Rhodothermota)(''로도써머스''(Rhodothermus)에서 유래)
- ''스피로헤타문''(Spirochaetota)(''스피로헤타''(Spirochaeta)에서 유래)
- ''시너지스테스문''(Synergistota)(''시너지스테스''(Synergistes)에서 유래)
- ''써모데술포박테리움문''(Thermodesulfobacteriota)(''써모데술포박테리움''(Thermodesulfobacterium)에서 유래)
- ''써모미크로비움문''(Thermomicrobiota)(''써모미크로비움''(Thermomicrobium)에서 유래)
- ''써모토가문''(Thermotogota)(''써모토가''(Thermotoga)에서 유래)
- ''베루코미크로비움문''(Verrucomicrobiota)(''베루코미크로비움''(Verrucomicrobium)에서 유래)
몇몇 종들은 발견자 또는 미생물학 분야의 유명 인물의 이름을 따서 명명되었다.[112][113] 속명은 사람에서 유래된 모든 이름이 여성 주격으로 표시되어야 하며, 이름에 따라 어미를 -a 또는 축소형 -ella로 변경한다.[114] 종명은 형용사 형태로 변환하거나(속명의 성에 따라 -nus(남성), -na(여성), -num(중성)을 추가) 라틴어화된 이름의 소유격으로 변환할 수 있다.[114]
많은 종(종명)은 그들이 존재하거나 발견된 장소의 이름을 따서 명명된다.[114] 종의 약자는 '''sp.'''(복수 '''spp.''')이며, 속명 뒤에 사용하여 해당 속의 종을 나타낸다. 세균이 알려져 있고 잘 연구되었지만 배양이 불가능한 경우, 이름에 ''Candidatus''라는 용어가 부여된다. 바소님은 새로운 조합의 원래 이름이다. 동의어는 분류군의 대체 이름, 즉 분류군이 두 번 잘못 기술된 경우이다. 분류군이 이전될 때 신 조합 (comb. nov.) 또는 신명 (nom. nov.)이 된다.
4. 2. 분류 체계
분류군이 늘어나면서 컴퓨터 기반의 분류 시스템이 개발되었다. 초창기 비(非) 네트워크 식별 소프트웨어는 Edwards(1978), Kellogg(1979), Schindler, Duben, and Lysenko(1979), Beers and Lockhard(1962), Gyllenberg(1965), Holmes and Hill(1985), Lapage et al.(1970), Lapage et al.(1973) 등이 널리 사용되었다.[66]오늘날, 정확하게 기술된 분류군은 Bergey's Manual of Systematic Bacteriology에서 검토되며, 이는 종의 식별을 돕는 것을 목표로 하며 최고의 권위로 간주된다.[34] 세균 및 고균의 분류 개요(TOBA)의 온라인 버전은 [https://web.archive.org/web/20130502073028/http://www.taxonomicoutline.org/]에서 확인할 수 있다.
''명명법상 유효한 원핵생물 이름 목록(LPSN)''은 현재 2천 개 이상의 공인된 이름과 해당 참고 문헌, 어원 및 다양한 참고 사항을 포함하는 온라인 데이터베이스이다.[67]
몇몇 다른 명명법 체계가 ''원핵생물 명명 규약'' 시스템의 단점을 보완하기 위해 제안되었다. SeqCode는 원핵생물 명명법을 관리하는 별도의 규칙 집합으로, 배양 균주를 기준 물질로 사용하는 대신, 게놈 염기서열을 사용하며 자체 이름 데이터베이스를 유지 관리한다.[68] GTDB는 마커 유전자 계통 발생학과 자체 규칙을 기반으로 하는 원핵생물 명명법을 제공하는 컴퓨터 데이터베이스이며, 일부 결과는 ''원핵생물 명명 규약'' 및 ''SeqCode'' 시스템에 적용되었다.[69][70]
다음 시스템은 보다 ''임시적인'' 규칙에 따라 분류학 데이터베이스를 제공한다.
- GenBank 분류학 브라우저는 큐레이터가 상당한 변경을 가한 GenBank 제출에 사용된 모든 분류군을 포함하며, 원핵생물에만 국한되지 않는다.[71]
- 'The All-Species Living Tree' Project (SILVA LTP)는 자체 유형의 분류학으로 주석이 달린 16S rRNA 염기서열 데이터베이스를 제공한다. 리보솜 데이터베이스 프로젝트(RDP)는 이와 유사한 프로젝트이다.[72]
- Greengenes는 버전 2부터 생명체의 웹 계통 발생학과 16S 데이터, GTDB 및 LTP의 이름을 결합한 시스템이다. 나무에 배치된 16S V4 영역 염기서열을 제공한다.[73]
- Open Tree of Life는 계통 발생적이며 원핵생물에 국한되지 않는다.
4. 3. 새로운 종의 기술
''계통 박테리아학 국제 저널/계통 및 진화 미생물학 국제 저널''(IJSB/IJSEM)은 새로운 원핵생물 분류군을 발표하는 공식적인 국제 포럼 역할을 하는 동료 심사를 거치는 저널이다.[78] 만약 어떤 종이 다른 동료 심사 저널에 게재된 경우, 저자는 적절한 설명과 함께 IJSEM에 요청서를 제출할 수 있으며, 내용이 정확하다면 새로운 종은 IJSEM의 유효성 검사 목록에 등재된다.[78] 새로운 종을 묘사하기 위한 최소 기준은 해당 종이 속한 분류군에 따라 달라진다.[78]Candidatus는 분류학적 명칭의 구성 요소로, 세균 배양 보존소에서 유지할 수 없는 세균에 사용된다. 배양이 불가능한 생물에 대한 임시 분류학적 지위이다. 예를 들어 "Candidatus Pelagibacter ubique"가 있다. 세균이 알려져 있고 잘 연구되었지만 배양이 불가능한 경우, 이름에 ''Candidatus''라는 용어가 부여된다.
종의 약자는 '''sp.'''(복수 '''spp.''')이며, 속명 뒤에 사용하여 해당 속의 종을 나타낸다. 종으로 아직 기술되지 않았거나, 종을 식별하기 위한 충분한 검사가 수행되지 않아 종이 알려지지 않은 속의 균주를 나타내는 데 자주 사용된다. 예를 들어 ''Halomonas'' sp. GFAJ-1와 같다.
바소님은 새로운 조합의 원래 이름, 즉 분류가 변경되기 전에 분류군에 부여된 첫 번째 이름이다. 동의어는 분류군의 대체 이름, 즉 분류군이 두 번 잘못 기술된 경우이다. 분류군이 이전될 때 신 조합 (comb. nov.) 또는 신명 (nom. nov.)이 된다.
4. 4. 종 개념
세균은 무성생식을 하며, 대부분 지역주의를 보이지 않으므로("Everything is everywhere영어"), 동물에게 가장 적합한 종의 개념은 전적으로 판단의 문제로 귀결된다.세균 및 고균의 명명된 종의 수(약 13,000개)[79]는 초기 진화, 유전적 다양성, 그리고 모든 생태계에서의 서식을 고려할 때 놀라울 정도로 적다. 이러한 현상의 이유는 세균과 거대 유기체 간의 종 개념의 차이, 순수 배양에서의 성장/특성화의 어려움(''위 참조'') 그리고 광범위한 수평적 유전자 전달로 인한 종의 구분이 모호해지기 때문이다.[80]
가장 일반적으로 받아들여지는 정의는 표현형 및 유전적 차이를 모두 고려하는 다상 종 정의이다.[81] 그러나 종을 구분하는 더 빠른 진단적인 임시 기준은 70% 미만의 DNA-DNA 혼성화이며,[82] 이는 97% 미만의 16S DNA 염기 서열 동일성에 해당한다.[83] 만약 이 기준이 동물 분류에 적용된다면, 영장류목은 단일 종이 될 것이라는 점이 지적되었다.[84] 이러한 이유로, 전체 유전체 서열을 기반으로 한 더욱 엄격한 종 정의가 제안되었다.[85]
- 종의 약자는 '''sp.'''(복수 '''spp.''')이며, 속명 뒤에 사용하여 해당 속의 종을 나타낸다. 종으로 아직 기술되지 않았거나, 종을 식별하기 위한 충분한 검사가 수행되지 않아 종이 알려지지 않은 속의 균주를 나타내는 데 자주 사용된다. 예를 들어 ''Halomonas'' sp. GFAJ-1 – 개방 명명법 참조
- 세균이 알려져 있고 잘 연구되었지만 배양이 불가능한 경우, 이름에 ''Candidatus''라는 용어가 부여된다.
- 바소님은 새로운 조합의 원래 이름, 즉 분류가 변경되기 전에 분류군에 부여된 첫 번째 이름이다.
- 동의어는 분류군의 대체 이름, 즉 분류군이 두 번 잘못 기술된 경우이다.
- 분류군이 이전될 때 신 조합 (comb. nov.) 또는 신명 (nom. nov.)이 된다.
5. 병리학과 계통 발생학의 관계
이상적으로 분류학적 분류는 분류군의 진화적 계통, 즉 계통 발생을 반영해야 한다. 그러나 특히 의학적 관점에서 볼 때, 표현형이 집단 간에 다를 경우 몇 가지 예외가 있다. 다음과 같은 분류의 문제점에 대한 몇 가지 예시가 있다.
; ''Escherichia coli'' (대장균)
감마프로테오박테리아 강의 장내세균과에 속하는 속인 ''시겔라''(''S. dysenteriae'', ''S. flexneri'', ''S. boydii'', ''S. sonnei'') 종은 진화론적 관점에서 보면 ''대장균''(다계통) 종의 균주이지만, 유전적 차이로 인해 병원성 균주의 경우 다른 질병을 유발한다.[86] 혼란스럽게도, 시가 독소를 생성하는 ''대장균'' 균주도 있는데, 이를 STEC이라고 한다.
대장균은 일부 균주가 유전체의 20%만 공유하기 때문에 분류가 제대로 되어 있지 않은 종이다.[87] 매우 다양하기 때문에 더 높은 분류학적 순위를 부여해야 한다.[87] 그러나, 이 종과 관련된 의학적 상태 때문에, 의학적 맥락에서 혼란을 피하기 위해 변경되지 않을 것이다.
; ''Bacillus cereus'' 그룹
''B. cereus 그룹''은 ''바실루스'' 속 종 (Firmicutes 문)에 속하며, B. anthracis, B. cereus, B. thuringiensis, B. mycoides, B. pseudomycoides, B. weihenstephanensis 및 B. medusa를 포함한다.[88] 이들은 99-100% 유사한 16S rRNA 서열을 갖는데, 이는 일반적으로 인용되는 종 구분 기준인 97%를 상회한다.[88] 그럼에도 불구하고, 이들은 의학적 이유(탄저병 등)로 인해 별도의 종으로 유지된다.[88]
; ''Yersinia pestis'' (페스트균)
페스트균(Yersinia pestis)은 사실상 가성결핵균(Yersinia pseudotuberculosis)의 변종이지만, 병원성 섬을 가지고 있어 15,000년에서 20,000년 전에 급격히 다른 병리학적 증상(각각 흑사병과 결핵 유사 증상)을 나타내게 되었다.[89]
5. 1. ''Escherichia coli'' (대장균)
감마프로테오박테리아 강의 장내세균과에 속하는 속인 ''시겔라''(''S. dysenteriae'', ''S. flexneri'', ''S. boydii'', ''S. sonnei'') 종은 진화론적 관점에서 보면 ''대장균''(다계통) 종의 균주이지만, 유전적 차이로 인해 병원성 균주의 경우 다른 질병을 유발한다.[86] 혼란스럽게도, 시가 독소를 생성하는 ''대장균'' 균주도 있는데, 이를 STEC이라고 한다.대장균은 일부 균주가 유전체의 20%만 공유하기 때문에 분류가 제대로 되어 있지 않은 종이다.[87] 매우 다양하기 때문에 더 높은 분류학적 순위를 부여해야 한다.[87] 그러나, 이 종과 관련된 의학적 상태 때문에, 의학적 맥락에서 혼란을 피하기 위해 변경되지 않을 것이다.
5. 2. ''Bacillus cereus'' 그룹
''B. cereus 그룹''은 ''바실루스'' 속 종 (Firmicutes 문)에 속하며, B. anthracis, B. cereus, B. thuringiensis, B. mycoides, B. pseudomycoides, B. weihenstephanensis 및 B. medusa를 포함한다.[88] 이들은 99-100% 유사한 16S rRNA 서열을 갖는데, 이는 일반적으로 인용되는 종 구분 기준인 97%를 상회한다.[88] 그럼에도 불구하고, 이들은 의학적 이유(탄저병 등)로 인해 별도의 종으로 유지된다.[88]5. 3. ''Yersinia pestis'' (페스트균)
페스트균(Yersinia pestis)은 사실상 가성결핵균(Yersinia pseudotuberculosis)의 변종이지만, 병원성 섬을 가지고 있어 15,000년에서 20,000년 전에 급격히 다른 병리학적 증상(각각 흑사병과 결핵 유사 증상)을 나타내게 되었다.[89]6. 분석
세균은 초기에는 형태(비브리오, 간균, 구균 등), 내생포자 유무, 그람 염색, 호기성 조건 및 운동성을 기준으로 분류되었다.[74] 이러한 시스템은 대사 표현형 연구를 통해 변화했으며, 대사 특성이 사용되었다.[74] 최근, 분자 계통 발생학의 출현으로 여러 유전자가 종을 식별하는 데 사용되며, 가장 중요한 유전자는 16S rRNA 유전자이며, 그 다음으로 23S, ITS 영역, gyrB 등이 더 나은 해상도를 확인하는 데 사용된다.[74] 오늘날 분리된 균주를 종 또는 속에 매칭하는 가장 빠른 방법은 보편적인 프라이머로 16S 유전자를 증폭하고 1.4kb 증폭 산물을 시퀀싱하여 전문 웹 기반 식별 데이터베이스, 즉 리보솜 데이터베이스 프로젝트(Ribosomal Database Project)[http://rdp.cme.msu.edu/] , ARB SILVA(ARB SILVA)[http://www.arb-silva.de/documentation/background/]를 통해 수행된다.[74][75][76][77]
다음과 같은 여러 식별 방법이 존재한다:[106]
- '''표현형 분석'''
- * 지방산 분석
- * 성장 조건(한천 배지, [http://www.biolog.com/ Biolog multiwell plates])
- '''유전적 분석'''
- * DNA-DNA 혼성화
- * DNA 프로파일링
- * 시퀀스
- * GC 비율
- '''계통 발생학적 분석'''
- * 16S 기반 계통 발생
- * 다른 유전자를 기반으로 한 계통 발생
- * 다중 유전자 시퀀스 분석
- * 전체 게놈 시퀀스 기반 분석
6. 1. 표현형 분석
세균은 초기에는 형태(비브리오, 간균, 구균 등), 내생포자 유무, 그람 염색, 호기성 조건 및 운동성을 기준으로 분류되었다.[74] 이러한 시스템은 대사 표현형 연구를 통해 변화했으며, 대사 특성이 사용되었다.[74]표현형 분석 방법에는 지방산 분석 및 성장 조건(한천 배지, Biolog multiwell plates)을 이용하는 방법이 있다.[106]
6. 2. 유전적 분석
세균은 초기에는 형태, 내생포자 유무, 그람 염색, 호기성 조건, 운동성 등을 기준으로 분류되었다.[74] 이후 대사 표현형 연구를 통해 대사 특성을 이용한 분류가 이루어졌다.[74] 최근에는 분자 계통 발생학의 발달로 여러 유전자가 종 식별에 사용되고 있다. 가장 중요한 유전자는 16S rRNA 유전자이며, 23S, ITS 영역, gyrB 등도 해상도를 높이는 데 사용된다.[74]오늘날 분리된 균주를 종이나 속에 빠르게 매칭하는 방법은 보편적인 프라이머로 16S 유전자를 증폭하고, 1.4kb 증폭 산물을 시퀀싱하여 전문 웹 기반 식별 데이터베이스를 이용하는 것이다. 이러한 데이터베이스에는 리보솜 데이터베이스 프로젝트(Ribosomal Database Project)[http://rdp.cme.msu.edu/]와 ARB SILVA(ARB SILVA)[http://www.arb-silva.de/documentation/background/] 등이 있다.[74][75][76][77]
세균 식별에는 다음과 같은 여러 방법이 사용된다.[106]
- '''유전적 분석'''
- * DNA-DNA 혼성화
- * DNA 프로파일링
- * 시퀀스
- * GC 비율
7. 기타
참조
[1]
서적
Systemae Naturae, sive regna tria naturae, systematics proposita per classes, ordines, genera & species.
http://visualiseur.b[...]
[2]
논문
Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya
[3]
논문
Antony van Leeuwenhoek: tercentenary of his discovery of bacteria
[4]
논문
An abstract of a letter from Mr. Anthony Leevvenhoek at Delft, dated Sep. 17, 1683, Containing Some Microscopical Observations, about Animals in the Scurf of the Teeth, the Substance Call'd Worms in the Nose, the Cuticula Consisting of Scales
https://zenodo.org/r[...]
[5]
논문
Part of a Letter from Mr Antony van Leeuwenhoek, concerning the Worms in Sheeps Livers, Gnats, and Animalcula in the Excrements of Frogs
[6]
논문
Part of a Letter from Mr Antony van Leeuwenhoek, F. R. S. concerning Green Weeds Growing in Water, and Some Animalcula Found about Them
[7]
문서
The history of ''Bergey's Manual''
https://books.google[...]
Springer-Verlag, New York
[8]
문서
The genus Aquaspirillum
Springer-Verlag. New York.
[9]
웹사이트
Etymology of the word "bacteria"
http://www.etymonlin[...]
2006-11-23
[10]
논문
Bericht über die Verhandlungen der 33. Versammlung deutscher Naturforscher und Aerzte, gehalten in Bonn von 18 bis 24 September 1857
https://archive.org/[...]
1857
[11]
서적
Generelle Morphologie der Organismen
https://archive.org/[...]
Reimer
1867
[12]
문서
Osservazione microscopiche e deduzioni patologiche sul cholera asiatico
[13]
문서
"Dritter Beitrag zur Erkenntniss grosser Organisation in der Richtung des kleinsten Raumes"
[14]
문서
"Beiträge zur Kenntnis der Organization der Infusorien und ihrer geographischen Verbreitung besonders in Sibirien"
[15]
문서
Protomonas
[16]
논문
''Pansporella perplexa'': Réflexions sur la biologie et la phylogénie des protozoaires
1925
[17]
서적
Nomenclature and Classification in Bacteria in Relation to Plant Diseases
[18]
문서
Genera Schizomycetum
[19]
문서
"Untersuchungen uber Bakterien, II"
[20]
문서
"The main outlines of bacterial classification"
[21]
서적
Bacteria
Livingston
[22]
서적
System der Bakterien
Gustav Fischer
[23]
문서
"Die Hauptlinien des naturalischen Bakteriensystems nebst einer Ubersicht der Garungsphenomene"
[24]
서적
Bergey's Manual of Determinative Bacteriology
Williams & Wilkins Co.
[25]
논문
Studies in the Nomenclature and Classification of Bacteria: The Problem of Bacterial Nomenclature
https://www.ncbi.nlm[...]
1916
[26]
문서
Organismen in der Pockenlymphe
[27]
문서
"Untersuchungen ilber Bakterien"
[28]
문서
Schizomycetaceae
[29]
논문
Studies in the Nomenclature and Classification of the Bacteria: II. The Primary Subdivisions of the Schizomycetes
1917-03
[30]
논문
International Bacteriological Code of Nomenclature
1948-03
[31]
서적
Manuel de classification et détermination des bactéries anaérobies
Masson
[32]
서적
Bactéries in Précis de Botanique
Masson
[33]
논문
Proposals concerning the higher taxa of bacteria
[34]
문서
Bergey's manual of systematic bacteriology
Springer, New York
[35]
문서
The higher taxa, or, a place for everything...?
The Williams & Wilkins Co., Baltimore
[36]
논문
Toward automatic reconstruction of a highly resolved tree of life
[37]
논문
Are archaebacteria merely derived 'prokaryotes'?
[38]
논문
The universal ancestor
[39]
논문
The natural evolutionary relationships among prokaryotes
[40]
간행물
"Virus"
http://www.eoearth.o[...]
Encyclopedia of Earth
2010
[41]
논문
The origin and evolution of Archaea: a state of the art
[42]
논문
There must be a prokaryote somewhere: microbiology's search for itself
1994-03-01
[43]
논문
An ancient divergence among the bacteria
[44]
논문
Perceptual selectivity is task dependent: The pop-out effect poops out
[45]
논문
Valid publication of names of two domains and seven kingdoms of prokaryotes
2024-01-22
[46]
논문
Woese, Carl in the forefront of bacterial evolution revolution
1990-05-22
[47]
논문
Bacterial evolution
[48]
논문
How many species of prokaryotes are there?
[49]
논문
The neomuran origin of archaebacteria, the negibacterial root of the universal tree and bacterial megaclassification
[50]
논문
Rooting the tree of life by transition analyses
[51]
논문
Mapping the Tree of Life: Progress and Prospects
[52]
논문
Rooting the Ribosomal Tree of Life
[53]
논문
Rooting the Ribosomal Tree of Life
[54]
논문
Evidence for a New Root of the Tree of Life
[55]
논문
Evidence for a Gram-positive, Eubacterial Root of the Tree of Life
[56]
논문
The origin of a derived superkingdom: How a gram-positive bacterium crossed the desert to become an archaeon
[57]
논문
Protein phylogenies and signature sequences: A reappraisal of evolutionary relationships among archaebacteria, eubacteria, and eukaryotes
[58]
논문
Life's third domain (Archaea): An established fact or an endangered paradigm?
[59]
논문
The Natural Evolutionary Relationships among Prokaryotes
[60]
논문
Origin and evolution of organisms as deduced from 5S ribosomal RNA sequences
[61]
논문
An alternative to archaebacterial dogma
[62]
논문
Eocytes: A new ribosome structure indicates a kingdom with a close relationship to eukaryotes
[63]
논문
Mapping evolution with three dimensional ribosome structure
[64]
논문
Evidence for an early prokaryotic endosymbiosis
[65]
서적
International Code of Nomenclature of Bacteria: Bacteriological Code, 1990 Revision
ASM Press
[66]
서적
The Prokaryotes: A Handbook on the Biology of Bacteria: Symbiotic Associations, Biotechnology, Applied Microbiology
Springer
[67]
문서
Introductory
introduction.html
[68]
논문
Why and how to use the SeqCode
2024-03
[69]
논문
Robust demarcation of 17 distinct Bacillus species clades, proposed as novel Bacillaceae genera, by phylogenomics and comparative genomic analyses: description of Robertmurraya kyonggiensis sp. nov. and proposal for an emended genus Bacillus limiting it only to the members of the Subtilis and Cereus clades of species
2020-11-01
[70]
논문
Proposal of names for 329 higher rank taxa defined in the Genome Taxonomy Database under two prokaryotic codes.
2023-01-17
[71]
웹사이트
Frequently Asked Questions
https://www.ncbi.nlm[...]
National Center for Biotechnology Information (US)
2020-02-21
[72]
논문
SILVA, RDP, Greengenes, NCBI and OTT — how do these taxonomies compare?
2017-03
[73]
논문
Greengenes2 unifies microbial data in a single reference tree
2024-05
[74]
논문
The Pseudomonas Story
[75]
논문
The ribosomal RNA database project
[76]
논문
The Ribosomal Database Project: Improved alignments and new tools for rRNA analysis
[77]
논문
SILVA: A comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB
[78]
웹사이트
minimal standards
https://lpsn.dsmz.de[...]
[79]
웹사이트
Number of published names
http://www.bacterio.[...]
[80]
논문
The bacterial species dilemma and the genomic-phylogenetic species concept
[81]
논문
Opinion: Re-evaluating prokaryotic species
https://biblio.ugent[...]
[82]
논문
International Committee on Systematic Bacteriology: Announcement of the report of the ad hoc Committee on Reconciliation of Approaches to Bacterial Systematics
[83]
논문
Taxonomic note: a place for DNA–DNA reassociation and 16S rDNA sequence analysis in the present species definition in bacteriology
[84]
논문
Biodiversity: Are microbial species threatened?
[85]
논문
Exclusivity offers a sound yet practical species criterion for bacteria despite abundant gene flow
[86]
논문
Escherichia coli in disguise: molecular origins of Shigella
[87]
논문
Complete genome sequence of DSM 30083(T), the type strain (U5/41(T)) of Escherichia coli, and a proposal for delineating subspecies in microbial taxonomy
[88]
논문
Use of 16S rRNA, 23S rRNA, and gyrB Gene Sequence Analysis to Determine Phylogenetic Relationships of Bacillus cereus Group Microorganisms
[89]
논문
Yersinia pestis, the cause of plague, is a recently emerged clone of Yersinia pseudotuberculosis
[90]
논문
Probable synonymy of the nitrogen-fixing genus Azotobacter and the genus Pseudomonas
[91]
논문
Azotobacter vinelandii: a Pseudomonas in disguise?
[92]
논문
Phylogenetic relationships between Bacillus species and related genera inferred from comparison of 3' end 16S rDNA and 5' end 16S-23S ITS nucleotide sequences
[93]
논문
A revision of Rhizobium Frank 1889, with an emended description of the genus, and the inclusion of all species of Agrobacterium Conn 1942 and Allorhizobium undicola de Lajudie et al. 1998 as new combinations: Rhizobium radiobacter, R. Rhizogenes, R. Rubi, R. Undicola and R. Vitis
[94]
논문
Agrobacterium is a definable genus of the family Rhizobiaceae
[95]
논문
Classification and nomenclature of Agrobacterium and Rhizobium
[96]
논문
Phylogenomics and Comparative Genomic Studies Robustly Support Division of the Genus ''Mycobacterium'' into an Emended Genus ''Mycobacterium'' and Four Novel Genera
[97]
논문
Same meat, different gravy: ignore the new names of mycobacteria
2019-07
[98]
웹사이트
Genus: Mycobacteroides
https://lpsn.dsmz.de[...]
[99]
논문
Phylogenetic framework for the phylum Tenericutes based on genome sequence data: proposal for the creation of a new order Mycoplasmoidales ord. nov., containing two new families Mycoplasmoidaceae fam. nov. and Metamycoplasmataceae fam. nov. harbouring Eperythrozoon, Ureaplasma and five novel genera
[100]
웹사이트
Genus: Mycoplasmoides
https://lpsn.dsmz.de[...]
[101]
논문
Recommended rejection of the names Malacoplasma gen. nov., Mesomycoplasma gen. nov., Metamycoplasma gen. nov., Metamycoplasmataceae fam. nov., Mycoplasmoidaceae fam. nov., Mycoplasmoidales ord. nov., Mycoplasmoides gen. nov., Mycoplasmopsis gen. nov. [Gupta, Sawnani, Adeolu, Alnajar and Oren 2018] and all proposed species comb. nov. placed therein
2019
[102]
논문
Judicial Opinions 112–122
https://www.research[...]
2022-08-10
[103]
서적
Biological Nomenclature
Edward Arnold, London
[104]
웹사이트
app. 9 of BC
https://www.ncbi.nlm[...]
[105]
웹사이트
Classification of Phyla
https://lpsn.dsmz.de[...]
[106]
서적
Brock Biology of Microorganisms
Pearson/Benjamin Cummings
[107]
논문
Emendation of Principle 8, Rules 5b, 8, 15, 33a, and Appendix 7 of the International Code of Nomenclature of Prokaryotes to include the categories of kingdom and domain
2023-11-01
[108]
논문
Valid publication of names of two domains and seven kingdoms of prokaryotes
2024-01-22
[109]
논문
Proposal for a new hierarchic classification system, Actinobacteria classis nov
[110]
간행물
Classification
classification.html
[111]
논문
Valid publication of the names of forty-two phyla of prokaryotes
[112]
문서
SCHROETER (J.). In: F. COHN (ed.), Kryptogamenflora von Schlesien. Band 3, Heft 3, Pilze. J.U. Kern's Verlag, Breslau, 1885-1889, pp. 1-814.
[113]
간행물
Salmonella
s/salmonella.html
[114]
웹사이트
Help! Latin! How to avoid the most common mistakes while giving Latin names to newly discovered prokaryotes
http://www.bacterio.[...]
2011-04-14
[115]
문서
R. E. BUCHANAN, Taxonomy, Annu. Rev. Microbiol. 1955.9:1-20. http://www.annualreviews.org/doi/pdf/10.1146/annurev.mi.09.100155.000245
[116]
문서
ftp://ftp.ncbi.nih.g[...]
[117]
서적
Systemae Naturae, sive regna tria naturae, systematics proposita per classes, ordines, genera & species.
http://visualiseur.b[...]
[118]
저널
Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com