분자 진화
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
분자 진화는 20세기 초 비교 생화학에서 시작되어 DNA, RNA, 단백질 서열을 이용해 생물의 진화 역사를 연구하는 학문이다. 1970년대 핵산 서열 분석을 통해 분자 진화는 리보솜 RNA 서열까지 확장되었고, 칼 우스는 리보솜 RNA 염기서열을 기준으로 미생물을 분류하는 방법을 제안하여 분자진화학의 시작을 알렸다. 분자 계통 발생 분석은 DNA 시퀀싱 데이터를 사용하여 계통 발생 나무를 만들고, 유전자 중복, de novo 유전자 생성, 레트로트랜스포지션, 키메라 유전자 형성 등 다양한 메커니즘을 통해 새로운 유전자가 발생한다. 또한 유전자 집단 진화, 게놈 구조의 진화, 돌연변이, 선택, 유전적 부동, 유전자 전환 등의 요인들이 분자 진화에 영향을 미친다.
더 읽어볼만한 페이지
- 분자 진화 - 중립 진화 이론
중립 진화 이론은 자연 선택 외에 중립적인 돌연변이와 유전적 부동이 생물 진화의 주요 원동력이라고 주장하며, 유전자 다형성, 분자 시계 등을 설명하고 자연 선택과 병립 가능하다. - 분자 진화 - 유전자 중복
유전자 중복은 DNA 서열 일부가 한 개체 내에서 두 번 이상 나타나는 현상으로, 진화의 원동력으로서 새로운 유전적 기능 획득, 종 분화, 질병 발생에 영향을 미치며, 복제 오류 등 다양한 기제를 통해 발생하고 신기능 획득, 부분기능화, 유전자 소실로 이어질 수 있으며, 유전체 연구 및 유전 공학에서 활용된다. - 진화생물학 - 살아있는 화석
살아있는 화석은 화석 기록에서 오랜 기간 동안 형태적 변화가 거의 없이 나타나는 생물을 의미하며, 진화와 종분화를 이해하는 데 중요한 역할을 한다. - 진화생물학 - 적응
적응은 생물이 환경에 더 잘 생존하고 번식하도록 돕는 진화 과정으로, 형태, 행동, 생리적 특징 등 다양한 측면에서 유전적 변화, 공진화, 의태 등의 방식으로 나타난다. - 분자생물학 - 단백질
단백질은 아미노산 중합체로 생체 구조 유지와 기능에 필수적이며, 아미노산 서열에 따라 고유한 3차원 구조를 형성하여 효소, 구조, 수송, 저장, 수축, 방어, 조절 단백질 등 다양한 기능을 수행하고, 인체 내에서 건강 유지와 질병 예방에 중요한 역할을 하는 필수 영양소이다. - 분자생물학 - 의학
의학은 질병의 진단, 예후, 치료, 예방을 연구하는 과학 및 실천 분야이며, 고대부터 발전하여 현대에는 다양한 전문 분야로 세분화되고 첨단 기술 발전에 따라 혁신적인 변화를 겪고 있다.
| 분자 진화 | |
|---|---|
| 분자 진화 | |
| 학문 분야 | 진화생물학과 분자생물학의 교차점 |
| 연구 대상 | 시간이 지남에 따른 DNA, RNA, 단백질의 변화 과정 및 패턴 |
| 주요 목표 | 분자 시계를 사용하여 종의 분기 시점을 추정 유전자와 게놈의 진화적 관계를 밝히기 위해 계통수를 구축 자연 선택과 유전적 부동이 분자 수준에서 어떻게 작용하는지 이해 |
2. 역사
분자 진화의 역사는 20세기 초 비교 생화학과 1950년대 면역 분석법, 겔 전기영동, 그리고 종이 크로마토그래피와 같은 "지문 분석" 방법을 이용하여 상동 단백질을 탐구하면서 시작되었다.[1][2] 단백질 서열 분석의 등장으로 분자생물학자들은 서열 비교를 기반으로 계통 발생도를 만들고, 상동 서열 간의 차이를 분자시계로 사용하여 최근 공통 조상 이후의 시간을 추정할 수 있게 되었다.[3][1] 종 내 및 종 간의 분자적 차이가 놀라울 정도로 큰 것은 1960년대 후반 분자 진화의 중립 이론에 영감을 주었다.[4][5][6] 중립 이론은 분자시계에 대한 이론적 근거를 제공했지만, 시계의 타당성에는 필요하지 않다. 1970년대 이후 핵산 서열 분석을 통해 분자 진화는 단백질을 넘어서서 고도로 보존된 리보솜 RNA 서열에 이르게 되었고, 이는 초기 생명의 역사에 대한 개념 재정립의 기초가 되었다.[1]
분자 계통 발생은 DNA, RNA, 단백질 서열을 이용하여 생물 간의 진화적 관계를 연구하는 학문이다. 이 방법은 생물 종 간의 유연 관계를 파악하고, 이를 계통 발생 나무로 시각화한다.
1977년 미국 일리노이 대학교의 생물학 교수 칼 우스는 리보솜 RNA의 염기서열을 기준으로 한 미생물 분류 방법을 제안하였다. 그는 형태 등의 구분이 힘든 미생물의 경우 유전체의 차이를 기준으로 분류를 정립하는 것이 보다 과학적인 방법이 될 것이라 보았다. 우스의 제안은 생물학 학자 사이에 큰 반향을 일으켰고 분자생물학의 성과를 분류학에 도입하여 분자진화학이 시작되었다.
1977년의 논문에서 우스는 리보솜 RNA를 기준으로 원핵생물을 진정세균과 고균으로 구분하였다. 1990년 자신의 이론을 더욱 발전시킨 우스는 계보다 상위 단위인 도메인을 제안하여 생물의 최상위 분류로서 원핵생물, 원생생물, 식물, 동물, 균류의 5 계 구분 대신 진정세균, 고균, 진핵생물의 3 도메인 구분을 제안하였다.[41] 1982년에 분자생물학 및 진화 학회(Society for Molecular Biology and Evolution)가 설립되었다.
3. 분자 계통 발생
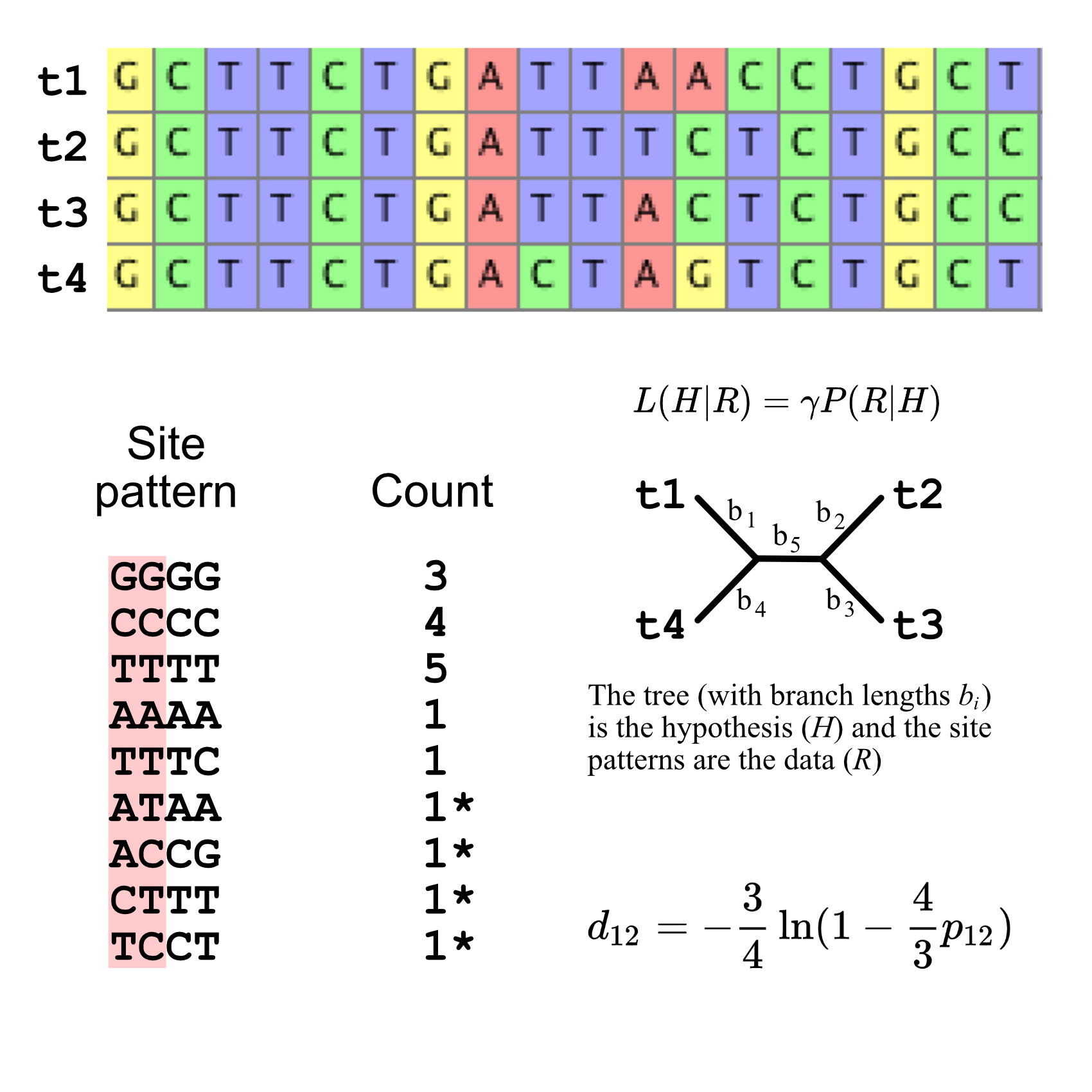
분자 계통 발생 분석은 DNA 시퀀싱 데이터를 활용한다. 다중 서열 정렬을 통해 상동성을 확인하고, 치환 모델을 적용하여 진화 과정에서 어떤 염기서열 패턴이 흔하게 나타나고 드물게 나타나는지 설명한다.
계통 발생 추론에는 여러 방법이 있으며, 일부는 유전자나 계통에 따라 진화 속도가 다를 수 있다는 이종속도(heterotachy)를 고려한다. 예를 들어, 헤모글로빈과 시토크롬 c는 서로 다른 속도로 진화한다.[7] 일반적으로 기능적으로 중요한 영역은 정제 선택의 영향으로 더 느리게 진화하며, 유사한 아미노산으로의 치환이 더 자주 일어난다.[7] 한 계통에서 빠른 진화는 방향 선택이 강하게 일어났음을 의미할 수 있다.[8]
3. 1. 분자 계통 발생 분석 단계
분자 계통 발생 분석은 DNA, RNA 또는 단백질 서열을 사용하여 생물 종 간의 진화적 관계를 파악하고, 이를 계통 발생 나무로 표현한다.
분자 계통 발생 추론은 DNA 시퀀싱 데이터를 기반으로 한다. 이 데이터는 정렬을 통해 어떤 위치(사이트)가 상동인지 확인된다. 치환 모델은 어떤 염기서열 패턴이 일반적이거나 드문지를 설명한다. 그 후, 정교한 계산 추론을 통해 하나 이상의 가능한 계통수를 생성한다.
일부 계통 발생 방법은 유전자나 단백질에 따라 진화 속도가 다를 수 있다는 점을 고려한다.[7] 예를 들어, 헤모글로빈과 시토크롬 c는 서로 다른 속도로 진화한다. 일반적으로 기능적으로 중요한 영역은 정제 선택으로 인해 더 느리게 진화하며, 유사한 아미노산을 포함하는 치환은 그렇지 않은 치환보다 더 자주 발생한다.[7]
4. 유전자 집단 진화
유전자 중복은 동일한 종 내에서 여러 상동성 단백질(파라로그)을 생성할 수 있다. 단백질의 계통 발생 분석은 단백질이 시간이 지남에 따라 어떻게 진화하고 구조와 기능이 변하는지 밝혀냈다.[9][10]
예를 들어, 리보뉴클레오티드 환원효소(RNR)는 다수의 구조적 및 기능적 변이체를 진화시켰다. '''I형''' RNR은 페리틴 서브유닛을 사용하며 보조인자로 사용하는 금속에 따라 다르다. '''II형''' RNR에서는 티일 라디칼이 아데노실코발라민 보조인자를 사용하여 생성되며 이 효소는 추가 서브유닛이 필요하지 않다(I형과는 대조적으로). '''III형''' RNR에서는 S-아데노실메티오닌이 4Fe-4S 클러스터에 결합하여 티일 라디칼이 생성된다. 즉, 단일 단백질 계열 내에서 수많은 구조적 및 기능적 메커니즘이 진화할 수 있다.[11]
개념 증명 연구에서 Bhattacharya와 동료들은 비효소적 산소 저장 단백질인 미오글로빈을 단 세 번의 돌연변이만으로 고효율 Kemp eliminase로 전환했다. 이는 단백질의 기능을 근본적으로 바꾸는 데 필요한 돌연변이는 소수에 불과함을 보여준다.[12] 유도 진화는 분자 진화에서 영감을 받은 방법을 사용하여 단백질을 설계하려는 시도이다.
5. 단일 유전자좌에서의 분자 진화
하나의 유전자좌에서의 변화는 새로운 돌연변이에서 시작되며, 자연선택, 유전적 부동, 유전자 전환의 조합에 의해 고정될 수 있다.[13]
5. 1. 돌연변이
돌연변이는 세포 또는 바이러스의 유전 물질(DNA 또는 RNA)에 발생하는 영구적이고 전달 가능한 변화이다. 돌연변이는 세포 분열 중 DNA 복제 과정의 오류나 방사선, 화학 물질, 기타 환경적 스트레스 요인, 바이러스, 또는 전위인자에 노출됨으로써 발생한다. DNA의 단 하나의 염기쌍에 대한 점 돌연변이가 단백질을 암호화하는 영역(Coding region)에 있을 경우, 이는 동의적 치환(아미노산 서열을 변경하지 않음)인지 또는 비동의적인지에 따라 특징지어진다. 다른 유형의 돌연변이는 DNA의 더 큰 부분을 수정하며 중복, 삽입, 결실, 역위 및 전좌를 일으킬 수 있다.[13]다양한 종류의 돌연변이의 비율 분포를 "돌연변이 스펙트럼"이라고 한다. 서로 다른 유형의 돌연변이는 매우 다양한 비율로 발생한다. 대부분의 유기체의 점 돌연변이율은 매우 낮으며, 세대당 부위당 대략 10−9에서 10−8 정도이다.[14] 하지만 일부 바이러스는 세대당 부위당 10−6 정도의 더 높은 돌연변이율을 갖는다.[15] 전이(A ↔ G 또는 C ↔ T)는 트랜스버전(퓨린(아데닌 또는 구아닌) ↔ 피리미딘(시토신 또는 티민, 또는 RNA에서는 우라실))보다 더 흔하다.[16] 아마도 인간에게 가장 흔한 돌연변이 유형은 짧은 반복 서열(예: 다양한 질병 관련 돌연변이의 기초가 되는 CAG 반복)의 길이 변화일 것이다. 이러한 STR 돌연변이는 세대당 10−3 정도의 비율로 발생할 수 있다.[17]
서로 다른 유형의 돌연변이의 서로 다른 빈도는 변이 도입의 편향(도착 편향)을 통해 진화에서 중요한 역할을 하고, 평행성, 경향 및 적응적 풍경의 탐색 가능성의 차이에 기여할 수 있다.[18][19] 돌연변이 편향은 평행 진화에 체계적이거나 예측 가능한 기여를 한다.[20] 1960년대 이후로 게놈 GC 함량은 돌연변이 경향을 반영하는 것으로 여겨져 왔다.[21][22] 돌연변이 편향은 또한 코돈 사용 편향에 기여한다.[23] 이러한 가설은 종종 중립성과 관련이 있지만, 최근의 이론적 및 실험적 결과는 돌연변이 경향이 변이 도입의 편향(도착 편향)을 통해 중립적 진화와 적응적 진화 모두에 영향을 미칠 수 있음을 입증했다.

5. 2. 선택
선택은 대립 유전자가 평균적으로 그것을 가진 개체에 더 큰 적응도, 즉 더 큰 생존이나 번식 능력을 부여할 때 발생할 수 있다. '''선택론적''' 접근 방식은 예를 들어 코돈 사용의 편향이 적어도 부분적으로는 심지어 약한 선택도 분자 진화를 형성할 수 있는 능력 때문이라는 점을 강조한다.[24]선택은 또한 유기체 적응도를 희생하여 유전자 수준에서 작용하여 유전체 내 갈등을 초래할 수 있다. 숙주 비용에도 불구하고 이기적 유전 요소에 대한 선택적 이점이 있을 수 있기 때문이다. 이러한 이기적 요소의 예로는 전위인자, 감수 분열 구동체 및 이기적 미토콘드리아가 있다.
선택은 검출될 수 있으며, Ka/Ks 비율, 맥도널드-크라이트먼 검정을 사용한다. 빠른 적응적 진화는 종종 유전체 내 갈등, 성적 대립 공진화, 그리고 면역계에 관여하는 유전자에서 발견된다.
5. 3. 유전적 부동
유전적 부동(Genetic drift)은 유한 집단에서 무작위 표본 추출의 확률적 효과로 인해 한 세대에서 다음 세대로 대립 유전자 빈도가 변하는 현상이다. 이러한 효과는 돌연변이가 고정될 때까지 축적될 수 있다. 중립 돌연변이의 경우, 세대당 고정률은 복제당 돌연변이율과 같다. 따라서 비교적 일정한 돌연변이율은 세대당 일정한 변화율을 생성한다(분자 시계).[25]선택 계수(selection coefficient)가 유효 집단 크기(effective population size)의 역수보다 작은 임계값 미만인 약간 해로운 돌연변이도 고정될 수 있다. 많은 게놈 특징은 작은 유효 집단 크기의 결과로 거의 중립적인 해로운 돌연변이의 축적으로 귀속되었다. 유효 집단 크기가 작을수록 선택의 비효율성으로 인해 더 다양한 돌연변이가 중립적인 것처럼 작동한다.
5. 4. 유전자 전환
유전자 전환은 재조합 과정에서 일어나는데, 이때 염기 손상이 상동적인 게놈 영역을 주형으로 사용하여 복구된다. 이는 편향된 과정일 수 있다. 즉, 하나의 대립 유전자가 유전자 전환 사건에서 다른 대립 유전자보다 주형이 될 확률이 더 높을 수 있다. 특히, GC 편향 유전자 전환은 게놈의 GC 함량을 증가시키는 경향이 있으며, 특히 재조합률이 높은 영역에서 그렇다.[26] 오류 수정 과정에서도 GC 편향에 대한 증거가 있다.[27] 이는 메틸시토신 탈아미노화의 높은 비율에 대한 적응일 수 있다고 생각되는데, 이는 C→T 전이로 이어질 수 있다.편향된 유전자 전환의 역학은 희귀할 때 선호되는 대립 유전자가 빈도가 지수적으로 증가하는 경향이 있다는 점에서 자연선택의 역학과 유사하다.
6. 게놈 구조 진화
게놈 구조는 반복 DNA의 양과 유기체 내 유전자 수에 영향을 받는다. 대부분의 박테리아, 초파리(*Drosophila*), 애기장대(*Arabidopsis*)와 같은 일부 유기체는 반복 서열이나 비암호화 DNA가 거의 없는, 특히 콤팩트한 게놈을 가지고 있다. 반면 포유류나 옥수수와 같은 다른 유기체는 많은 양의 반복 DNA, 긴 인트론, 그리고 유전자 사이의 상당한 간격을 가지고 있다. C 값 역설은 유기체의 '복잡성'과 게놈 크기 사이의 상관관계가 부족하다는 것을 의미한다.
개미 *Myrmecia pilosula*는 단 한 쌍의 염색체만 가지고 있는 반면,[29] 뱀혀고사리 Ophioglossum reticulatum는 최대 1,260개의 염색체를 가지고 있다.[30]
핵 게놈 외에도, 세포소기관은 자체 유전 물질을 포함하고 있다. 미토콘드리아와 엽록체 DNA는 분류군에 따라 다르지만, 특히 전자 전달계 구성 요소와 같은 막 단백질은 대부분 세포소기관에서 암호화된다.
6. 1. 게놈 크기
게놈 크기는 반복 DNA의 양과 유기체 내 유전자 수의 영향을 받는다. 대부분의 박테리아, 초파리(*Drosophila*), 애기장대(*Arabidopsis*)와 같은 일부 유기체는 반복 서열이나 비암호화 DNA가 거의 없는, 특히 콤팩트한 게놈을 가지고 있다. 포유류나 옥수수와 같은 다른 유기체는 많은 양의 반복 DNA, 긴 인트론, 그리고 유전자 사이의 상당한 간격을 가지고 있다. C 값 역설은 유기체의 '복잡성'과 게놈 크기 사이의 상관관계가 부족하다는 것을 의미한다. 이른바 역설에 대한 설명은 두 가지이다. 첫째, 반복적인 유전 요소는 많은 유기체의 게놈의 큰 부분을 차지하여 반수체 게놈의 DNA 함량을 증가시킨다. 반복적인 유전 요소는 종종 전이인자에서 유래한다.둘째, 유전자 수는 반드시 유기체의 발생 단계 또는 조직 유형의 수를 나타내는 것은 아니다. 발생 단계 또는 조직 유형이 적은 유기체는 발생 표현형에 영향을 미치는 많은 수의 유전자를 가질 수 있으며, 이는 발생 유전자 계열에 비해 유전자 함량을 증가시킨다.
게놈 크기에 대한 중립적인 설명은 집단 크기가 작을 때 많은 돌연변이가 거의 중립적으로 된다는 것을 시사한다. 따라서 작은 집단에서는 반복적인 내용과 기타 '정크' DNA가 유기체를 경쟁상 불리하게 만들지 않고 축적될 수 있다. 다세포 진핵생물에서 게놈 크기가 광범위하게 강한 선택을 받고 있다는 것을 시사하는 증거는 거의 없다. 유전자 함량과 무관하게 게놈 크기는 대부분의 생리적 특성과 상관관계가 낮으며, 포유류를 포함한 많은 진핵생물은 매우 많은 양의 반복 DNA를 가지고 있다.
그러나 조류는 비행에 대한 에너지 요구량 변화에 대한 반응으로 게놈 크기 감소에 대한 강한 선택을 경험했을 가능성이 높다. 사람과 달리 조류는 핵을 가진 적혈구를 생성하며, 더 큰 핵은 산소 운반 수준을 낮춘다. 조류의 신진대사는 주로 비행 때문에 포유류보다 훨씬 높으며 산소 요구량이 높다. 따라서 대부분의 조류는 반복 요소가 거의 없는 작고 콤팩트한 게놈을 가지고 있다. 간접적인 증거는 현대 조류의 비조류 수각류 공룡 조상[28]도 내온성 및 높은 에너지 요구량에 따라 게놈 크기가 감소했다는 것을 시사한다. 많은 박테리아도 복제 시간과 에너지 소비가 적합성과 매우 밀접하게 관련되어 있기 때문에 작은 게놈 크기에 대한 선택을 경험했다.
6. 2. 염색체 수와 구성
개미 *Myrmecia pilosula*는 단 한 쌍의 염색체만 가지고 있는 반면,[29] 뱀혀고사리 Ophioglossum reticulatum는 최대 1,260개의 염색체를 가지고 있다.[30] 생물체 게놈의 염색체 수는 반드시 게놈 내 DNA의 양과 상관관계가 있는 것은 아니다. 게놈 전체의 재조합 양은 종에 따라 염색체당 하나 또는 염색체 팔당 하나씩의 교차를 통해 염색체 수에 의해 직접 조절된다.[31]염색체 수의 변화는 다른 염색체 수가 잡종에서 생식 장벽 역할을 할 수 있기 때문에 종 분화에 중요한 역할을 할 수 있다. 인간의 2번 염색체는 두 개의 침팬지 염색체가 융합되어 만들어졌으며, 여전히 중앙 텔로미어와 퇴화된 두 번째 동원체를 포함하고 있다. 특히 식물에서 자주 발생하는 다배수체, 특히 이질다배수체는 모계 종과의 생식적 비호환성을 초래할 수도 있다. *Agrodiatus*속의 청색 나비는 n=10에서 n=134에 이르는 다양한 염색체 수를 가지고 있으며, 현재까지 확인된 종 분화율 중 하나의 가장 높은 비율을 가지고 있다.[32]
섬모충류 게놈은 각 유전자를 개별 염색체에 보관한다.
6. 3. 세포소기관
핵 게놈 외에도, 세포소기관은 자체 유전 물질을 포함하고 있다. 미토콘드리아와 엽록체 DNA는 분류군에 따라 다르지만, 특히 전자 전달계 구성 요소와 같은 막 단백질은 대부분 세포소기관에서 암호화된다. 엽록체와 미토콘드리아는 대부분의 종에서 모계 유전되며, 세포소기관이 난세포를 통해 전달되어야 하기 때문이다. 드물게, 어떤 종류의 담치는 아버지에게서 아들에게 미토콘드리아를 물려받는 것으로 알려져 있다.
7. 새로운 유전자 기원
새로운 유전자는 유전자 중복, *de novo* 유전자 생성, 레트로트랜스포지션, 키메라 유전자 형성, 비암호화 서열의 기존 유전자로의 통합, 유전자 절단 등 여러 가지 유전적 메커니즘을 통해 발생한다.[33]
유전자 중복은 초기에는 중복성을 초래하지만, 중복된 유전자 서열에 돌연변이가 일어나 새로운 기능이 생겨나거나, 원래 조상 유전자가 수행하던 기능의 일부만을 담당하도록 특수화될 수 있다. 레트로트랜스포지션은 mRNA를 DNA로 복사하여 게놈에 삽입하는 방식으로 유전자를 복제하며, 새로운 게놈 위치에 삽입되고 인트론이 없는 특징을 가지며 때로는 새로운 발현 패턴과 기능을 갖기도 한다. 키메라 유전자는 중복, 결실 또는 불완전한 레트로트랜스포지션으로 인해 두 개의 다른 암호화 서열 일부가 결합하여 새로운 유전자 서열을 생성할 때 형성된다. 이는 조절 변화를 일으키고 단백질 도메인을 재배열하여 새로운 적응 기능을 만들 수 있다.
- De novo* 유전자 생성은 이전에는 기능이 없던 DNA로부터 단백질 암호화 유전자와 비암호화 유전자를 생성하는 현상이다.[33] 예를 들어, Levine과 동료들은 ''초파리(D. melanogaster)'' 게놈에서 5개의 새로운 유전자 기원을 보고했다. 효모, 벼 그리고 인간과 같은 다른 생물에서도 유전자의 *de novo* 기원이 확인되었다. *De novo* 유전자는 이미 낮은 수준으로 발현되는 무작위 전사체로부터 진화할 수 있다.[34]
7. 1. 유전자 중복
유전자 중복은 동일한 종 내에서 여러 상동성 단백질(파라로그)을 생성할 수 있다. 단백질의 계통 발생 분석은 단백질이 시간이 지남에 따라 어떻게 진화하고 구조와 기능이 변하는지 밝혀냈다.[9][10]예를 들어, 리보뉴클레오티드 환원효소(RNR)는 다수의 구조적 및 기능적 변이체를 진화시켰다. '''I형''' RNR은 페리틴 서브유닛을 사용하며 보조인자로 사용하는 금속에 따라 다릅니다. '''II형''' RNR에서는 티일 라디칼이 아데노실코발라민 보조인자를 사용하여 생성되며 이 효소는 추가 서브유닛이 필요하지 않다(I형과는 대조적으로). '''III형''' RNR에서는 S-아데노실메티오닌이 4Fe-4S 클러스터에 결합하여 티일 라디칼이 생성된다. 즉, 단일 단백질 계열 내에서 수많은 구조적 및 기능적 메커니즘이 진화할 수 있다.[11]
개념 증명 연구에서 Bhattacharya와 동료들은 비효소적 산소 저장 단백질인 미오글로빈을 단 세 번의 돌연변이만으로 고효율 Kemp eliminase로 전환했다. 이는 단백질의 기능을 근본적으로 바꾸는 데 필요한 돌연변이는 소수에 불과함을 보여준다.[12]
유전자 중복은 처음에는 중복성을 초래한다. 그러나 중복된 유전자 서열은 돌연변이를 일으켜 새로운 기능을 개발하거나 특수화되어 새로운 유전자가 원래의 조상 기능의 하위 집합을 수행할 수 있다.
7. 2. *De novo* 유전자 생성
- De novo* 유전자 생성은 이전에는 기능이 없던 DNA로부터 단백질 암호화 유전자와 비암호화 유전자를 생성하는 현상이다.[33] 예를 들어, Levine과 동료들은 초파리(D. melanogaster) 게놈에서 5개의 새로운 유전자 기원을 보고했다. 효모, 벼, 인간과 같은 다른 생물에서도 유전자의 *de novo* 기원이 확인되었다. *De novo* 유전자는 이미 낮은 수준으로 발현되는 무작위 전사체로부터 진화할 수 있다.[34]
7. 3. 레트로트랜스포지션
레트로트랜스포지션은 mRNA를 DNA로 복사하여 게놈에 삽입하는 방식으로 유전자를 복제한다.[33] 레트로유전자는 대개 새로운 게놈 위치에 삽입되며, 인트론이 없고, 때로는 새로운 발현 패턴과 기능을 갖게 된다.7. 4. 키메라 유전자 형성
키메라 유전자는 유전자 중복, 결실 또는 불완전한 레트로트랜스포지션이 두 개의 다른 암호화 서열의 일부를 결합하여 새로운 유전자 서열을 생성할 때 형성된다.[33] 키메라는 종종 조절 변화를 일으키고 단백질 도메인을 재배열하여 새로운 적응 기능을 생성할 수 있다.8. 구성적 중립 진화
구성적 중립 진화(CNE)는 과잉 용량, 사전 억제 및 래칫팅의 원리를 통해 복잡한 시스템이 출현하여 개체군으로 확산될 수 있다고 설명하며, 스플라이소좀의 기원부터 미생물 군집의 복잡한 상호 의존성에 이르기까지 다양한 분야에 적용되어 왔다.[35][36][37]
9. 관련 학술지 및 학회
분자생물학 및 진화 학회(The Society for Molecular Biology and Evolution)는 학술지 "분자생물학 및 진화(Molecular Biology and Evolution)"와 "게놈 생물학 및 진화(Genome Biology and Evolution)"를 발간하며 매년 국제 학술대회를 개최한다. 분자 진화 관련 학술지로는 "분자 진화 저널(Journal of Molecular Evolution)"과 "분자 계통 발생 및 진화(Molecular Phylogenetics and Evolution)"가 있다. 분자 진화 연구는 유전학, 분자생물학, 유전체학, 계통분류학, 진화생물학 분야의 학술지에도 게재된다.
참조
[1]
논문
Paradox and persuasion: negotiating the place of molecular evolution within evolutionary biology
[2]
논문
Naturalists, molecular biologists, and the challenges of molecular evolution
[3]
논문
Molecules as documents of evolutionary history
1965-03-00
[4]
논문
Evolutionary rate at the molecular level
1968-02-00
[5]
논문
Non-Darwinian evolution
1969-05-00
[6]
서적
The Neutral Theory of Molecular Evolution
Cambridge University Press, Cambridge
[7]
논문
Sequence divergence, functional constraint, and selection in protein evolution
2003-00-00
[8]
논문
Beginner's Guide on the Use of PAML to Detect Positive Selection
2023-04-04
[9]
논문
ASIC and ENaC type sodium channels: conformational states and the structures of the ion selectivity filters
https://zenodo.org/r[...]
2017-02-00
[10]
논문
Epithelial sodium channel (ENaC) family: Phylogeny, structure-function, tissue distribution, and associated inherited diseases
2016-04-00
[11]
논문
Comprehensive phylogenetic analysis of the ribonucleotide reductase family reveals an ancestral clade
2022-09-00
[12]
논문
NMR-guided directed evolution
2022-10-00
[13]
웹사이트
What are Genetic Mutation?
https://www.singerin[...]
2016-03-23
[14]
논문
Experimental estimates of germline mutation rate in eukaryotes: a phylogenetic meta-analysis
2023-07-19
[15]
논문
Complexities of Viral Mutation Rates
2018-07-15
[16]
웹사이트
Transitions vs transversions
https://www.mun.ca/b[...]
[17]
논문
Mutation of human short tandem repeats
[18]
논문
Mutation bias interacts with composition bias to influence adaptive evolution
[19]
서적
Mutation-Driven Evolution
Oxford University Press
[20]
서적
Mutation, Randomness and Evolution
Oxford, Oxford
[21]
논문
On the Evolution of the Base Composition of DNA
[22]
논문
On the Genetic Basis of Variation and Heterogeneity of DNA Base Composition
[23]
논문
Climbing mount probable: mutation as a cause of nonrandomness in evolution
[24]
논문
Selection on codon bias
2008-12-00
[25]
서적
The Origins of Genome Architecture
Sinauer
[26]
논문
Biased gene conversion and the evolution of mammalian genomic landscapes
[27]
논문
GC-content evolution in mammalian genomes: the biased gene conversion hypothesis
2001-10-00
[28]
논문
Origin of avian genome size and structure in non-avian dinosaurs
2007-03-00
[29]
논문
Myrmecia pilosula, an Ant with Only One Pair of Chromosomes
1986-03-00
[30]
서적
Vegetables
https://archive.org/[...]
PROTA
2013-03-10
[31]
논문
Recombination is proportional to the number of chromosome arms in mammals
2001-04-00
[32]
논문
Karyotypic diversity and speciation in Agrodiaetus butterflies
2007-03-00
[33]
논문
New genes from non-coding sequence: the role of de novo protein-coding genes in eukaryotic evolutionary innovation
2015-09-00
[34]
논문
Putatively noncoding transcripts show extensive association with ribosomes
[35]
논문
On the possibility of constructive neutral evolution
1999-08-00
[36]
논문
Constructive neutral evolution: exploring evolutionary theory's curious disconnect
2012-10-01
[37]
논문
Constructive Neutral Evolution 20 Years Later
2021-04-01
[38]
논문
How a neutral evolutionary ratchet can build cellular complexity
2011-07-01
[39]
논문
Domestication of self-splicing introns during eukaryogenesis: the rise of the complex spliceosomal machinery
2017-12-01
[40]
논문
The generality of Constructive Neutral Evolution
https://www.reposito[...]
2018-03-19
[41]
간행물
1977년 칼 우스의 고균 제안
동아사이언스
2008-03-01
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com
