단백질 슈퍼패밀리
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
단백질 슈퍼패밀리는 진화적으로 관련된 단백질 그룹으로, 서열, 구조, 메커니즘의 유사성을 기반으로 식별된다. 서열 유사성은 상동성을 추론하는 데 가장 일반적으로 사용되지만, 구조적 유사성이나 메커니즘 유사성을 통해서도 관련성을 파악할 수 있다. 단백질 슈퍼패밀리는 공통 조상을 식별하는 현재 능력의 한계를 나타내며, 진화 연구에 중요한 자료가 된다. α/β 하이드롤라제 슈퍼패밀리, 알칼리성 인산분해효소 슈퍼패밀리, 글로빈 슈퍼패밀리, 면역글로불린 슈퍼패밀리 등 다양한 예시가 존재하며, Pfam, SCOP, CATH 등 여러 데이터베이스에서 단백질 슈퍼패밀리에 대한 정보를 제공한다.
더 읽어볼만한 페이지
- 단백질의 분류 - 단백질 패밀리
단백질 패밀리는 서열 유사성을 기준으로 단백질을 분류하는 그룹으로, 진화적 기원을 공유하며 서열, 기능, 구조의 유사성을 기준으로 다양한 범위로 분류되고, Pfam, PROSITE, SCOP, CATH 등의 데이터베이스에서 정보를 제공하며 생물학적 연구에 활용된다. - 단백질의 분류 - 수송체 분류 데이터베이스
수송체 분류 데이터베이스는 이온 통로, 포린, 기공 형성 독소 등 다양한 수송체들을 기능, 구조, 작용 기전 등에 따라 분류하는 데이터베이스이며, 이러한 수송체들은 생명체의 생존과 기능 유지에 필수적인 역할을 수행하여 질병 치료 및 예방 전략 개발에 기여한다. - 단백질 슈퍼패밀리 - 글로빈
글로빈은 글로빈 슈퍼패밀리에 속하는 단백질로, 8개의 α 나선으로 구성된 '글로빈 폴드'라는 3차원 구조를 가지며, 헤모글로빈, 미오글로빈 등 다양한 종류가 존재하고 산소 운반, 저장, 센서 기능 등을 수행하며, 공통 조상으로부터 진화하여 M/F, S, T 세 가지 주요 계열로 나뉜다. - 단백질 슈퍼패밀리 - 미오신
미오신은 액틴 필라멘트를 따라 움직이며 힘을 생성하는 단백질 분자로, 근육 수축, 세포 운동, 소포 수송 등 다양한 생체 내 기능에 관여하며, 근육형, 비근육형, 프로세시브형, 논프로세시브형으로 분류된다. - 분자 진화 - 중립 진화 이론
중립 진화 이론은 자연 선택 외에 중립적인 돌연변이와 유전적 부동이 생물 진화의 주요 원동력이라고 주장하며, 유전자 다형성, 분자 시계 등을 설명하고 자연 선택과 병립 가능하다. - 분자 진화 - 유전자 중복
유전자 중복은 DNA 서열 일부가 한 개체 내에서 두 번 이상 나타나는 현상으로, 진화의 원동력으로서 새로운 유전적 기능 획득, 종 분화, 질병 발생에 영향을 미치며, 복제 오류 등 다양한 기제를 통해 발생하고 신기능 획득, 부분기능화, 유전자 소실로 이어질 수 있으며, 유전체 연구 및 유전 공학에서 활용된다.
2. 식별
단백질 슈퍼패밀리는 여러 가지 방법을 사용하여 식별된다. 진화적으로 멀리 떨어진 구성원들을 묶는 방법과, 밀접하게 관련된 구성원들을 식별하는 방법은 다를 수 있다.
2. 1. 서열 유사성

역사적으로, 서로 다른 아미노산 서열의 유사성은 상동성을 추론하는 가장 일반적인 방법이었다.[5] 서열 유사성은 유사한 서열이 유전자 중복과 분기 진화의 결과일 가능성이 높고, 수렴 진화의 결과일 가능성은 낮기 때문에 관련성을 예측하는 좋은 지표로 간주된다. 아미노산 서열은 일반적으로 DNA 서열보다 더 잘 보존되므로(중복 유전 부호 때문) 더 민감한 검출 방법이다. 일부 아미노산은 유사한 특성(예: 전하, 소수성, 크기)을 가지므로, 이를 교환하는 보존적 돌연변이는 종종 기능에 중립적이다. 단백질의 가장 보존된 서열 영역은 일반적으로 촉매 부위 및 결합 부위와 같이 기능적으로 중요한 영역에 해당하는데, 이러한 영역은 서열 변화에 덜 관대하기 때문이다.
서열 유사성을 사용하여 상동성을 추론하는 데는 몇 가지 제한 사항이 있다. 동일한 구조를 생성하기 위해 보장되는 최소 서열 유사성 수준은 없다. 오랜 진화 기간 동안 관련 단백질은 서로 감지 가능한 서열 유사성을 나타내지 않을 수 있다. 많은 삽입 및 삭제가 있는 서열은 때때로 서열 정렬하기 어려울 수 있으며, 따라서 상동 서열 영역을 식별하기 어려울 수 있다. 예를 들어, 프로테아제의 PA 클랜에서는 촉매 삼중체의 잔기를 포함하여 슈퍼패밀리를 통해 단일 잔기도 보존되지 않는다. 반대로, 슈퍼패밀리를 구성하는 개별 패밀리는 서열 정렬을 기반으로 정의된다. 예를 들어, PA 클랜 내의 C04 프로테아제 패밀리가 있다.
그럼에도 불구하고, 알려진 서열의 수가 알려진 단백질 3차 구조의 수를 훨씬 능가하므로 서열 유사성은 관련성을 추론하는 데 가장 일반적으로 사용되는 증거 형태이다.[6] 구조적 정보가 없는 경우 서열 유사성은 어떤 단백질을 슈퍼패밀리에 할당할 수 있는지에 대한 한계를 제한한다.[6]
2. 2. 구조적 유사성
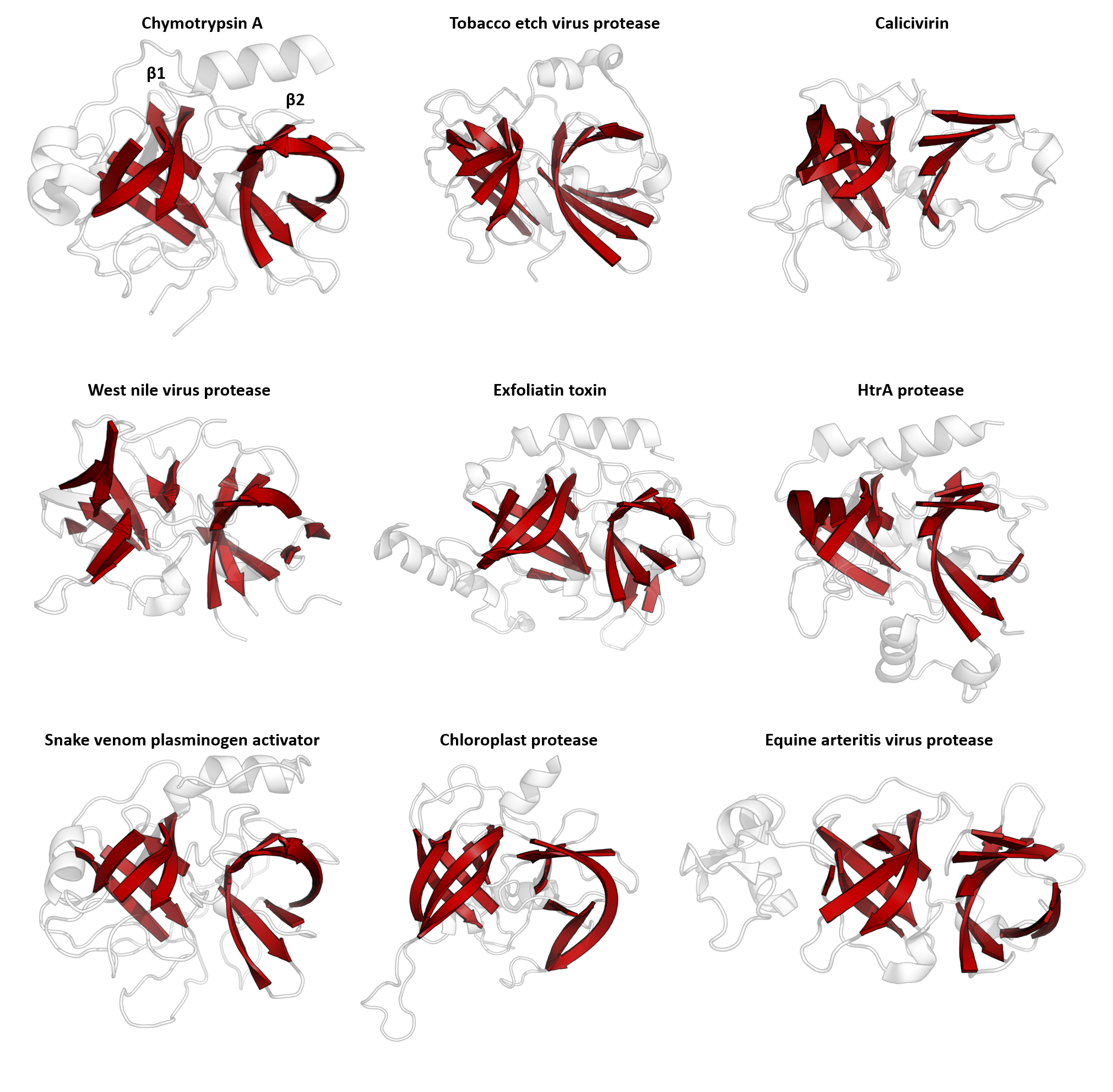
단백질의 구조는 서열보다 진화적으로 훨씬 더 보존되어, 매우 유사한 구조를 가진 단백질들이 완전히 다른 서열을 가질 수 있다.[7] 매우 긴 진화적 시간 척도에서, 극히 적은 잔기만이 감지 가능한 아미노산 서열 보존을 나타내지만, 이차 구조 요소와 삼차 구조 모티프는 매우 잘 보존된다. 일부 단백질 역학[8]과 단백질 구조의 형태 변화도 세르핀 슈퍼패밀리에서 관찰되는 것처럼 보존될 수 있다.[9] 결과적으로, 단백질의 삼차 구조는 서열에서 관련성에 대한 증거가 전혀 없는 경우에도 단백질 간의 상동성을 감지하는 데 사용될 수 있다. 구조적 정렬 프로그램 (예: DALI)은 관심 단백질의 3D 구조를 사용하여 유사한 폴드를 가진 단백질을 찾는다.[10] 그러나 드물게 관련 단백질이 구조적으로 달라지도록 진화할 수 있으며,[11] 이 경우 관련성은 다른 방법으로만 추론할 수 있다.[12][13][14]
2. 3. 메커니즘 유사성
슈퍼패밀리 내 효소의 촉매 메커니즘은 일반적으로 보존되지만, 기질 특이성은 상당히 다를 수 있다.[15] 촉매 잔기는 단백질 서열에서도 동일한 순서로 나타나는 경향이 있다.[16] 프로테아제 PA 클랜 내의 패밀리들은 촉매 삼합체 잔기가 촉매 작용을 수행하기 위해 발산 진화했지만, 모든 구성원들은 단백질, 펩타이드 또는 아미노산에 대해 유사한 메커니즘을 사용하여 공유 결합, 친핵성 촉매를 수행한다.[17] 그러나 메커니즘만으로는 관련성을 추론하기에 충분하지 않다. 일부 촉매 메커니즘은 여러 번 독립적으로 수렴 진화되었으므로, 별도의 슈퍼패밀리를 형성하며,[18][19][20] 일부 슈퍼패밀리에서는 다양하고 다른 (하지만 종종 화학적으로 유사한) 메커니즘을 나타낸다.[15][21]3. 진화적 의의
단백질 슈퍼패밀리는 공통 조상을 식별하는 현재 능력의 한계를 나타낸다.[22] 이들은 현재 가능한 직접적인 증거에 기반한 가장 큰 진화 집단이며, 현재 연구되고 있는 가장 오래된 진화 사건 중 하나이다. 일부 슈퍼패밀리는 모든 계에 구성원을 가지고 있는데, 이는 해당 슈퍼패밀리의 마지막 공통 조상이 모든 생명의 마지막 보편 공통 조상(LUCA)에 있었다는 것을 나타낸다.[23]
슈퍼패밀리 구성원은 조상 단백질이 조상 종에 존재했던 단백질 형태(orthology)인 서로 다른 종에 존재할 수 있다. 반대로, 단백질은 동일한 종에 존재할 수 있지만, 유전자 중복을 통해 유전자가 게놈에서 유전자 중복된 단일 단백질(paralogy)에서 진화했다.
3. 1. 다양화
대부분의 단백질은 여러 개의 도메인을 포함하고 있다. 진핵생물 단백질의 66~80%는 여러 개의 도메인을 가지고 있는 반면, 원핵생물 단백질의 약 40~60%는 여러 개의 도메인을 가지고 있다.[5] 시간이 지남에 따라, 많은 도메인 슈퍼패밀리들이 서로 혼합되었다. 실제로 "일관되게 고립된 슈퍼패밀리"를 발견하는 것은 매우 드물다.[5][1] 도메인이 결합할 때, N-말단에서 C-말단 도메인 순서("도메인 아키텍처")는 일반적으로 잘 보존된다. 또한, 자연계에서 관찰되는 도메인 조합의 수는 가능성의 수에 비해 적으며, 이는 모든 조합에 선택이 작용함을 시사한다.[5]4. 예시
- α/β 하이드롤라제 슈퍼패밀리: 구성원들은 8개의 가닥으로 연결된 α/β 시트를 공유하며, 나선이 연결되어 있고, 동일한 순서의 촉매 삼합체 잔기를 포함한다.[24] 프로테아제, 리파제, 과산화효소, 에스테라제, 에폭시드 가수분해효소, 탈할로젠화효소 등이 여기에 포함된다.[25]
- 알칼리성 인산분해효소 슈퍼패밀리: 구성원들은 αβα 샌드위치 구조[26]를 공유하며, 공통적인 메커니즘을 통해 일반적인 다중 반응을 수행한다.[27]
- 글로빈 슈퍼패밀리: 구성원들은 8개의 알파 나선 구형 글로빈 폴드를 공유한다.[28][29]
- 면역글로불린 슈퍼패밀리: 구성원들은 두 개의 역평행 시트 (Ig-폴드)의 샌드위치와 같은 구조를 공유하며, 인식, 결합 및 부착에 관여한다.[30][31]
- PA 클랜: 구성원들은 키모트립신과 같은 이중 β-배럴 폴드와 유사한 단백질 분해 메커니즘을 공유하지만, 서열 동일성은 10% 미만이다. 이 클랜에는 시스테인 프로테아제와 세린 프로테아제 (다른 친핵체)가 모두 포함된다.[2][32]
- Ras 슈퍼패밀리: 구성원들은 5개의 α-나선으로 둘러싸인 6가닥 β 시트의 공통적인 촉매 G 도메인을 공유한다.[33]
- RSH 슈퍼패밀리: 구성원들은 스트린젠트 반응에서 ppGpp 알람온을 가수분해 및/또는 합성하는 능력을 공유한다.[34]
- Serpin 슈퍼패밀리: 구성원들은 큰 입체 구조 변화를 겪을 수 있는 고에너지, 스트레스된 폴드를 공유하며, 이는 일반적으로 구조를 파괴하여 세린 프로테아제 및 시스테인 프로테아제를 억제하는 데 사용된다.[9]
- TIM 배럴 슈퍼패밀리: 구성원들은 큰 α8β8 배럴 구조를 공유한다. 이는 가장 일반적인 단백질 폴드 중 하나이며, 이 슈퍼패밀리의 단일 계통성은 여전히 논쟁의 대상이다.[35][36]
5. 단백질 슈퍼패밀리 관련 자료
다음은 단백질 슈퍼패밀리와 단백질 폴드를 문서화하는 여러 생물학 데이터베이스들이다.
마찬가지로, 표적 구조와 구조적 상동성을 가진 단백질을 PDB에서 검색하는 알고리즘도 있다.
- DALI - 거리 정렬 매트릭스 방법을 기반으로 한 구조 정렬
참조
[1]
논문
Dali server: conservation mapping in 3D
2010-07
[2]
논문
MEROPS: the database of proteolytic enzymes, their substrates and inhibitors
2012-01
[3]
논문
Updating the sequence-based classification of glycosyl hydrolases
1996-06
[4]
웹사이트
Clustal FAQ #Symbols
http://www.ebi.ac.uk[...]
2014-12-08
[5]
논문
The folding and evolution of multidomain proteins
2007-04
[6]
논문
SUPFAM--a database of potential protein superfamily relationships derived by comparing sequence-based and structure-based families: implications for structural genomics and function annotation in genomes
2002-01
[7]
논문
Protein families and their evolution-a structural perspective
2005
[8]
논문
Sequence evolution correlates with structural dynamics
2012-09
[9]
논문
The serpins are an expanding superfamily of structurally similar but functionally diverse proteins. Evolution, mechanism of inhibition, novel functions, and a revised nomenclature
2001-09
[10]
논문
Dali server update
2016-07
[11]
논문
Cross-Over between Discrete and Continuous Protein Structure Space: Insights into Automatic Classification and Networks of Protein Structures
2009
[12]
논문
Evolution of primate α and θ defensins revealed by analysis of genomes
2014-06
[13]
논문
Structural drift: a possible path to protein fold change
2005-04
[14]
논문
Proteins that switch folds
2010-08
[15]
서적
Function Diversity within Folds and Superfamilies
Springer Netherlands
2017
[16]
논문
Causes of evolutionary rate variation among protein sites
2016-02
[17]
논문
Handicap-Recover Evolution Leads to a Chemically Versatile, Nucleophile-Permissive Protease
2015-09
[18]
논문
Intrinsic evolutionary constraints on protease structure, enzyme acylation, and the identity of the catalytic triad
2013-02
[19]
논문
An evolving hierarchical family classification for glycosyltransferases
2003-04
[20]
논문
Independent evolution of four heme peroxidase superfamilies
2015-05
[21]
논문
The Structure–Function Linkage Database
2013-11-23
[22]
논문
Protein structure and evolutionary history determine sequence space topology
2005-03
[23]
논문
Protein superfamily evolution and the last universal common ancestor (LUCA)
2006-10
[24]
논문
Alpha/beta hydrolase fold: an update
[25]
논문
Alpha/beta hydrolase fold enzymes: the family keeps growing
1999-12
[26]
웹사이트
SCOP
http://scop.mrc-lmb.[...]
2014-05-28
[27]
논문
Efficient, crosswise catalytic promiscuity among enzymes that catalyze phosphoryl transfer
2013-01
[28]
서적
Introduction to protein structure
Garland Pub.
1999
[29]
논문
Aplysia limacina myoglobin. Crystallographic analysis at 1.6 A resolution
1989-02
[30]
논문
The immunoglobulin fold. Structural classification, sequence patterns and common core
1994-09
[31]
논문
Cell adhesion molecules 1: immunoglobulin superfamily
[32]
논문
Viral cysteine proteases are homologous to the trypsin-like family of serine proteases: structural and functional implications
1988-11
[33]
논문
The guanine nucleotide-binding switch in three dimensions
2001-11
[34]
논문
The RelA/SpoT Homolog (RSH) Superfamily: Distribution and Functional Evolution of ppGpp Synthetases and Hydrolases across the Tree of Life
2011-08-09
[35]
논문
One fold with many functions: the evolutionary relationships between TIM barrel families based on their sequences, structures and functions
2002-08
[36]
논문
An α/β-barrel full of evolutionary trouble
[37]
논문
Dali server: conservation mapping in 3D
2010-07
[38]
논문
MEROPS: the database of proteolytic enzymes, their substrates and inhibitors
2012-01
[39]
논문
Updating the sequence-based classification of glycosyl hydrolases
1996-06
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com

