유전체학
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
유전체학은 생물의 유전체를 연구하는 학문으로, DNA 염기 서열 분석 기술의 발전과 함께 비약적으로 발전했다. 1950년대 DNA 구조 규명 이후, 1970년대 DNA 염기 서열 분석 기술이 개발되었고, 2000년대 들어 차세대 염기서열 분석 기술(NGS)의 등장으로 유전체 해독 비용과 시간이 크게 줄어들었다. 유전체학은 유전체의 시퀀싱, 어셈블리, 주석 과정을 통해 유전자 기능과 상호작용을 연구하며, 기능 유전체학, 구조 유전체학, 후성유전체학, 메타게노믹스 등의 세부 분야로 나뉜다. 의학, 생명공학, 농업, 법의학 등 다양한 분야에 응용되며, 질병 연구, 맞춤 의학, 유전자 조작, 품종 개량 등에 기여한다.
더 읽어볼만한 페이지
- 유전체학 - 발현체학
- 유전체학 - 유전형 분석
유전자형 분석은 DNA 염기 서열 분석, RFLP, PCR, DNA 마이크로어레이 등 다양한 기술을 통해 유전형을 파악하는 방법으로 질병 진단, 품종 개량 등 여러 분야에 활용되며 윤리적 문제와 사회적 영향을 동반하지만, 혁신적인 변화를 가져올 것으로 예상되는 기술이다. - 생물학에 관한 - 해부학
해부학은 생물체의 구조와 구성 요소를 연구하는 학문으로, 육안 해부학과 현미경 해부학으로 나뉘며, 인체 해부학 외에도 동물, 식물, 미술 해부학 등 다양한 분야가 존재한다. - 생물학에 관한 - 죽음
죽음은 생명 활동의 영구적 종식으로 의학, 법, 사회, 종교, 심리, 생물학 등 다양한 관점에서 해석되며, 전통적인 심폐사 외에 뇌사도 죽음으로 인정되고, 개발도상국은 전염병, 선진국은 노화 관련 질병이 주요 사망 원인이며, 문화와 종교에 따라 다양한 이해와 관습이 존재하고, 수명 연장, 냉동 보존술, 존엄사, 안락사 등에 대한 논의가 이루어지고 있다.
| 유전체학 | |
|---|---|
| 개요 | |
| 학문 분야 | 유전학 |
| 정의 | 생물의 유전체 전체를 연구하는 학문 분야 |
| 상세 정보 | |
| 연구 대상 | 유전체의 구조 기능 진화 지도 제작 편집 |
| 연구 방법 | 유전자 지도의 작성 유전자 서열 결정 생물 정보학 통계학 분석 |
| 응용 분야 | 의학 생명공학 농업 범죄 수사 |
| 관련 기술 | 유전자 편집 기술 (예: CRISPR 기술) DNA 염기서열 분석 기술 |
| 역사 | |
| 발전 배경 | 분자생물학의 발전과 유전체 분석 기술의 발전 |
| 주요 연구 | 인간 게놈 프로젝트 다양한 생물 종의 유전체 해독 |
| 미래 전망 | 개인 맞춤 의료 질병 예측 및 예방 새로운 품종 개발 |
| 관련 학문 분야 | |
| 관련 학문 | 생물정보학 유전체 편집 분자생물학 단백질체학 대사체학 전사체학 후성유전체학 |
| 기타 | |
| 관련 개념 | 유전체 유전자 DNA RNA 염색체 |
| 참고 문헌 | |
| 관련 저널 | Genomics (학술지) Genome Research Nature Genetics |
2. 역사
로잘린드 프랭클린이 DNA의 나선형 구조를 확인하고, 제임스 왓슨과 프랜시스 크릭이 1953년에 DNA 구조를 발표했으며, 프레드 생어가 1955년에 아미노산 서열을 발표한 이후, 핵산 염기 서열 분석은 초기 분자 생물학자들의 주요 목표가 되었다.[1] 1964년, 로버트 W. 홀리와 동료들은 최초로 결정된 핵산 염기 서열인 알라닌 전이 RNA의 리보뉴클레오타이드 서열을 발표했다.[1][1] 마셜 니렌버그와 필립 레더는 유전 암호의 삼중항 특성을 밝혀냈고, 실험에서 64개의 코돈 중 54개의 서열을 결정할 수 있었다.[1] 1972년, 발터 피어스와 겐트 대학교 분자 생물학 연구소의 연구진은 최초로 유전자 서열, 즉 박테리오파지 MS2 외피 단백질 유전자를 결정했다.[1] 피어스 그룹은 MS2 외피 단백질 연구를 확장하여, 박테리오파지 MS2-RNA 및 원숭이 바이러스 40의 전체 뉴클레오타이드 서열을 각각 1976년과 1978년에 결정했다.[1][1]
프레더릭 생어는 DNA 염기서열 분석 기술 개발에 핵심적인 역할을 했다.[1] 1975년, 그는 앨런 컬슨과 함께 방사성 동위원소로 표지된 뉴클레오티드와 DNA 중합효소를 사용하여 "플러스 앤 마이너스 기술"이라고 부르는 염기서열 분석 절차를 발표했다.[1][1] 1977년에 그의 연구팀은 φX174의 5,386개의 뉴클레오티드 대부분을 염기서열 분석하여, 최초로 완전하게 염기서열이 분석된 DNA 기반 게놈을 완성했다.[1] "플러스 앤 마이너스" 방법의 개선으로 생어 시퀀싱 방법이 개발되었다.[1][1] 같은 해 하버드 대학교의 월터 길버트와 앨런 맥섬은 맥섬-길버트 시퀀싱을 독자적으로 개발했다.[1][1] 핵산 염기서열 분석에 대한 획기적인 업적으로, 길버트와 생어는 1980년 노벨상 화학 부문을 재조합 DNA 연구의 폴 버그와 함께 공동 수상했다.
최초의 유전체 연구는 영국 케임브리지의 MRC센터의 프레드 생어에 의해서였다. 생어는 파이 x174라는 바이러스의 유전체 서열을 해석했고, 이 연구결과는 Journal of Molecular Biology에 실렸다. 곧이어 그는 미토콘드리아 유전체서열을 생산했다. 이러한 결과에 자극을 받은 미국연구자와 정치가들은 인간 유전체 프로젝트를 하게 되었고, 2003년에 인간 유전체 서열이 영국과 미국에 의해 공동 작업으로 발표되었다.
1981년 인간 미토콘드리아 유전체(16,568 bp) 해독을 시작으로,[1] 다양한 생물종의 유전체 해독이 이루어졌다. 최초의 유전체 연구는 영국 케임브리지의 MRC센터의 프레드 생어에 의해서였으며, 그는 DNA 염기서열해석법으로 노벨 화학상을 받았다. 최초의 완전한 진핵생물 세포소기관 유전체인 인간 미토콘드리아 (16,568 bp, 약 16.6 kb [킬로베이스])는 1981년에 보고되었으며,[1] 1986년에는 최초의 엽록체 유전체가 보고되었다.[1] 이후, 효모, 인플루엔자균, 예쁜꼬마선충, 초파리, 애기장대 등 다양한 생물종의 유전체 해독이 이어졌다.[1]
1990년, 인간 게놈 프로젝트가 시작되었다. 이는 인간 유전체 전체 염기서열을 해독하는 것을 목표로 하는 국제적인 협력 프로젝트였다. 2003년에 인간 유전체 서열이 영국과 미국에 의해 공동 작업으로 발표되었다.[1] 2001년 초에 인간 게놈의 대략적인 초안이 완성되었고,[1] 2003년에 완료된 이 프로젝트는 특정 개인의 전체 유전체를 해독했으며, 2007년까지 이 서열은 "완료"로 선언되었다.[1] 이는 생명과학 역사상 기념비적인 사건으로 기록되었으며, 유전체학 연구의 폭발적인 성장을 이끌었다. 그 이후 수년 동안, 많은 다른 개인의 유전체가 해독되었으며, 이는 부분적으로 2012년 10월에 1,092개의 유전체 해독을 발표한 1000 게놈 프로젝트의 지원을 받았다.[1]
2000년대 중반, 차세대 염기서열 분석(Next-Generation Sequencing, NGS) 기술이 등장하면서 유전체 해독 비용과 시간이 획기적으로 단축되었다. NGS 기술은 한 번에 수많은 DNA 조각을 병렬로 분석하여 대량의 염기서열 데이터를 빠르게 생산할 수 있다. NGS 기술의 발전은 다양한 생물종의 유전체 해독을 가속화하고, 개인 유전체 분석, 질병 진단, 맞춤 의학 등 다양한 분야에서 유전체 정보를 활용하는 것을 가능하게 했다. 역사적으로 염기 서열 분석은 공동 게놈 연구소와 같은 중앙 집중형 시설에서 이루어졌으나, 염기 서열 결정 기술이 발전하고 벤치탑형 시퀀서가 개발되면서 평균적인 학술 연구실에서도 시퀀서를 사용할 수 있게 되었다. 현재는 3세대 염기서열 분석 기술(긴 서열 해독 기술)도 개발되어 유전체 연구의 새로운 지평을 열고 있다.
2. 1. 초기 유전체 연구
로잘린드 프랭클린이 DNA의 나선형 구조를 확인하고, 제임스 왓슨과 프랜시스 크릭이 1953년에 DNA 구조를 발표했으며, 프레드 생어가 1955년에 아미노산 서열을 발표한 이후, 핵산 염기 서열 분석은 초기 분자 생물학자들의 주요 목표가 되었다.[1] 1964년, 로버트 W. 홀리와 동료들은 최초로 결정된 핵산 염기 서열인 알라닌 전이 RNA의 리보뉴클레오타이드 서열을 발표했다.[1][1] 마셜 니렌버그와 필립 레더는 유전 암호의 삼중항 특성을 밝혀냈고, 실험에서 64개의 코돈 중 54개의 서열을 결정할 수 있었다.[1] 1972년, 발터 피어스와 겐트 대학교 분자 생물학 연구소의 연구진은 최초로 유전자 서열, 즉 박테리오파지 MS2 외피 단백질 유전자를 결정했다.[1] 피어스 그룹은 MS2 외피 단백질 연구를 확장하여, 박테리오파지 MS2-RNA 및 원숭이 바이러스 40의 전체 뉴클레오타이드 서열을 각각 1976년과 1978년에 결정했다.[1][1]프레더릭 생어는 DNA 염기서열 분석 기술 개발에 핵심적인 역할을 했다.[1] 1975년, 그는 앨런 컬슨과 함께 방사성 동위원소로 표지된 뉴클레오티드와 DNA 중합효소를 사용하여 "플러스 앤 마이너스 기술"이라고 부르는 염기서열 분석 절차를 발표했다.[1][1] 1977년에 그의 연구팀은 φX174의 5,386개의 뉴클레오티드 대부분을 염기서열 분석하여, 최초로 완전하게 염기서열이 분석된 DNA 기반 게놈을 완성했다.[1] "플러스 앤 마이너스" 방법의 개선으로 생어 시퀀싱 방법이 개발되었다.[1][1] 같은 해 하버드 대학교의 월터 길버트와 앨런 맥섬은 맥섬-길버트 시퀀싱을 독자적으로 개발했다.[1][1] 핵산 염기서열 분석에 대한 획기적인 업적으로, 길버트와 생어는 1980년 노벨상 화학 부문을 재조합 DNA 연구의 폴 버그와 함께 공동 수상했다.
최초의 유전체 연구는 영국 케임브리지의 MRC센터의 프레드 생어에 의해서였다. 생어는 파이 x174라는 바이러스의 유전체 서열을 해석했고, 이 연구결과는 Journal of Molecular Biology에 실렸다. 곧이어 그는 미토콘드리아 유전체서열을 생산했다. 이러한 결과에 자극을 받은 미국연구자와 정치가들은 인간 유전체 프로젝트를 하게 되었고, 2003년에 인간 유전체 서열이 영국과 미국에 의해 공동 작업으로 발표되었다.
2. 2. 인간 유전체 프로젝트와 유전체학의 발전
1981년 인간 미토콘드리아 유전체(16,568 bp) 해독을 시작으로,[1] 다양한 생물종의 유전체 해독이 이루어졌다. 최초의 유전체 연구는 영국 케임브리지의 MRC센터의 프레드 생어에 의해서였으며, 그는 DNA 염기서열해석법으로 노벨 화학상을 받았다. 최초의 완전한 진핵생물 세포소기관 유전체인 인간 미토콘드리아 (16,568 bp, 약 16.6 kb [킬로베이스])는 1981년에 보고되었으며,[1] 1986년에는 최초의 엽록체 유전체가 보고되었다.[1] 이후, 효모, 인플루엔자균, 예쁜꼬마선충, 초파리, 애기장대 등 다양한 생물종의 유전체 해독이 이어졌다.[1]1990년, 인간 게놈 프로젝트가 시작되었다. 이는 인간 유전체 전체 염기서열을 해독하는 것을 목표로 하는 국제적인 협력 프로젝트였다. 2003년에 인간 유전체 서열이 영국과 미국에 의해 공동 작업으로 발표되었다.[1] 2001년 초에 인간 게놈의 대략적인 초안이 완성되었고,[1] 2003년에 완료된 이 프로젝트는 특정 개인의 전체 유전체를 해독했으며, 2007년까지 이 서열은 "완료"로 선언되었다.[1] 이는 생명과학 역사상 기념비적인 사건으로 기록되었으며, 유전체학 연구의 폭발적인 성장을 이끌었다. 그 이후 수년 동안, 많은 다른 개인의 유전체가 해독되었으며, 이는 부분적으로 2012년 10월에 1,092개의 유전체 해독을 발표한 1000 게놈 프로젝트의 지원을 받았다.[1]
2. 3. 차세대 염기서열 분석 기술 (NGS)의 등장과 발전
2000년대 중반, 차세대 염기서열 분석(Next-Generation Sequencing, NGS) 기술이 등장하면서 유전체 해독 비용과 시간이 획기적으로 단축되었다. NGS 기술은 한 번에 수많은 DNA 조각을 병렬로 분석하여 대량의 염기서열 데이터를 빠르게 생산할 수 있다. NGS 기술의 발전은 다양한 생물종의 유전체 해독을 가속화하고, 개인 유전체 분석, 질병 진단, 맞춤 의학 등 다양한 분야에서 유전체 정보를 활용하는 것을 가능하게 했다. 역사적으로 염기 서열 분석은 공동 게놈 연구소와 같은 중앙 집중형 시설에서 이루어졌으나, 염기 서열 결정 기술이 발전하고 벤치탑형 시퀀서가 개발되면서 평균적인 학술 연구실에서도 시퀀서를 사용할 수 있게 되었다. 현재는 3세대 염기서열 분석 기술(긴 서열 해독 기술)도 개발되어 유전체 연구의 새로운 지평을 열고 있다.3. 유전체 해독
유전체 해독은 크게 샷건 시퀀싱과 고처리량 시퀀싱(차세대 염기서열 분석)으로 나뉜다.[1]
역사적으로 시퀀싱은 비용이 많이 드는 장비와 필요한 기술 지원을 갖춘 연구 실험실을 보유한 중앙 집중식 시설(공동 게놈 연구소와 같은 대규모 독립 기관에서 지역 분자 생물학 핵심 시설에 이르기까지)인 "시퀀싱 센터"에서 수행되었다. 그러나 시퀀싱 기술이 계속 발전함에 따라, 효과적인 턴어라운드 벤치톱 시퀀서의 새로운 세대가 일반적인 학술 실험실의 손에 들어왔다.[1]
'''샷건 시퀀싱 (Shotgun Sequencing)'''
샷건 시퀀싱은 DNA를 무작위로 작은 조각으로 잘라낸 후, 각 조각의 염기서열을 분석하고, 중첩되는 부분을 찾아 이어 붙여 전체 염기서열을 완성하는 방법이다.[1] 1000 염기쌍보다 긴 DNA 서열 분석을 위해 설계된 이 방법은, 빠르게 확장되는, 거의 무작위적인 샷건 발사 패턴과 유사하여 명명되었다.[1]
전통적으로 샷건 시퀀싱에는 생어 시퀀싱이 주로 사용되었다.[1] 이 방법은 DNA 중합효소에 의해 체인 종결 디데옥시뉴클레오티드가 선택적으로 삽입되는 것을 기반으로 한다.[1] 초기 대규모 게놈 염기서열 분석은 와 같은 모세관 염기서열 분석기를 통해 자동화되었다.[1]
최근에는 대규모 자동화된 게놈 분석에 고처리량 시퀀싱(NGS) 방법이 주로 활용된다. 생어 방법은 여전히 소규모 프로젝트나 긴 연속 DNA 서열 리드(>500 뉴클레오티드)를 얻는 데 사용된다.
'''고처리량 시퀀싱 (High-throughput Sequencing)'''
고처리량 시퀀싱(High-throughput Sequencing), 즉 차세대 염기서열 분석(NGS)은 수천 또는 수백만 개의 DNA 조각을 한 번에 생성하는 병렬화 분석 기술이다.[1] 이는 표준 염료 종결자 방법보다 DNA 시퀀싱 비용을 낮추기 위한 것이다.[1]
2000년대 초 생어의 해독법을 능가하는 차세대 해독기 기술이 발달했고, 그것이 NGS라고 통칭된다. 미국의 일루미나사는 NGS의 기술에서 가장 많은 해독기를 팔았고, 2019년 기준으로는 약 150 염기까지 읽는 Hi-seq과 Nova-seq계열의 해독기가 짧은 서열 해독의 주류를 이루고 있다. 미국의 Complete Genomics사는 하바드 조지 처치교수등의 특허를 활용하여, 일루미나사의 해독기와 경쟁되는 기게를 만들었는데, 중국의 BGI사에 인수되어 2018년부터 MGI-seq이란 해독기를 생산하고 있다.
과거에는 염기 서열 분석이 시퀀싱 센터라는 중앙 집중형 시설에서 이루어졌으나, 염기 서열 결정 기술이 발전하고 벤치탑형 시퀀서가 개발되면서, 평균적인 학술 연구실에서도 시퀀서를 사용할 수 있게 되었다.
'''3세대 염기서열 분석 기술'''
3세대 염기서열 분석 기술은 긴 DNA 서열을 생산할 수 있는 기술이다. 미국의 PacBio사가 비슷한 기술로 긴 서열을 생산하고 있으며, 이러한 긴 서열 해독기를 3세대 해독기라고 부르기도 한다. 현재 NGS중 가장 발전된 것은 나노포어 기술인데, 월터 길버드의 제자인 조지 처치 하바드대 교수가 발명하였고, 현재 영국의 Oxford Nanopore사가 라이센싱을 해서 상용화했다.
3. 1. 샷건 시퀀싱 (Shotgun Sequencing)
샷건 시퀀싱은 DNA를 무작위로 작은 조각으로 잘라낸 후, 각 조각의 염기서열을 분석하고, 중첩되는 부분을 찾아 이어 붙여 전체 염기서열을 완성하는 방법이다.[1] 1000 염기쌍보다 긴 DNA 서열 분석을 위해 설계된 이 방법은, 빠르게 확장되는, 거의 무작위적인 샷건 발사 패턴과 유사하여 명명되었다.[1]전통적으로 샷건 시퀀싱에는 생어 방법이 주로 사용되었다.[1] 이 방법은 DNA 중합효소에 의해 체인 종결 디데옥시뉴클레오티드가 선택적으로 삽입되는 것을 기반으로 한다.[1] 초기 대규모 게놈 염기서열 분석은 와 같은 모세관 염기서열 분석기를 통해 자동화되었다.[1]
하지만 최근에는 대규모 자동화된 게놈 분석에 고처리량 시퀀싱(NGS) 방법이 주로 활용된다. 생어 방법은 여전히 소규모 프로젝트나 긴 연속 DNA 서열 리드(>500 뉴클레오티드)를 얻는 데 사용된다.
배열 어셈블리는 대량의 DNA 서열 단편을 정렬하고 연결하여 원래 서열을 재구성하는 과정이다.[102]
3. 2. 고처리량 시퀀싱 (High-throughput Sequencing)
고처리량 시퀀싱(High-throughput Sequencing), 즉 차세대 염기서열 분석(NGS)은 수천 또는 수백만 개의 DNA 조각을 한 번에 생성하는 병렬화 분석 기술이다.[1][1] 이는 표준 염료 종결자 방법보다 DNA 시퀀싱 비용을 낮추기 위한 것이다.[1] 초고처리량 시퀀싱에서는 최대 500,000개의 시퀀스 합성 작동이 병렬로 실행될 수 있다.[1][1] NGS는 기존의 생어 시퀀싱보다 훨씬 빠르고 저렴하게 대량의 염기서열 데이터를 생산할 수 있다는 장점이 있다.일루미나(Illumina) 염료 시퀀싱은 가역 염료 종결자를 기반으로 하며, 1996년 파스칼 마이어와 로랑 파리넬리에 의해 개발되었다.[1] DNA 분자와 프라이머를 슬라이드에 부착하고 중합 효소로 증폭시켜 "DNA 콜로니"를 형성한다. 염기 서열 결정을 위해 네 가지 유형의 가역 종결자 염기(RT-염기)를 추가하고 비통합 뉴클레오티드를 제거한다. 효소 반응과 이미지 캡처를 분리하면 최적의 처리량을 얻을 수 있으며, 이론적으로 무제한의 시퀀싱 용량을 얻을 수 있다. 최적 구성을 사용하면 기기 처리량은 카메라의 A/D 변환 속도에만 의존한다.[1]
이온 반도체 시퀀싱(Ion Torrent)은 염기가 통합될 때마다 수소 이온의 방출을 측정하는 방식이다. 템플릿 DNA가 포함된 마이크로웰에 단일 뉴클레오티드를 채우면, 뉴클레오티드가 템플릿 가닥과 상보적일 경우 통합되고 수소 이온이 방출되며,이 방출은 ISFET 이온 센서를 트리거한다.[1]
2000년대 초 생어의 해독법을 능가하는 차세대 해독기 기술이 발달했고, 그것이 NGS라고 통칭된다. 현재 NGS중 가장 발전된게 나노포어기술이다, 월터 길버드의 수제자인 조지 처치 하바드 교수가 발명한 것으로, 현재 영국의 옥스포드나노포어사가 라이센싱을 해서 상용화했다. 미국의 일루미나사는 NGS의 기술에서 가장 많은 해독기를 팔았고, 2019년 기준으로는 약 150 염기까지 읽는 Hi-seq과 Nova-seq계열의 해독기가 짧은 서열 해독의 주류를 이루고 있다. 미국의 Complete Genomics사는 하바드 조지 처치교수등의 특허를 활용하여, 일루미나사의 해독기와 경쟁되는 기게를 만들었는데, 중국의 BGI사에 인수되어 2018년부터 MGI-seq이란 해독기를 생산하고 있다.
과거에는 염기 서열 분석이 시퀀싱 센터라는 중앙 집중형 시설에서 이루어졌으나, 염기 서열 결정 기술이 발전하고 벤치탑형 시퀀서가 개발되면서, 평균적인 학술 연구실에서도 시퀀서를 사용할 수 있게 되었다.
3. 3. 3세대 염기서열 분석 기술
3세대 염기서열 분석 기술은 긴 DNA 서열을 생산할 수 있는 기술이다. 미국의 PacBio사가 비슷한 기술로 긴 서열을 생산하고 있으며, 이러한 긴 서열 해독기를 3세대 해독기라고 부르기도 한다. 3세대 염기서열 분석 기술은 긴 DNA 분자를 자르지 않고 직접 읽어내는 기술로, 반복 서열, 구조 변이 등 기존 NGS 기술로 분석하기 어려운 영역의 해독에 유리하다. 대표적인 3세대 염기서열 분석 기술로는 PacBio의 SMRT 시퀀싱, Oxford Nanopore의 나노포어 시퀀싱 등이 있다. 현재 NGS중 가장 발전된 것은 나노포어 기술인데, 월터 길버드의 제자인 조지 처치 하바드대 교수가 발명하였고, 현재 영국의 옥스포드 나노포어사가 라이센싱을 해서 상용화했다.4. 유전체 분석
유기체가 선택된 후, 유전체 프로젝트는 DNA 염기서열 분석, 원본 염색체의 표현을 생성하기 위한 염기서열 조립, 그리고 해당 표현의 주석 및 분석의 세 가지 구성 요소를 포함한다.[1]
게놈 프로젝트
어떤 생물이 대상이 선택된 후, 다음 세 단계를 거친다.: 시퀀싱, 어셈블리, 어노테이션.
=== 시퀀스 어셈블리 (Sequence Assembly) ===
시퀀스 어셈블리(Sequence Assembly)는 원래 시퀀스를 재구성하기 위해 훨씬 더 긴 DNA 시퀀스의 단편을 시퀀스 정렬하고 병합하는 것을 말한다.[1] 현재의 DNA 시퀀싱 기술은 전체 게놈을 연속적인 시퀀스로 읽을 수 없기 때문에 이러한 과정이 필요하다. 대신, 사용된 기술에 따라 20~1000개의 염기 사이의 작은 조각을 읽는다. PacBio 또는 Oxford Nanopore와 같은 3세대 시퀀싱 기술은 일반적으로 10-100kb 길이의 시퀀싱 리드를 생성하지만 약 1%의 높은 오류율을 보인다.[1] 일반적으로 리드라고 불리는 짧은 단편은 샷건 시퀀싱 게놈 DNA 또는 유전자 전사체(EST)에서 발생한다.[1]
어셈블리는 크게 두 가지 접근 방식으로 분류할 수 있다. 하나는 과거에 시퀀싱된 것과 유사하지 않은 게놈을 위한 ''드 노보''(de novo) 어셈블리이고, 다른 하나는 조립 과정에서 밀접하게 관련된 유기체의 기존 시퀀스를 참조로 사용하는 비교 어셈블리이다.[1] 비교 어셈블리와 비교하여 ''드 노보'' 어셈블리는 계산적으로 어렵고(NP-hard), 단일 분자 시퀀싱(NGS) 기술에는 적합하지 않다. ''드 노보'' 어셈블리 패러다임 내에는 오일러 경로 전략과 오버랩-레이아웃-컨센서스(OLC) 전략의 두 가지 주요 조립 전략이 있다. OLC 전략은 궁극적으로 NP-hard 문제인 오버랩 그래프를 통해 해밀턴 경로를 생성하려고 시도한다. 오일러 경로 전략은 드 브루인 그래프를 통해 오일러 경로를 찾으려고 시도하기 때문에 계산적으로 더 다루기 쉽다.[1]
=== 유전체 주석 (Genome Annotation) ===
DNA 서열 조립 자체는 추가적인 분석 없이는 거의 가치가 없다.[1] 게놈 주석은 생물학적 정보를 DNA 서열에 부착하는 과정으로, 다음 세 가지 주요 단계로 구성된다.[1]
# 단백질을 암호화하지 않는 게놈의 부분을 식별
# 유전자 예측이라고 하는 과정인 게놈의 요소를 식별
# 이러한 요소에 생물학적 정보를 부착.
자동 주석 도구는 수동 주석(일명 큐레이션)과 달리 인간의 전문 지식과 잠재적인 실험적 검증을 포함하여 이러한 단계를 ''in silico''로 수행하려고 시도한다.[1] 이상적으로, 이러한 접근 방식은 동일한 주석 파이프라인에서 공존하고 서로를 보완한다.
전통적으로, 주석의 기본 수준은 유사성을 찾기 위해 BLAST를 사용하고, 상동성에 기반하여 게놈에 주석을 다는 것이다.[1] 최근에는 주석 플랫폼에 추가 정보가 추가되었다. 추가 정보를 통해 수동 주석자는 동일한 주석이 주어진 유전자 간의 불일치를 해결할 수 있다. 일부 데이터베이스는 게놈 컨텍스트 정보, 유사성 점수, 실험 데이터 및 기타 리소스 통합을 사용하여 Subsystems 접근 방식을 통해 게놈 주석을 제공한다. 다른 데이터베이스(예: Ensembl)는 수동으로 관리되는 데이터 소스와 자동 게놈 주석 파이프라인의 다양한 소프트웨어 도구에 의존한다.[1] ''구조 주석''은 게놈 요소, 주로 ORF 및 그 위치 또는 유전자 구조의 식별로 구성된다. ''기능 주석''은 게놈 요소에 생물학적 정보를 부착하는 것으로 구성된다.
4. 1. 시퀀스 어셈블리 (Sequence Assembly)
시퀀스 어셈블리(Sequence Assembly)는 원래 시퀀스를 재구성하기 위해 훨씬 더 긴 DNA 시퀀스의 단편을 시퀀스 정렬하고 병합하는 것을 말한다.[1] 현재의 DNA 시퀀싱 기술은 전체 게놈을 연속적인 시퀀스로 읽을 수 없기 때문에 이러한 과정이 필요하다. 대신, 사용된 기술에 따라 20~1000개의 염기 사이의 작은 조각을 읽는다. PacBio 또는 Oxford Nanopore와 같은 3세대 시퀀싱 기술은 일반적으로 10-100kb 길이의 시퀀싱 리드를 생성하지만 약 1%의 높은 오류율을 보인다.[1] 일반적으로 리드라고 불리는 짧은 단편은 샷건 시퀀싱 게놈 DNA 또는 유전자 전사체(EST)에서 발생한다.[1]어셈블리는 크게 두 가지 접근 방식으로 분류할 수 있다. 하나는 과거에 시퀀싱된 것과 유사하지 않은 게놈을 위한 ''드 노보''(de novo) 어셈블리이고, 다른 하나는 조립 과정에서 밀접하게 관련된 유기체의 기존 시퀀스를 참조로 사용하는 비교 어셈블리이다.[1] 비교 어셈블리와 비교하여 ''드 노보'' 어셈블리는 계산적으로 어렵고(NP-hard), 단일 분자 시퀀싱(NGS) 기술에는 적합하지 않다. ''드 노보'' 어셈블리 패러다임 내에는 오일러 경로 전략과 오버랩-레이아웃-컨센서스(OLC) 전략의 두 가지 주요 조립 전략이 있다. OLC 전략은 궁극적으로 NP-hard 문제인 오버랩 그래프를 통해 해밀턴 경로를 생성하려고 시도한다. 오일러 경로 전략은 드 브루인 그래프를 통해 오일러 경로를 찾으려고 시도하기 때문에 계산적으로 더 다루기 쉽다.[1]
4. 2. 유전체 주석 (Genome Annotation)
DNA 서열 조립 자체는 추가적인 분석 없이는 거의 가치가 없다.[1] 게놈 주석은 생물학적 정보를 DNA 서열에 부착하는 과정으로, 다음 세 가지 주요 단계로 구성된다.[1]# 단백질을 암호화하지 않는 게놈의 부분을 식별
# 유전자 예측이라고 하는 과정인 게놈의 요소를 식별
# 이러한 요소에 생물학적 정보를 부착.
자동 주석 도구는 수동 주석(일명 큐레이션)과 달리 인간의 전문 지식과 잠재적인 실험적 검증을 포함하여 이러한 단계를 ''in silico''로 수행하려고 시도한다.[1] 이상적으로, 이러한 접근 방식은 동일한 주석 파이프라인에서 공존하고 서로를 보완한다.
전통적으로, 주석의 기본 수준은 유사성을 찾기 위해 BLAST를 사용하고, 상동성에 기반하여 게놈에 주석을 다는 것이다.[1] 최근에는 주석 플랫폼에 추가 정보가 추가되었다. 추가 정보를 통해 수동 주석자는 동일한 주석이 주어진 유전자 간의 불일치를 해결할 수 있다. 일부 데이터베이스는 게놈 컨텍스트 정보, 유사성 점수, 실험 데이터 및 기타 리소스 통합을 사용하여 Subsystems 접근 방식을 통해 게놈 주석을 제공한다. 다른 데이터베이스(예: Ensembl)는 수동으로 관리되는 데이터 소스와 자동 게놈 주석 파이프라인의 다양한 소프트웨어 도구에 의존한다.[1] ''구조 주석''은 게놈 요소, 주로 ORF 및 그 위치 또는 유전자 구조의 식별로 구성된다. ''기능 주석''은 게놈 요소에 생물학적 정보를 부착하는 것으로 구성된다.
5. 유전체학의 연구 분야
5. 1. 기능 유전체학 (Functional Genomics)
기능 유전체학은 분자 생물학의 한 분야로, 유전체 프로젝트(예: 게놈 서열 분석 프로젝트)에 의해 생성된 방대한 데이터를 활용하여 유전자 (및 단백질)의 기능과 상호 작용을 설명하려고 시도한다. 기능 유전체학은 유전체의 정적인 측면, 즉 DNA 염기 서열이나 구조와는 대조적으로, 유전자 전사, 번역, 그리고 단백질-단백질 상호작용과 같은 동적인 측면에 초점을 맞춘다. 기능 유전체학은 유전자, RNA 전사체 및 단백질 산물 수준에서 DNA의 기능에 대한 질문에 답하려 한다.전체 게놈에 대한 지식은 다양한 조건에서의 유전자 발현 패턴에 주로 관련된 기능 유전체학 분야의 가능성을 창출했으며, 여기서 가장 중요한 도구는 마이크로어레이와 생물정보학이다.
5. 2. 구조 유전체학 (Structural Genomics)
구조 유전체학은 주어진 게놈에 의해 암호화된 모든 단백질의 3차원 구조를 설명하는 것을 목표로 한다.[1][1] 이 게놈 기반 접근 방식은 실험 및 모델링 접근 방식의 조합을 통해 고속 구조 결정을 가능하게 한다. 구조 유전체학은 특정 단백질에 집중하기보다는 게놈에 의해 암호화된 모든 단백질의 구조를 결정하려고 시도한다는 점이 전통적인 구조 예측과 다르다.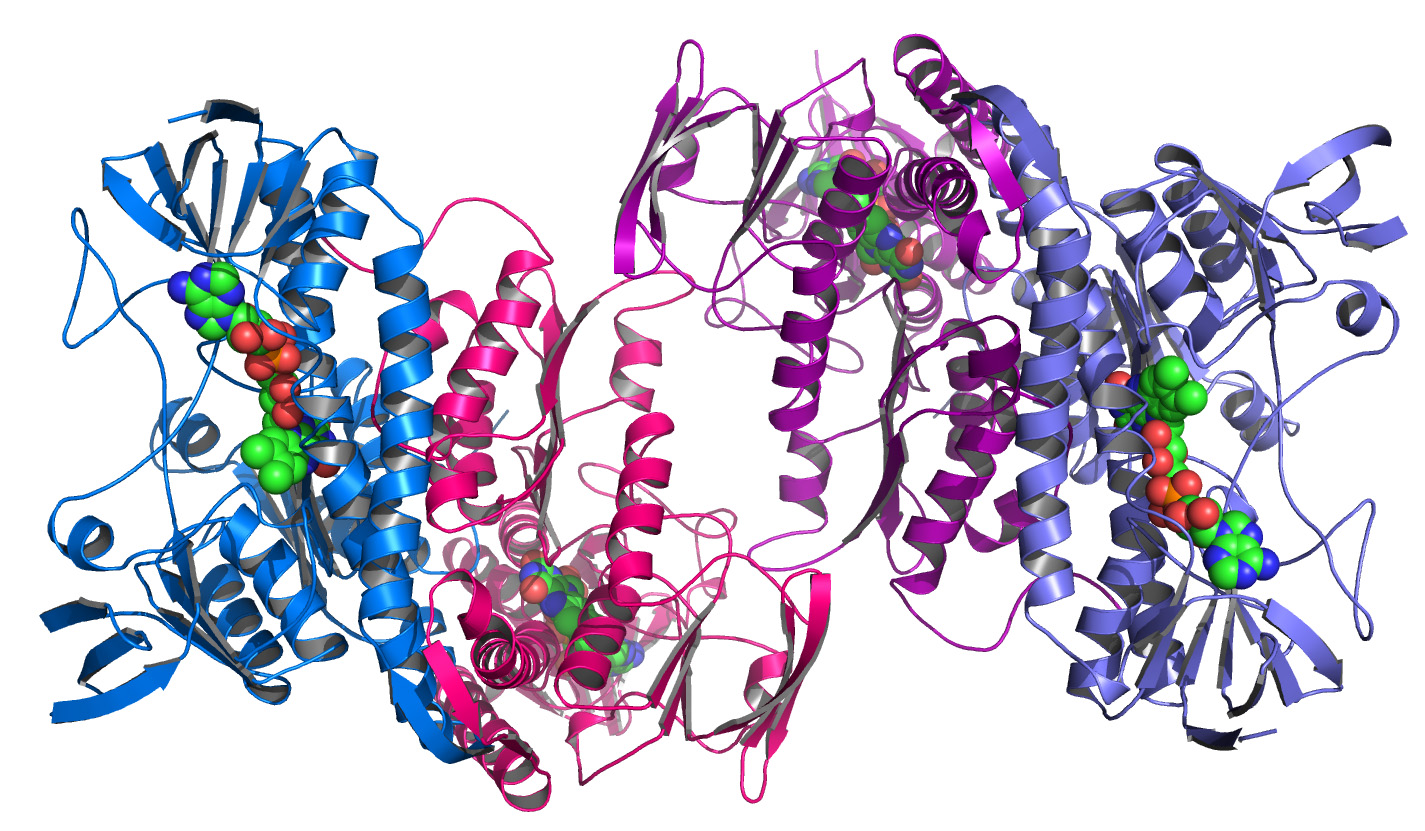
전체 게놈 서열이 사용 가능해짐에 따라, 특히 수많은 서열 게놈과 이전에 풀린 단백질 구조의 가용성이 과학자들이 이전에 풀린 상동체의 구조를 기반으로 단백질 구조를 모델링할 수 있게 해주기 때문에 실험 및 모델링 접근 방식을 결합하여 구조 예측을 더 빠르게 수행할 수 있다. 구조 유전체학은 게놈 서열을 사용한 실험적 방법이나, 알려진 구조의 단백질에 대한 서열 또는 구조적 상동성을 기반으로 하거나, 알려진 구조와의 상동성이 없는 단백질에 대한 화학적 및 물리적 원리를 기반으로 하는 모델링 기반 접근 방식을 포함하여, 구조 결정을 위한 다양한 접근 방식을 포함한다. 전통적인 구조 생물학과는 달리, 구조 유전체학적 노력을 통한 단백질 구조의 결정은 종종 (하지만 항상 그런 것은 아님) 단백질 기능에 대해 알려지기 전에 이루어진다. 이는 구조 생물정보학, 즉, 3D 구조로부터 단백질 기능을 결정하는 데 새로운 과제를 제기한다.[1]
5. 3. 후성유전체학 (Epigenomics)
후성유전체학은 후성유전 변형의 완전한 집합을 연구하는 학문으로, 세포의 유전 물질에 대한 연구를 하며, 이는 후성유전체로 알려져 있다.[1] 후성유전적 변형은 DNA 염기 서열을 변경하지 않고 유전자 발현에 영향을 미치는 세포의 DNA 또는 히스톤에 대한 가역적인 변형이다 (Russell 2010, p. 475). 가장 잘 특징 지어지는 두 가지 후성유전적 변형은 DNA 메틸화와 히스톤 변형이다.[2] 후성유전적 변형은 유전자 발현과 조절에 중요한 역할을 하며, 분화/발달[3] 및 종양 발생과 같은 수많은 세포 과정에 관여한다.[1] 전반적인 수준에서 후성유전학 연구는 최근에 유전체학적 고처리량 분석법의 적용을 통해 가능해졌다.[1]5. 4. 메타게노믹스 (Metagenomics)
메타게노믹스는 환경 샘플에서 직접 회수된 ''메타게놈'', 즉 유전 물질을 연구하는 분야이다. 이 광범위한 분야는 환경 유전체학, 생태 유전체학 또는 군집 유전체학이라고도 불린다.[1] 전통적인 미생물학 및 미생물 게놈 시퀀싱은 배양된 클론 미생물 배양에 의존하지만, 초기 환경 유전자 시퀀싱은 특정 유전자(종종 16S rRNA 유전자)를 클로닝하여 자연 샘플에서 다양성 프로파일을 생성했다.[1] 이러한 연구를 통해 미생물 생물 다양성의 대다수가 배양 기반 방법으로 놓치고 있다는 사실이 밝혀졌다.[1]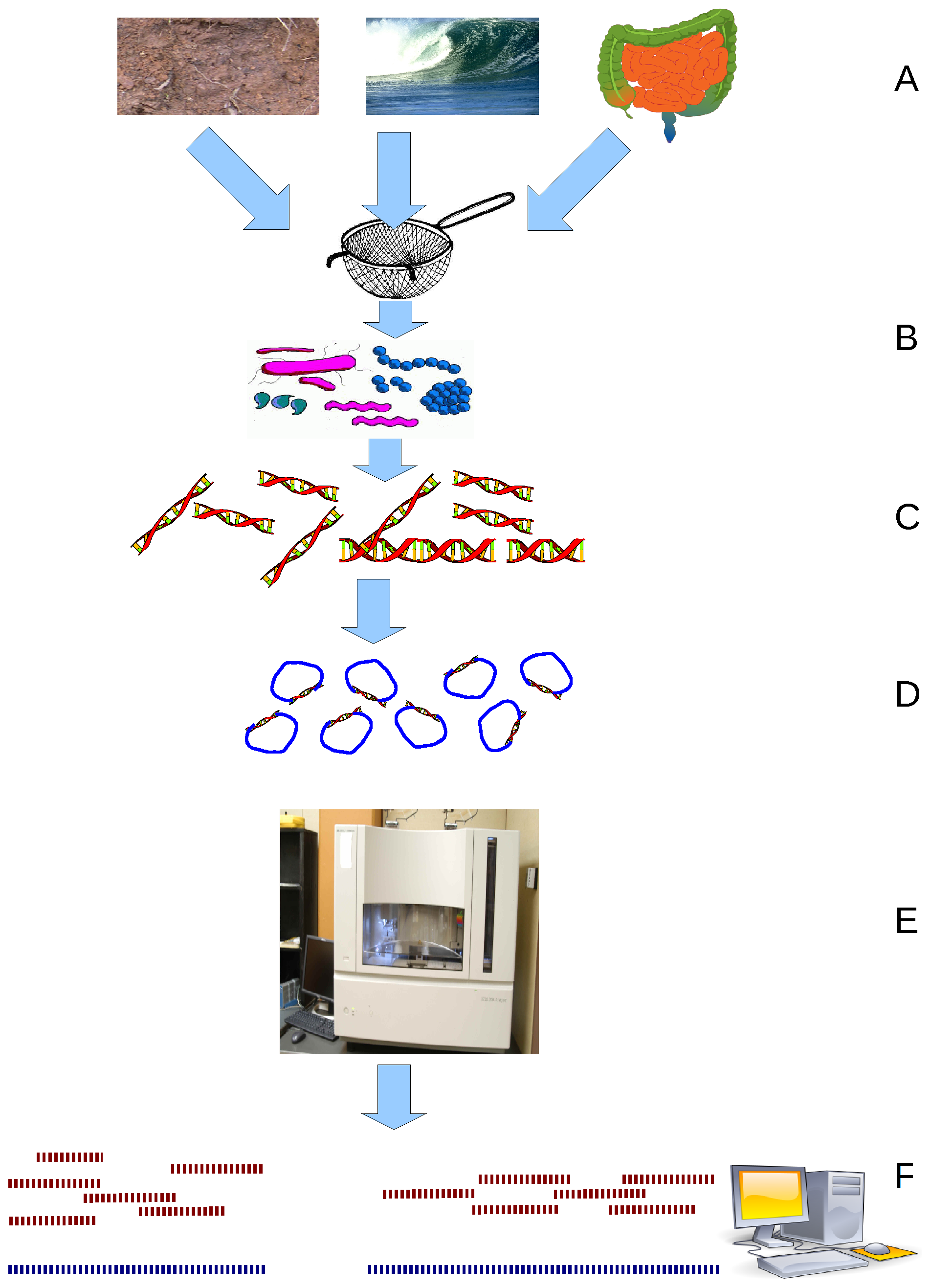
최근 연구에서는 "샷건" 생거 시퀀싱 또는 대규모 병렬 파이로시퀀싱을 사용하여 샘플링된 군집의 모든 구성원으로부터 모든 유전자의 대체로 편향되지 않은 샘플을 얻는다.[1] 메타게노믹스는 이전에 숨겨져 있던 미생물 생명의 다양성을 드러내는 강력한 힘을 가지고 있기 때문에 전체 생명 세계에 대한 이해를 혁신할 수 있는 잠재력을 가진 미생물 세계를 볼 수 있는 강력한 렌즈를 제공한다.[1][1]
6. 유전체학의 응용
유전체학은 의학, 생명공학, 인류학 및 기타 사회 과학을 포함한 많은 분야에서 응용 분야를 제공했다.[1]
==== 의학 및 보건 ====
차세대 유전체 기술 덕분에 임상의와 생의학 연구자들은 대규모 연구 집단에서 수집되는 유전체 데이터의 양을 획기적으로 늘릴 수 있게 되었다.[1] 유전체 데이터를 질병 연구에서 다양한 종류의 데이터와 통합하는 새로운 정보학적 접근 방식은 약물 반응 및 질병의 유전적 기반을 더 잘 이해할 수 있도록 돕는다.[1]
이안 애슐리가 이끄는 스탠포드 팀은 인간 유전체를 의학적으로 해석하기 위한 최초의 도구를 개발하는 등 유전체를 의학에 적용하려는 초기 노력을 기울였다. 브리검 여성 병원, 브로드 연구소, 하버드 의과대학의 Genomes2People 연구 프로그램은 2012년에 유전체학을 건강에 접목하는 경험적 연구를 수행하기 위해 설립되었다. 브리검 여성 병원은 2019년 8월에 예방 유전체 클리닉을 개설했고, 매사추세츠 종합 병원이 한 달 후에 뒤따랐다. ''All of Us'' 연구 프로그램은 정밀 의학 연구 플랫폼의 중요한 구성 요소가 되기 위해 100만 명의 참가자로부터 유전자 서열 데이터를 수집하는 것을 목표로 한다. ''영국 바이오뱅크'' 이니셔티브는 깊이 있는 유전체 및 표현형 데이터를 가진 50만 명 이상의 개인을 연구했다.[4]
==== 생명공학 ====
유전체학 지식의 증가는 합성 생물학의 더욱 정교한 응용을 가능하게 했다.[1] 2010년 J. 크레이그 벤터 연구소의 연구자들은 ''마이코플라스마 제니탈리움(Mycoplasma genitalium)''의 유전체에서 파생된 부분적으로 합성된 세균 종인 ''마이코플라스마 라보라토리움(Mycoplasma laboratorium)''의 생성을 발표했다.[1] 유전체학은 유용 유전자 발굴, 유전자 조작 등 생명공학 분야 발전에 기여하고 있으며, 유전체 정보를 바탕으로 유용 미생물, 식물, 동물을 개발하고, 새로운 바이오 소재, 바이오 연료 등을 생산하는 연구가 진행되고 있다.
6. 1. 의학 및 보건
차세대 유전체 기술 덕분에 임상의와 생의학 연구자들은 대규모 연구 집단에서 수집되는 유전체 데이터의 양을 획기적으로 늘릴 수 있게 되었다.[1] 유전체 데이터를 질병 연구에서 다양한 종류의 데이터와 통합하는 새로운 정보학적 접근 방식은 약물 반응 및 질병의 유전적 기반을 더 잘 이해할 수 있도록 돕는다.[1]이안 애슐리가 이끄는 스탠포드 팀은 인간 유전체를 의학적으로 해석하기 위한 최초의 도구를 개발하는 등 유전체를 의학에 적용하려는 초기 노력을 기울였다. 브리검 여성 병원, 브로드 연구소, 하버드 의과대학의 Genomes2People 연구 프로그램은 2012년에 유전체학을 건강에 접목하는 경험적 연구를 수행하기 위해 설립되었다. 브리검 여성 병원은 2019년 8월에 예방 유전체 클리닉을 개설했고, 매사추세츠 종합 병원이 한 달 후에 뒤따랐다. ''All of Us'' 연구 프로그램은 정밀 의학 연구 플랫폼의 중요한 구성 요소가 되기 위해 100만 명의 참가자로부터 유전자 서열 데이터를 수집하는 것을 목표로 한다. ''영국 바이오뱅크'' 이니셔티브는 깊이 있는 유전체 및 표현형 데이터를 가진 50만 명 이상의 개인을 연구했다.[4]
6. 2. 생명공학
유전체학 지식의 증가는 합성 생물학의 더욱 정교한 응용을 가능하게 했다.[1] 2010년 J. 크레이그 벤터 연구소의 연구자들은 ''마이코플라스마 제니탈리움(Mycoplasma genitalium)''의 유전체에서 파생된 부분적으로 합성된 세균 종인 ''마이코플라스마 라보라토리움(Mycoplasma laboratorium)''의 생성을 발표했다.[1] 유전체학은 유용 유전자 발굴, 유전자 조작 등 생명공학 분야 발전에 기여하고 있으며, 유전체 정보를 바탕으로 유용 미생물, 식물, 동물을 개발하고, 새로운 바이오 소재, 바이오 연료 등을 생산하는 연구가 진행되고 있다.6. 3. 농업 및 축산업
6. 4. 법의학
6. 5. 인류학 및 진화학
6. 6. 보존 유전체학 (Conservation Genomics)
집단 유전체학은 유전체 염기 서열 분석 방법을 사용하여, 집단 간의 DNA 염기 서열을 대규모로 비교하는 연구 분야이다.[1] 경관 유전체학은 환경과 유전적 변이 패턴 간의 관계를 식별한다.[1]보존 과학자들은 유전체 염기 서열 분석을 통해 수집된 정보를 사용하여, 종의 보존에 중요한 유전적 요소를 더 잘 평가할 수 있다.[1] 유전체 데이터를 사용하여 진화 과정의 영향을 평가하고 주어진 집단 전체의 변이 패턴을 감지함으로써, 보존 과학자들은 주어진 종을 지원하는 계획을 수립할 수 있다.[1]
7. 한국의 유전체학 연구
7. 1. 한국의 주요 유전체 연구 기관 및 프로젝트
8. 윤리적, 법적, 사회적 문제 (ELSI)
참조
[1]
논문
A high-resolution protein architecture of the budding yeast genome
2021-03-01
[2]
논문
Phase separation directs ubiquitination of gene-body nucleosomes
2020-03-01
[3]
논문
Delayed puberty, gonadotropin abnormalities and subfertility in male Padi2/Padi4 double knockout mice
2022-10-01
[4]
논문
The UK Biobank resource with deep phenotyping and genomic data
2018-10-01
[5]
논문
An integrated map of genetic variation from 1,092 human genomes
2012-11-01
[6]
논문
Genomics and the future of conservation genetics
2010-10-01
[7]
논문
Sequence and organization of the human mitochondrial genome
1981-04-01
[8]
논문
Shotgun DNA sequencing using cloned DNase I-generated fragments
1981-07-01
[9]
논문
Sequencing the genome from nematode to human: changing methods, changing science
2003-06-01
[10]
논문
Clinical assessment incorporating a personal genome
2010-05-01
[11]
논문
Synthetic genomes: The next step for the synthetic genome
2011-05-01
[12]
웹사이트
Benchtop sequencers ship off
http://blogs.nature.[...]
2012-12-22
[13]
서적
Genomes and what to make of them
University of Chicago Press
[14]
뉴스
Human gene number slashed
http://news.bbc.co.u[...]
2012-12-21
[15]
서적
Bioinformatics
[16]
논문
Expectations from structural genomics
2000-01-01
[17]
논문
A tour of structural genomics
2001-10-01
[18]
논문
Steady progress and recent breakthroughs in the accuracy of automated genome annotation
http://www.genomics.[...]
2013-01-04
[19]
논문
Prophage genomics
2003-06-01
[20]
논문
Genomics. Genome project standards in a new era of sequencing
2009-10-01
[21]
논문
Making the most of "omics" for symbiosis research
2012-08-01
[22]
논문
Genomes for all
2006-01-01
[23]
서적
Regenesis: how synthetic biology will reinvent nature and ourselves
Basic Books
[24]
논문
Rise, fall and resurrection of chromosome territories: a historical perspective. Part I. The rise of chromosome territories
https://pubmed.ncbi.[...]
[25]
간행물
Genomics
https://archive.org/[...]
Macmillan Reference USA
2002-11-08
[26]
서적
The Stanford Encyclopedia of Philosophy
[27]
웹사이트
Two Boston Health Systems Enter the Growing Direct-to-Consumer Gene Sequencing Market by Opening Preventative Genomics Clinics, but Can Patients Afford the Service?
https://www.darkdail[...]
The Dark Intelligence Group
2020-01-03
[28]
논문
Powering Preventative Medicine
http://www.bio-itwor[...]
2014-12-17
[29]
논문
Phased whole-genome genetic risk in a family quartet using a major allele reference sequence
2011-09-01
[30]
논문
Clinical interpretation and implications of whole-genome sequencing
2014-03-01
[31]
특허
Method of nucleic acid amplification
http://www.google.co[...]
[32]
논문
Environmental shotgun sequencing: its potential and challenges for studying the hidden world of microbes
2007-03-01
[33]
논문
Badomics words and the power and peril of the ome-meme
2012-07-01
[34]
논문
Ensembl 2013
2013-01-01
[35]
웹사이트
Genome Project Statistics
https://www.ncbi.nlm[...]
2011-11-18
[36]
논문
Genomics, health care, and society
2011-09
[37]
논문
Genomics of cardiovascular disease
2011-12
[38]
논문
Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene
1976-04
[39]
논문
Complete nucleotide sequence of SV40 DNA
1978-05
[40]
논문
Whole-genome random sequencing and assembly of Haemophilus influenzae Rd
1995-07
[41]
논문
Phage_Finder: automated identification and classification of prophage regions in complete bacterial genome sequences
2006-11
[42]
서적
Epigenetics: the ultimate mystery of inheritance
WW Norton
2011
[43]
논문
Challenges and opportunities of genetic approaches to biological conservation
2010-09-01
[44]
논문
Molecular configuration in sodium thymonucleate
1953-04
[45]
논문
A phylogeny-driven genomic encyclopaedia of Bacteria and Archaea
2009-12
[46]
논문
A new method for sequencing DNA
1977-02
[47]
논문
Life with 6000 genes
1996-10
[48]
논문
Advanced sequencing technologies and their wider impact in microbiology
2007-05
[49]
서적
The Cyanobacteria: Molecular Biology, Genomics and Evolution
http://www.horizonpr[...]
Caister Academic Press
[50]
논문
Nucleotide sequences in the yeast alanine transfer ribonucleic acid
1965-05
[51]
논문
Structure of a Ribonucleic Acid
1965-03
[52]
논문
Impact of culture-independent studies on the emerging phylogenetic view of bacterial diversity
1998-09
[53]
간행물
An Introduction to Next-Generation Sequencing Technology
http://www.illumina.[...]
Illumina, Inc.
2012-02-28
[54]
논문
Genomics and proteomics in solving brain complexity
2013-07
[55]
논문
Whole genome shotgun sequencing guided by bioinformatics pipelines--an optimized approach for an established technique
2003-12
[56]
서적
Concepts of genetics
Pearson Education
[57]
논문
Principles and challenges of genomewide DNA methylation analysis
2010-03
[58]
서적
Intermediate Greek-English Lexicon
Martino Fine Books
2013
[59]
논문
Personalized medicine and human genetic diversity
2014-07
[60]
논문
The power and promise of population genomics: from genotyping to genome typing
2003-12
[61]
서적
Metagenomics: Theory, Methods and Applications
Caister Academic Press
[62]
서적
Metagenomics: Current Innovations and Future Trends
Caister Academic Press
[63]
논문
Next-generation DNA sequencing methods
http://www.lcg.unam.[...]
[64]
논문
Towards a comprehensive structural coverage of completed genomes: a structural genomics viewpoint
2007-03
[65]
논문
The fast changing landscape of sequencing technologies and their impact on microbial genome assemblies and annotation
[66]
논문
An interdependent metabolic patchwork in the nested symbiosis of mealybugs
2011-08
[67]
서적
Drawing the map of life: inside the Human Genome Project
https://archive.org/[...]
Basic Books
[68]
서적
Bacteriophage: Genetics and Molecular Biology
http://www.horizonpr[...]
Caister Academic Press
[69]
논문
Nucleotide sequence of the gene coding for the bacteriophage MS2 coat protein
1972-05
[70]
웹사이트
home
https://nanoporetech[...]
[71]
논문
Genomics: In search of rare human variants
2010-10
[72]
웹사이트
Dog Genome Assembled: Canine Genome Now Available to Research Community Worldwide
http://www.genome.go[...]
2004-07-14
[73]
웹사이트
NIH-funded genome centers to accelerate precision medicine discoveries
https://www.nih.gov/[...]
National Institutes of Health
2018-09-25
[74]
논문
RNA codewords and protein synthesis, VII. On the general nature of the RNA code
1965-05
[75]
논문
A decade of GigaScience: A perspective on conservation genetics
2022-06
[76]
간행물
Genome, n
http://www.oed.com/v[...]
Oxford University Press
2008
[77]
논문
Chloroplast gene organization deduced from complete sequence of liverwort Marchantia polymorpha chloroplast DNA
[78]
논문
The complete DNA sequence of yeast chromosome III
1992-05
[79]
웹사이트
Home
https://www.pacb.com[...]
[80]
서적
Bioinformatics and functional genomics
https://archive.org/[...]
Wiley-Blackwell
[81]
논문
Genome assembly reborn: recent computational challenges
2009-07
[82]
논문
A tale of three next generation sequencing platforms: comparison of Ion Torrent, Pacific Biosciences and Illumina MiSeq sequencers
2012-07
[83]
웹사이트
Top U.S. medical centers roll out DNA sequencing clinics for healthy (and often wealthy) clients
https://www.statnews[...]
2019-08-16
[84]
논문
A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase
1975-05
[85]
논문
Nucleotide sequence of bacteriophage phi X174 DNA
1977-02
[86]
논문
DNA sequencing with chain-terminating inhibitors
1977-12
[87]
웹사이트
Nobel lecture: Determination of nucleotide sequences in DNA
http://nobelprize.or[...]
Nobelprize.org
2010-10-18
[88]
논문
Theodor and Marcella Boveri: chromosomes and cytoplasm in heredity and development
2008-03
[89]
뉴스
Data Deluge
http://www.the-scien[...]
2011-10-01
[90]
논문
The complete nucleotide sequence of the tobacco chloroplast genome: its gene organization and expression
1986-09
[91]
논문
A strategy of DNA sequencing employing computer programs
1979-06
[92]
논문
Genome annotation: from sequence to biology
2001-07
[93]
서적
Principles of genetics
McGraw Hill
[94]
논문
Keeping up with the next generation: massively parallel sequencing in clinical diagnostics
2008-11
[95]
논문
Massively parallel sequencing: the next big thing in genetic medicine
2009-08
[96]
웹사이트
Complete genomes: Viruses
https://www.ncbi.nlm[...]
2011-11-17
[97]
웹사이트
WHO definitions of genetics and genomics
https://www.who.int/[...]
World Health Organization
[98]
뉴스
Here"s an Omical Tale: Scientists Discover Spreading Suffix
https://www.wsj.com/[...]
2012-08-13
[99]
논문
The wholeness in suffix -omics, -omes, and the word om
2007-12
[100]
논문
The complete nucleotide sequence of the mitochondrial genome of Tetraodon nigroviridis
2006-04
[101]
뉴스
Scientists Start a Genomic Catalog of Earth's Abundant Microbes
https://www.nytimes.[...]
2009-12-29
[102]
서적
"ビッグデータ時代のゲノミクス情報処理"
コロナ社
2014-10
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com

