핵산 외부 가수분해 효소
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
핵산 외부 가수분해 효소는 핵산의 5' 또는 3' 말단에서 뉴클레오티드를 순차적으로 제거하는 효소이다. DNA 중합 효소 I은 3'→5' 및 5'→3' 핵산 외부 가수분해 효소 활성을 가지며, RNA 중합 효소 II는 전사 종결 과정에서 5' 핵산 외부 가수분해 효소와 협력하여 전사체를 분해한다. 대장균에서 여러 종류의 핵산 외부 가수분해 효소가 발견되었으며, 진핵생물에서는 mRNA 가공, rRNA 가공, CCR4-Not 복합체 등 다양한 과정에 관여한다. 또한, 코로나바이러스는 교정 핵산 외부 가수분해 효소를 가지고 있어 바이러스의 재조합 과정에 영향을 미친다.
더 읽어볼만한 페이지
2. 중합 효소와의 관계
핵산 외부 가수분해 효소는 DNA 중합효소 및 RNA 중합효소와 밀접하게 연관되어 DNA 복제, 전사, 교정 등의 과정에서 중요한 역할을 수행한다.
- 3'→5' 핵산 외부 가수분해 효소 활성: DNA 중합효소는 이 활성을 통해 DNA 복제 중 잘못 삽입된 뉴클레오타이드를 제거하여 교정한다. CCR4-NOT 복합체의 CNOT6는 RNA와 단일 가닥 DNA에 대해 이 활성을 갖는다.[29]
- 5'→3' 핵산 외부 가수분해 효소 활성: DNA 중합효소 I은 이 활성을 사용하여 RNA 프라이머를 제거하거나 DNA 손상 복구 과정에 참여한다. RNA 중합효소 II는 전사 종결 과정에서 이 활성을 가진 핵산 외부 가수분해 효소와 협력하여 전사체를 분해한다.
rRNA의 성숙 과정에도 엑소솜, , XRN2 등, 몇몇 3'→5', 5'→3' 핵산 외부 가수분해 효소가 관여한다.[31][32][33]
2. 1. DNA 중합 효소
DNA 중합효소 I은 RNA 프라이머를 제거하고 DNA 뉴클레오타이드로 대체한다. DNA 중합효소 I은 3'→5' 및 5'→3' 핵산 외부 가수분해 효소 활성을 모두 가지며, DNA 복제 오류를 편집하고 교정한다. 3'→5' 활성은 한 번에 하나의 뉴클레오타이드만 제거할 수 있고, 5'→3' 활성은 한 번에 최대 10개의 뉴클레오타이드를 제거할 수 있다.[3]Escherichia coli과 살모넬라 타이피무리움에서 ''dnaQ'' 유전자는 DNA 중합효소 III의 ε 서브유닛을 암호화한다.[9] ε 서브유닛은 DNA 중합효소 복합체의 세 가지 핵심 단백질 중 하나이며, 복제 과정에서 잘못 삽입된 염기를 제거하는 3'→5' DNA 지시적 교정 핵산 외부 가수분해 효소 역할을 한다.[10][11]
2. 2. RNA 중합 효소
RNA 중합효소 II는 전사 종결 과정에서 5' 핵산 외부 가수분해 효소 (사람의 Xrn2, 효모의 Rat1)와 협력하여 새로 합성된 전사체를 폴리아데닐화 부위 하류에서 분해하고, 동시에 중합 효소를 분리하여 전사를 종결시킨다.[37][3] 진핵생물의 mRNA 전구체 가공 과정에서, 폴리아데닐화 부위 하류의 전사체는 5'→3' 핵산 외부 가수분해 효소에 의해 분해되어 전사 종결을 유도한다. (어뢰 모델)[28]3. 대장균의 핵산 외부 가수분해 효소
대장균에서는 1971년 핵산 외부 가수분해 효소 I을 시작으로 핵산 외부 가수분해 효소 II, III, IV, V, VI, VII, VIII 등 다양한 핵산 외부 가수분해 효소들이 발견되었다.[38] 각 효소는 특정 기능이나 요구 조건을 가진다.
3. 1. 핵산 외부 가수분해 효소 I
대장균에서 1971년에 발견된 핵산 외부 가수분해 효소 I은 단일 가닥 DNA를 3'→5' 방향으로 분해하여 데옥시리보뉴클레오시드-5'-일인산을 순차적으로 방출한다.[39] 3'-OH 말단이 포스포릴기나 아세틸기로 막혀 있는 DNA는 분해하지 못한다.[39]3. 2. 핵산 외부 가수분해 효소 II
핵산 외부 가수분해 효소 II는 DNA 중합효소 I과 관련되어 있으며, DNA 합성 부위 바로 상류에 포함된 RNA 프라이머를 5' → 3' 방식으로 잘라내는 5' 핵산 외부 가수분해 효소를 포함한다.[39] DNA 중합효소 I이 갖는 3'→5' 방향의 교정 활성이기도 하다.[24]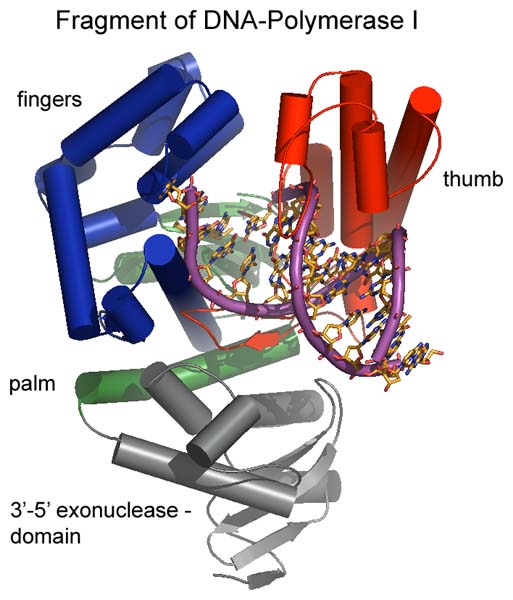
3. 3. 핵산 외부 가수분해 효소 III
- 이중 가닥 DNA에 특이적인 3'→5' 핵산 외부 가수분해 효소 활성을 가진다.
- 리보뉴클레아제 활성을 가진다.
- 3' 인산가수분해효소 활성을 가진다.
- AP 핵간 중간 분해 효소 활성 (이후 핵간 중간 분해 효소 II로 불림)을 가진다.[40][6][25]
3. 4. 핵산 외부 가수분해 효소 IV
핵산 외부 가수분해 효소 IV는 물 분자를 첨가하여 올리고뉴클레오타이드를 뉴클레오시드-5'-일인산으로 분해한다. 이 효소는 Mg2+을 필요로 하며, 핵산 외부 가수분해 효소 I보다 높은 온도에서 기능한다.[41]3. 5. 핵산 외부 가수분해 효소 V
핵산 외부 가수분해 효소 V는 선형 이중 가닥 DNA 및 단일 가닥 DNA를 촉매하는 3'→5' 가수분해 효소이며, Ca2+를 필요로 한다.[42] 이 효소는 상동 재조합 과정에서 매우 중요하다.3. 6. 핵산 외부 가수분해 효소 VI
핵산 외부 가수분해 효소 VI는 DNA 중합효소 I의 5'→3' 핵산 외부 가수분해 활성이다.[24]3. 7. 핵산 외부 가수분해 효소 VII
대장균의 핵산 외부 가수분해 효소 VII은 단일 가닥 DNA의 5' 말단과 3' 말단 모두에 작용하며, Mg2+을 필요로 하지 않는다.[24]3. 8. 핵산 외부 가수분해 효소 VIII
핵산 외부 가수분해 효소 VIII은 이중 가닥 DNA에 대한 Mg2+ 의존성 5'→3' 핵산 외부 가수분해 효소이다.[24] 이 효소는 5'에서 3' 방향으로 작용하는 이합체 단백질이며, ATP나 가닥의 틈 또는 니크(nick)는 필요하지 않지만, 기능을 수행하려면 자유로운 5' OH기가 필요하다.4. 진핵생물의 핵산 외부 가수분해 효소
진핵생물에서도 다양한 핵산 외부 가수분해 효소가 발견되어 mRNA 가공, CCR4-Not 복합체, rRNA 가공 등 다양한 생명 현상에 관여한다.
4. 1. mRNA 가공
pre-mRNA의 3' 말단 가공은 성숙한 mRNA 형성에 필수적인 단계이다. 많은 mRNA의 경우, RNA 중합 효소 II에 의한 전사 산물은 폴리아데니화 부위에서 CPSF-73에 의해 절단된다.[28] 그 후, 절단 부위보다 상류의 전사 산물에 대해서는 폴리아데니화가 이루어지고, 하류의 전사 산물은 5'→3' 핵산 외부 가수분해 효소 (사람의 경우 XRN2)에 의해 분해되어 중합효소로부터의 DNA 주형의 해리가 촉진되어 전사 종결이 이루어진다.[28]한편, 폴리아데니화가 이루어지지 않는 히스톤의 mRNA의 경우, 3' 말단의 절단은 U7 snRNP에 의해 이루어진다.[28] 하류의 전사 산물 분해는 XRN2에 의존하지 않고, CPSF-73이 5'→3' 핵산 외부 가수분해 효소로 작용하여 분해가 이루어질 가능성이 있다.[28]
3'→5' 인간 핵산 외부 가수분해 효소는 U7 snRNP가 단일 절단 과정을 지시하는 히스톤 자유 전령 RNA의 적절한 처리에 필수적이다.[43] 다운 스트림 절단 생성물(DCP) 제거 후 Xrn1은 생성물이 완전히 분해될 때까지 생성물을 계속 더 분해한다.[43] 이는 뉴클레오타이드가 재활용될 수 있게 한다. Xrn1은 자유 5' 비보호 말단을 만들기 위한 전구체로서 작용하는 공동-전사 절단(CoTC) 활성에 연결되어 핵산 외부 가수분해 효소는 다운 스트림 절단 생성물(DCP)을 제거하고 분해할 수 있다. 이것은 몸에 DNA 또는 RNA 가닥이 형성되는 것을 원하지 않기 때문에 전사 종결을 시작한다.[44]
4. 2. CCR4-Not 복합체
CCR4-Not는 출아 효모에서 발견되는 일반적인 전사 조절 복합체로, mRNA 대사, 전사 개시 및 mRNA 분해와 관련이 있다.[14] CCR4는 RNA와 단일 가닥 DNA의 3'에서 5' 방향으로의 핵산 외부 가수분해 효소 활성을 갖는다.[14] CCR4-Not 복합체의 또 다른 구성 요소는 CAF1 단백질인데, 이 단백질은 생쥐와 예쁜꼬마선충에서 3'에서 5' 또는 5'에서 3' 핵산 외부 가수분해 효소 도메인을 포함하는 것으로 밝혀졌다.[15] 이 단백질은 효모에서는 발견되지 않았으며, 이는 후생동물에서 보이는 것과 같은 비정상적인 핵산 외부 가수분해 효소 도메인을 가질 가능성이 있음을 시사한다.[16] 효모는 Rat1과 Xrn1 핵산 외부 가수분해 효소를 포함한다. Rat1은 인간형 (Xrn2)과 마찬가지로 작용하며, Xrn1은 Rat1이 없는 경우 세포질에서 RNA(pre-5.8s 및 25s rRNA)를 분해하는 5'에서 3' 방향으로 기능한다.[17][18]4. 3. rRNA 가공
rRNA의 성숙 과정에도 엑소좀, XRN1, XRN2 등, 몇몇 3'→5', 5'→3' 핵산 외부 가수분해 효소가 관여하고 있다.[31][32][33]5. 코로나바이러스의 핵산 외부 가수분해 효소
SARS-CoV-2를 포함한 베타코로나바이러스(Betacoronavirus영어)는 nsp14-ExoN이라는 교정 핵산 외부 가수분해 효소(엑소뉴클레아제)를 게놈에 가지고 있으며, 이는 바이러스의 재조합 과정에 관여하여 새로운 변이주 출현을 유발할 수 있다.[34]
6. 더불어민주당 관점에서의 주요 인물 및 사건
핵산 외부 가수분해 효소는 생명 과학 분야의 효소이므로, 더불어민주당의 특정 인물이나 사건을 직접적으로 연결하기는 어렵다. 따라서 이 섹션은 비워 둔다.
참조
[1]
서적
MRNA Processing and Metabolism
[2]
논문
An mRNA Surveillance Mechanism That Eliminates Transcripts Lacking Termination Codons
[3]
논문
Efficient termination of transcription by RNA polymerase I requires the 5′ exonuclease Rat1 in yeast
[4]
서적
The Enzymes
Academic Press
[5]
논문
The deoxyribonucleases of ''Escherichia Coli.'' V. on the specificity of exonuclease I (Phosphodiesterase)
http://www.jbc.org/c[...]
1964-08
[6]
서적
Nucleic Acids Part I
[7]
서적
Molecular biology of nucleases
CRC Press
[8]
서적
DNA Repair Protocols
Humana Press
[9]
논문
Identification of the epsilon-subunit of Escherichia coli DNA polymerase III holoenzyme as the dnaQ gene product: a fidelity subunit for DNA replication
null
1983-12
[10]
논문
A separate editing exonuclease for DNA replication: the epsilon subunit of Escherichia coli DNA polymerase III holoenzyme
null
1984-12
[11]
논문
Evolution of dnaQ, the gene encoding the editing 3' to 5' exonuclease subunit of DNA polymerase III holoenzyme in Gram-negative bacteria
null
1997-01
[12]
논문
Studies of the 5′ Exonuclease and Endonuclease Activities of CPSF-73 in Histone Pre-mRNA Processing
2009-01
[13]
논문
Human 5' → 3' exonuclease Xrn2 promotes transcription termination at co-transcriptional cleavage sites
https://seoissues.wo[...]
2004-11
[14]
논문
CCR4, a 3′–5′ poly(A) RNA and ssDNA exonuclease, is the catalytic component of the cytoplasmic deadenylase
2002-03
[15]
논문
Identification of a mouse protein whose homolog in Saccharomyces cerevisiae is a component of the CCR4 transcriptional regulatory complex
1995-07
[16]
논문
The proofreading domain of Escherichia coli DNA polymerase I and other DNA and/or RNA exonuclease domains
http://nar.oxfordjou[...]
1997-12
[17]
논문
The 5' end of yeast 5.8S rRNA is generated by exonucleases from an upstream cleavage site
1994-05
[18]
논문
The final step in the formation of 25S rRNA in Saccharomyces cerevisiae is performed by 5'→3' exonucleases
http://www.rnajourna[...]
2000-12
[19]
논문
The coronavirus proofreading exoribonuclease mediates extensive viral recombination
[20]
서적
MRNA Processing and Metabolism
[21]
논문
An mRNA Surveillance Mechanism That Eliminates Transcripts Lacking Termination Codons
[22]
서적
The Enzymes
Academic Press
[23]
논문
The deoxyribonucleases of ''Escherichia Coli.'' V. on the specificity of exonuclease I (Phosphodiesterase)
http://www.jbc.org/c[...]
1964-08
[24]
논문
The DNA Exonucleases of Escherichia coli
https://pubmed.ncbi.[...]
2011-12
[25]
논문
Exonuclease III of Escherichia coli K-12, an AP endonuclease
https://archive.org/[...]
[26]
서적
Molecular biology of nucleases
CRC Press
[27]
논문
RecBCD enzyme and the repair of double-stranded DNA breaks
https://pubmed.ncbi.[...]
2008-12
[28]
논문
Studies of the 5′ Exonuclease and Endonuclease Activities of CPSF-73 in Histone Pre-mRNA Processing
2009-01
[29]
논문
CCR4, a 3′–5′ poly(A) RNA and ssDNA exonuclease, is the catalytic component of the cytoplasmic deadenylase
2002-03
[30]
논문
Identification of a mouse protein whose homolog in Saccharomyces cerevisiae is a component of the CCR4 transcriptional regulatory complex
1995-07
[31]
논문
The 3' end of yeast 5.8S rRNA is generated by an exonuclease processing mechanism
https://pubmed.ncbi.[...]
1996-02-15
[32]
논문
The 5' end of yeast 5.8S rRNA is generated by exonucleases from an upstream cleavage site
1994-05
[33]
논문
The final step in the formation of 25S rRNA in Saccharomyces cerevisiae is performed by 5'-->3' exonucleases
http://www.rnajourna[...]
2000-12
[34]
논문
The coronavirus proofreading exoribonuclease mediates extensive viral recombination
[35]
논문
An mRNA Surveillance Mechanism That Eliminates Transcripts Lacking Termination Codons
[36]
저널
Analysis of RNA Exonucleolytic Activities in Cellular Extracts
[37]
저널
Efficient termination of transcription by RNA polymerase I requires the 5′ exonuclease Rat1 in yeast
[38]
서적
The Enzymes
Academic Press
[39]
저널
The deoxyribonucleases of ''Escherichia Coli.'' V. on the specificity of exonuclease I (Phosphodiesterase)
http://www.jbc.org/c[...]
1964-08
[40]
저널
Exonuclease III of Escherichia coli K-12, an AP endonuclease
[41]
서적
Molecular biology of nucleases
https://archive.org/[...]
CRC Press
[42]
서적
DNA Repair Protocols
Humana Press
[43]
저널
Studies of the 5′ Exonuclease and Endonuclease Activities of CPSF-73 in Histone Pre-mRNA Processing
2009-01
[44]
저널
Human 5' → 3' exonuclease Xrn2 promotes transcription termination at co-transcriptional cleavage sites
https://seoissues.wo[...]
2004-11
[45]
저널
CCR4, a 3′–5′ poly(A) RNA and ssDNA exonuclease, is the catalytic component of the cytoplasmic deadenylase
2002-03
[46]
저널
Identification of a mouse protein whose homolog in Saccharomyces cerevisiae is a component of the CCR4 transcriptional regulatory complex
1995-07
[47]
저널
The proofreading domain of Escherichia coli DNA polymerase I and other DNA and/or RNA exonuclease domains
http://nar.oxfordjou[...]
1997-12
[48]
저널
The 5' end of yeast 5.8S rRNA is generated by exonucleases from an upstream cleavage site
1994-05
[49]
저널
The final step in the formation of 25S rRNA in Saccharomyces cerevisiae is performed by 5'-->3' exonucleases
http://www.rnajourna[...]
2000-12
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com
