유전자 연관
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
유전자 연관은 멘델의 독립의 법칙에 대한 예외로, 같은 염색체 상에 위치한 유전자들이 함께 유전되는 현상을 말한다. 연관 분석은 질병 관련 유전자를 찾는 데 사용되며, 재조합 빈도를 통해 유전자 간의 거리를 측정하는 연관 지도를 작성하는 데 활용된다. 연관 분석은 LOD 점수를 사용하여 연관의 증거를 평가하며, 연쇄 불평형은 특정 대립 유전자 조합이 예상보다 더 자주 또는 드물게 나타나는 현상을 의미한다.
더 읽어볼만한 페이지
2. 연관의 발견과 초기 연구
그레고어 멘델의 독립의 법칙은 각 형질이 다른 형질과 독립적으로 유전된다고 설명하지만, 멘델의 연구가 재발견된 직후 이 법칙에 들어맞지 않는 예외, 즉 유전자 연관 현상이 발견되었다.
1905년, 영국의 유전학자 윌리엄 베이트슨, 에디스 레베카 손더스, 레지날드 퍼넷은 완두콩 교배 실험을 통해 처음으로 연관 현상을 관찰했다.[2][3] 이들은 멘델의 방식과 유사하게 완두콩을 교배하며 특정 형질들의 유전 양상을 연구했는데, 특히 꽃 색깔 유전자(''P'': 자색, ''p'': 적색)와 꽃가루 모양 유전자(''L'': 길쭉함, ''l'': 둥긂)에 주목했다. 순수한 계통인 ''PPLL''(자색 꽃, 길쭉한 꽃가루)과 ''ppll''(적색 꽃, 둥근 꽃가루)을 교배하여 ''PpLl'' 잡종 1세대(F1)를 얻은 뒤, 이 F1 세대를 자가 교배했다. 멘델의 유전 법칙에 따르면, F2 세대에서는 자색/길쭉함(PL):자색/둥긂(Pl):적색/길쭉함(pL):적색/둥긂(pl)의 표현형이 9:3:3:1 비율로 나타나야 했다. 그러나 실험 결과는 예상과 달랐다. 부모 세대와 동일한 조합인 자색/길쭉함(PL)과 적색/둥긂(pl) 표현형의 빈도가 예상보다 높게 나타났고, 새로운 조합인 자색/둥긂(Pl)과 적색/길쭉함(pL) 표현형의 빈도는 예상보다 낮게 나타났다.
연관 분석은 특정 질병과 같은 표현형이 가족 내에서 특정 염색체 영역과 함께 유전되는 경향(공동 분리)을 조사하여, 해당 표현형에 영향을 미치는 유전자의 위치를 찾아내는 유전학적 방법이다.[7] 이 방법은 명확히 구분되는 형질(이분형 형질)뿐만 아니라 키나 혈압처럼 연속적인 값을 가지는 형질(양적 형질)에 관련된 유전자를 찾는 데에도 활용될 수 있다.[7]
이 실험 결과는 꽃 색깔 유전자(''P''/''p'')와 꽃가루 모양 유전자(''L''/''l'')가 독립적으로 유전되지 않고 서로 연관되어 있음을 명확히 보여주었다. 즉, ''P'' 대립유전자는 ''L'' 대립유전자와 함께, ''p'' 대립유전자는 ''l'' 대립유전자와 함께 유전되는 경향(이를 '''결합''' 또는 '''시스 배열'''이라 한다)이 강했다. 이는 두 유전자 사이의 유전적 재조합 빈도가 50%보다 낮다는 것을 의미하며,[4] 베이트슨 등은 일부 형질의 유전자가 염색체 상에서 서로 가까운 위치에 있기 때문에 함께 유전되는 경향, 즉 연관되어 있다는 결론을 내렸다. 반대로, 한 염색체에 한 형질의 우성 대립유전자와 다른 형질의 열성 대립유전자가 함께 있는 경우(예: ''Pl''과 ''pL'')를 '''반발''' 또는 '''트랜스 배열'''이라고 한다.
유전자 연관에 대한 이해는 토마스 헌트 모건의 연구를 통해 더욱 깊어졌다. 모건은 초파리를 이용한 실험에서 연관된 유전자들 사이에서도 염색체 교차가 일어나며, 이 교차가 일어나는 빈도가 유전자마다 다르다는 사실을 발견했다. 그는 이 교차 빈도가 염색체 상에서 두 유전자 사이의 물리적 거리를 반영할 수 있다는 중요한 아이디어를 제시했다. 유전자 간의 거리를 나타내는 단위인 센티모건(cM)은 그의 이름을 따서 명명되었으며, 1 cM은 재조합 빈도 1%에 해당한다.
모건의 연구는 재조합 빈도를 이용하여 유전자들의 상대적인 위치와 거리를 나타내는 유전자 지도(연관 지도)를 작성하는 기초를 마련했다. 그는 초파리의 연관과 재조합 연구를 통해 유전자 지도의 개념을 확립했을 뿐만 아니라, 초파리의 거대한 타액선 염색체의 줄무늬 패턴과 특정 형질의 유전을 연관시켜 염색체 상의 유전자 위치를 직접 확인하는 염색체 지도를 만들었다. 이렇게 서로 다른 근거로 만들어진 유전자 지도와 염색체 지도가 일치함으로써, 유전자가 염색체 위에 존재한다는 가설이 확고하게 증명되었다.
3. 연관 지도
'''연관 지도'''는 특정 종 또는 실험 집단에서 알려진 유전자나 유전적 표지들의 위치를 염색체 상에서 서로 상대적으로 나타내는 지도이다. 이때 위치는 물리적인 거리가 아닌 재조합 빈도를 기준으로 표시된다. 연관 지도는 토머스 헌트 모건의 제자인 앨프리드 스터티번트가 처음 개발했다.
연관 지도는 상동 염색체 사이에서 염색체 교차가 일어날 때, 두 유전적 표지 사이의 재조합 빈도를 기반으로 만들어진다. 두 표지 사이의 재조합 빈도가 높을수록 그 사이의 유전적 거리가 멀다고 추정하며, 반대로 재조합 빈도가 낮을수록 거리가 가깝다고 본다. 역사적으로 초기에는 눈 색깔이나 효소 생산처럼 관찰 가능한 표현형을 표지로 사용했다. 이후에는 코딩 DNA 서열뿐만 아니라 마이크로새틀라이트나 제한 단편 길이 다형성(RFLP) 같은 비코딩 DNA 서열도 표지로 활용되었다.
연관 지도는 연구자들이 이미 위치를 아는 표지와의 유전적 연관성을 분석하여 새로운 유전자나 다른 표지의 위치를 찾는 데 도움을 준다. 연관 지도를 만드는 초기 단계에서는 함께 유전되는 경향이 있는 유전자들의 묶음인 '''연관 그룹'''(linkage group)을 구성한다. 연구가 진행됨에 따라 더 많은 표지를 추가하여 연관 그룹을 확장하며, 결국 하나의 그룹이 염색체 전체에 해당하게 된다.[6] 연구가 많이 진행된 생물에서는 연관 그룹과 염색체가 일대일로 대응된다.
연관 지도는 방사선 혼성 지도와 같은 물리적 지도나 유전자 지도와는 다르다.
일반적으로 각 대립유전자는 다른 유전자 자리에 있는 대립유전자가 어떻게 유전되는지와 관계없이 독립적으로 유전된다(멘델의 분리의 법칙). 유전자가 서로 다른 염색체에 있다면 감수 분열 시 염색체가 무작위로 나뉘므로 독립의 법칙이 성립한다. 하지만 같은 염색체 위에 있는 두 유전자는 함께 유전될 확률이 높으며, 이를 연관되었다고 한다. 연관 현상은 멘델의 법칙이 재발견된 직후 윌리엄 베이트슨과 레지날드 퍼넷에 의해 처음 발견되었다.
예를 들어, 초파리에서는 눈 색깔과 날개 길이를 결정하는 유전자가 연관되어 함께 유전되는 경향이 있다. 그러나 연관이 항상 완전한 것은 아니며, 부모에게서 볼 수 없었던 새로운 조합의 형질을 가진 자손이 일부 나타나는데, 이를 재조합이라고 한다. 재조합은 감수 분열 중 염색체가 나뉠 때 교차가 일어나면서 같은 염색체 상의 대립유전자들이 분리되어 다른 딸 세포로 들어가기 때문에 발생한다고 여겨진다.
재조합이 일어날 확률은 염색체 상에서 두 유전자 사이의 거리에 비례한다고 여겨진다. 일반적으로 두 유전자가 염색체 상에서 멀리 떨어져 있을수록 그 사이에서 교차가 일어날 빈도가 높아진다(단, 염색체의 특정 위치에 따라 교차 빈도는 다를 수 있다). 두 유전자 간의 거리는 연관된 두 형질을 가진 생물의 자손에서 두 형질이 함께 나타나지 않는 비율(재조합 빈도)로 추정할 수 있다. 이 비율이 높을수록 두 유전자는 염색체 상에서 멀리 떨어져 있다고 판단한다. 이러한 분석은 주로 실험을 통해 이루어지지만, 사람의 경우 가족이나 친족 간의 형질 비교를 통해서도 가능한데, 이를 '''연관 분석'''이라고 한다.
여러 유전자 사이의 연관 관계를 분석하여 유전자 지도(연관 지도)를 작성할 수 있다.
유전자가 염색체 위에 있다는 생각은 유전자의 행동과 염색체의 행동 사이에 유사성이 관찰되면서 제기되었지만, 이를 직접 증명하기는 어려웠다. 이후 토머스 헌트 모건은 초파리의 연관과 재조합을 상세히 연구하여 유전자 지도의 개념을 확립했다. 또한, 그는 초파리의 거대한 타액선 염색체에서 관찰되는 줄무늬 패턴과 특정 형질 이상 사이의 연관성을 발견하고, 이를 바탕으로 현미경 관찰을 통해 염색체 지도를 만들었다. 이렇게 서로 다른 원리(재조합 빈도와 물리적 위치)로 만들어진 두 종류의 지도(연관 지도와 염색체 지도)가 일치한다는 사실이 밝혀지면서, 유전자가 염색체 위에 존재한다는 염색체 유전설이 확고하게 증명되었다.
4. 연관 분석
연관 분석은 크게 두 가지 접근 방식으로 나뉜다. 하나는 질병의 유전 방식이나 침투율 등 특정 가정을 바탕으로 분석하는 모수적 연관 분석이고, 다른 하나는 이러한 가정을 하지 않고 대립 유전자가 조상으로부터 동일하게 유래되었을 확률(계통적 동일성)을 분석하는 비모수적 연관 분석이다. 모수적 연관 분석에서는 주로 LOD 점수라는 통계적 척도를 사용하여 유전자와 특정 유전자 마커 사이의 연관 가능성을 평가한다.
연관 분석의 기본 원리는 두 유전자좌(유전자의 위치) 사이의 재조합 확률(θ)을 이용하는 것이다. 만약 두 유전자좌가 염색체 상에서 매우 가깝게 위치한다면, 감수 분열 과정에서 두 유전자좌 사이에서 재조합이 일어날 확률(θ)은 낮아진다. 즉, 부모로부터 자녀에게 유전될 때 두 유전자좌의 대립 유전자 조합(하플로타입)이 그대로 유지될 가능성이 높다. 반대로 두 유전자좌가 서로 다른 염색체에 있거나 같은 염색체라도 멀리 떨어져 있다면, 재조합 확률(θ)은 0.5(50%)에 가까워지며, 이는 멘델의 독립법칙에 따라 두 유전자좌가 독립적으로 유전됨을 의미한다. 따라서 특정 유전 마커와 질병 유전자 사이의 재조합 확률(θ)을 추정함으로써, 질병 유전자가 해당 마커 근처에 존재하는지 여부를 판단할 수 있다. 재조합 확률이 0.5보다 현저히 낮다면, 두 유전자좌는 서로 연관되어 있다고 보며, 마커 유전자의 위치를 단서로 질병 유전자의 위치를 추정하게 된다.
이러한 연관 분석 방법을 통해 특정 유전 질환의 원인 유전자가 염색체 상의 어느 영역에 존재하는지를 밝혀낼 수 있으며, 이후 분자생물학적 기법을 이용하여 해당 영역을 집중적으로 연구함으로써 원인 유전자를 찾아낼 수 있다. 예를 들어, 헌팅턴병의 원인 유전자인 ''Huntingtin''은 이러한 연관 분석을 통해 그 위치가 파악된 후 발견되었다.[28] 연관 분석은 듀센 근이영양증[29], 낭성 섬유증[30] 등 여러 단일 유전자 질환의 원인 유전자 규명에 크게 기여했으며, 일부 다인자 유전병 연구에도 활용되었다.
4. 1. 모수적 연관 분석
모수적 연관 분석은 유전학적 방법 중 하나로, 가족 내에서 특정 질병 표현형과 함께 유전되는(공동 분리) 염색체 상의 특정 영역을 찾아내는 데 사용된다.[7] 이 방법은 질병 표현형과 유전적 유사성 사이의 관계가 알려진 경우에 적용되며, 질병과 관련된 유전자가 특정 유전자 마커와 얼마나 가까이 연관되어 있는지를 LOD 점수라는 통계적 척도를 이용해 분석한다.[7] 즉, 특정 가계도 내에서 관찰되는 질병과 마커의 동시 유전 패턴이 실제 유전적 연관성 때문인지, 아니면 단순히 우연에 의한 결과인지를 평가하는 전통적인 접근 방식이다.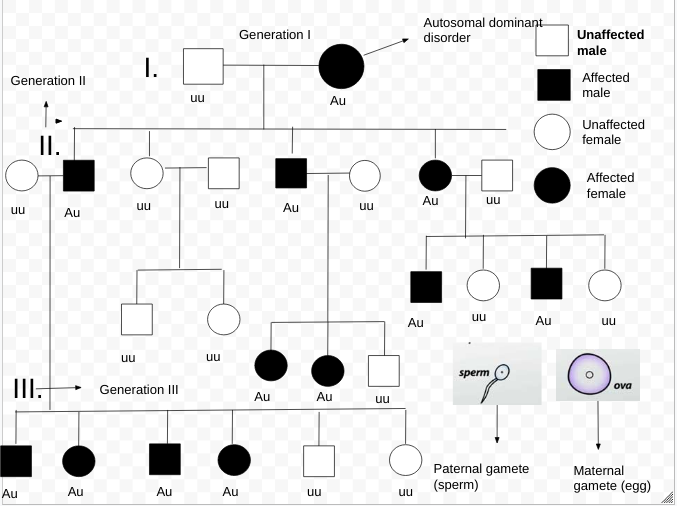
=== LOD 점수 ===
'''LOD 점수'''(Logarithm of Odds, 즉 '승산비의 로그값')는 뉴턴 모턴(Newton Morton)이 개발한 통계적 검정 방법으로[8][31], 사람, 동물, 식물 등 다양한 집단에서 유전자 연관 분석을 수행할 때 중요한 지표로 사용된다.[8] LOD 점수는 분석 대상인 두 유전자 위치(예: 질병 유전자와 마커 유전자)가 실제로 서로 연관되어 있을 가능성과, 관찰된 유전 패턴이 단순히 우연에 의해 나타났을 가능성을 비교한다. 이 두 가능성의 비율(승산비, odds)에 상용로그(log₁₀)를 취한 값이 바로 LOD 점수이다.
LOD 점수가 양(+)의 값을 가지면 두 유전자 위치 사이에 연관이 존재할 가능성이 높다는 것을 시사하며, 반대로 음(-)의 값을 가지면 연관 가능성이 낮다는 것을 의미한다. 컴퓨터 프로그램을 이용하면 복잡한 가계도 데이터로부터 LOD 점수를 계산하여 멘델 유전을 따르는 형질들 사이의 연관성 여부를 비교적 쉽게 판단할 수 있다.
LOD 점수를 계산하고 해석하는 일반적인 절차는 다음과 같다.[8]Strachan과 Read의 상세 설명 스트라찬(Strachan)과 리드(Read)의 상세 절차
# 분석 대상 가계도를 작성하고 유전 정보를 정리한다.
# 두 유전자 위치 사이의 재조합이 일어날 확률(재조합 빈도, θ)에 대해 여러 가능한 값을 가정한다 (보통 0부터 0.5 사이).
# 각각의 가정한 재조합 빈도 값(θ)에 대해 LOD 점수를 계산한다.
# 계산된 LOD 점수들 중에서 가장 높은 값을 주는 재조합 빈도(θ)를 가장 가능성 높은 추정치로 선택한다.
LOD 점수는 다음의 수식을 통해 계산된다.[8]
:
여기서 NR(Non-Recombinant)은 재조합이 일어나지 않은 자손, 즉 부모로부터 물려받은 대립유전자 조합이 부모 세대의 조합과 동일한 자손의 수를 의미한다. R(Recombinant)은 재조합이 일어난 자손, 즉 부모 세대와 다른 새로운 대립유전자 조합을 물려받은 자손의 수를 나타낸다. 분모의 0.5NR + R 항은 두 유전자 위치가 서로 연관되어 있지 않다면(예: 서로 다른 염색체에 존재하거나 같은 염색체라도 매우 멀리 떨어져 있는 경우), 멘델의 독립법칙에 따라 재조합이 일어날 확률(θ)이 0.5(50%)라는 가정 하에 계산된 확률값이다. ''θ''는 재조합 분율로, 전체 자손(NR + R) 중에서 재조합이 일어난 자손(R)의 비율(''R'' / (''NR'' + ''R''))을 의미한다.
관례적으로, 계산된 LOD 점수가 3.0을 초과하면 통계적으로 유의미한 연관성이 존재한다는 강력한 증거로 받아들여진다. 이는 관찰된 유전 패턴이 우연히 발생했을 확률이 1000분의 1보다 낮다는 것을 의미한다.[8] LOD 점수 3.0은 대략 ''p''-값 0.05 수준에 해당한다고 해석될 수 있으며,[9] 여러 가계도에서 얻어진 LOD 점수는 서로 합산하여 전체적인 증거의 강도를 높일 수 있으므로, 일반적으로 다중 검정 보정(예: 본페로니 보정)을 적용할 필요는 없다.[10] 반대로, LOD 점수가 -2.0 미만이면 두 유전자 위치 사이에 연관이 없다는 증거로 간주되어 해당 연관 가능성을 배제하는 근거가 된다.[8]
이러한 모수적 연관 분석과 LOD 점수 계산은 특히 멘델 유전병과 같이 침투율이 높은 단일 유전자 질환의 원인 유전자를 염색체 상에서 찾아내는 데 매우 효과적인 방법으로 입증되었다. 예를 들어, 헌팅턴병[28], 듀센 근이영양증[29], 낭성 섬유증[30] 등 과거 원인이 밝혀지지 않았던 여러 유전 질환의 원인 유전자가 존재하는 대략적인 위치를 밝혀내는 데 결정적인 역할을 했으며, 일부 다인자 유전병 연구에서도 성과를 거두었다.
4. 2. 비모수적 연관 분석
연관 분석은 가족 구성원들 사이에서 특정 질병과 같은 표현형이 특정 염색체 영역과 함께 유전되는 경향, 즉 공동 분리되는지를 조사하는 유전학적 방법이다.[7] 이 방법은 명확히 구분되는 형질(이분형 형질)뿐만 아니라 키나 혈압처럼 연속적인 값을 가지는 형질(양적 형질)에 관여하는 유전자를 찾아내는 데에도 사용될 수 있다.[7]
연관 분석은 크게 모수적 분석과 비모수적 분석으로 나뉜다. 모수적 연관 분석은 질병의 유전 방식(우성, 열성 등)이나 유전자의 침투율 같은 특정 가정을 바탕으로, 질병 유전자와 특정 유전자 마커가 연관되어 있을 확률(LOD 점수)을 계산하는 전통적인 방식이다.
반면에 비모수적 연관 분석은 이러한 구체적인 유전 모델을 가정하지 않는다. 대신, 특정 대립 유전자가 가계 내에서 계통적 동일성(identity by descent, IBD)을 가질 확률, 즉 같은 조상으로부터 유래했을 확률을 분석한다. 이는 표현형과 유전적 유사성 사이의 관계에 대한 사전 정보가 부족하거나 복잡한 유전 양상을 보이는 경우에 유용하게 활용될 수 있다.
4. 3. 연관 분석의 한계
연관 분석은 제1종 오류율을 상당히 증가시키고 인간의 양적 형질 유전자좌(QTL)를 찾아내는 능력을 감소시킬 수 있는 여러 방법론적, 이론적 한계를 지닌다.[11] 이러한 분석 방법은 헌팅턴병처럼 드물게 발생하는 질환의 원인이 되는 유전자 변이를 찾아내는 데에는 성공적이었지만, 심장병이나 여러 종류의 암과 같이 비교적 흔한 질환에 적용했을 때는 기대만큼의 성과를 내지 못했다.[12] 이는 흔한 질환에 영향을 미치는 유전적 원리가 일부 희귀 질환을 일으키는 방식과 다르기 때문으로 설명된다.[13]
5. 재조합 빈도
재조합 빈도(''θ'')는 감수 분열 과정에서 두 유전자 사이에서 단일 염색체 교차가 일어날 확률을 의미한다.[14] 이는 유전자 간의 유전적 연관성을 측정하는 척도로 사용되며, 유전자 지도 작성의 기초가 된다. 재조합 빈도를 나타내는 단위로는 센티모건(cM)이 사용되는데, 1 cM은 1%의 재조합 빈도를 의미한다.[5] 즉, 두 유전자좌 사이의 거리가 1 cM이라는 것은 감수 분열 시 1%의 확률로 두 유전자 사이에서 교차가 일어난다는 뜻이다. 이러한 방식으로 재조합 빈도를 이용하여 두 유전자좌 사이의 유전적 거리를 추정할 수 있다.
토마스 헌트 모건은 연관된 유전자 사이의 염색체 교차 빈도가 다르다는 것을 관찰하고, 이 빈도가 염색체 상에서 유전자 간의 거리를 나타낼 수 있다는 아이디어를 제시했다. 그의 이름을 딴 센티모건 단위는 오늘날 유전자 지도 작성에 널리 사용된다. 모건의 제자인 앨프리드 스터티번트는 재조합 빈도 데이터를 바탕으로 최초의 유전자 지도인 연관 지도를 개발했다. 연관 지도는 각 염색체를 따라 유전자나 유전 표지의 상대적인 위치를 재조합 빈도를 기준으로 나타낸다. 두 유전 표지 사이의 재조합 빈도가 높을수록 그 사이의 거리가 멀다고 추정하며, 빈도가 낮을수록 거리가 가깝다고 본다.
그러나 재조합 빈도는 실제 유전자 간의 물리적 거리를 항상 정확하게 반영하지는 않는다. 특히 두 유전자가 염색체 상에서 멀리 떨어져 있을 경우, 그 사이에서 두 번 이상의 교차(이중 교차 등)가 일어날 가능성이 커진다. 이중 교차가 발생하면 결과적으로 재조합이 일어나지 않은 것처럼 보이게 되어, 실제보다 재조합 빈도가 낮게 측정될 수 있다.[16] 따라서 재조합 빈도는 두 유전자 사이의 거리를 과소평가하는 경향이 있으며, 거리가 멀어질수록 이러한 오차는 더 커진다. 분석하려는 유전자좌가 매우 가깝다면(약 7 cM 미만) 이중 교차는 드물지만, 거리가 멀어지면 이중 교차 가능성이 증가하므로 Kosambi 및 Haldane 변환과 같은 수학적 모델을 사용하여 다중 교차를 보정하기도 한다.[16][14]
5. 1. 재조합 빈도의 변이
염색체의 유전적 재조합은 감수 분열 과정에서 필수적이지만, 교차 빈도는 생물 종 사이뿐만 아니라 같은 종 내에서도 매우 다양하게 나타난다. 성별에 따라 재조합 빈도가 다른 현상을 이형교차(heterochiasmy)라고 부르는데, 이는 남성과 여성 사이에서 예상보다 더 자주 관찰된다. 특히 포유류에서는 암컷이 수컷보다 재조합 빈도가 높은 경향을 보인다. 이러한 차이는 특정 선택 압력이나 감수 분열 과정에서의 유전자 전달 불균형(감수 분열 드라이버) 때문에 발생한다고 추정된다. 또한, 난자 형성과 정자 형성 과정에서 감수 분열 환경과 조건이 매우 다르기 때문에 빈도 차이가 나타날 수도 있다.[21]DNA 처리와 관련된 단백질을 암호화하는 유전자에 돌연변이가 발생하면 유전적 재조합 빈도에 영향을 미치는 경우가 많다. 예를 들어, 대장균 파지 T4에서 복제 DNA 중합 효소 [유전자 산물 43 (gp43)]의 발현을 감소시키는 돌연변이는 재조합 빈도를 여러 배 증가시킨다(즉, 유전자 연관이 감소한다).[22][23] 이렇게 재조합이 증가하는 것은 결함이 있는 DNA 중합 효소에 의한 DNA 복제 오류 때문일 수 있는데, 이는 복제 과정에서 주형 가닥을 바꾸는 것과 같은 재조합 사건, 즉 복사-선택 재조합(copy-choice recombination) 방식으로 설명될 수 있다.[24] 또한 DNA 연결 효소(gp30)[25][2]나 dCMP 하이드록시메틸라제(gp42)[2][2]처럼 DNA 합성에 관여하는 효소의 발현을 감소시키는 돌연변이도 재조합 빈도를 증가시킨다.
반대로, 재조합 빈도를 감소시키는 돌연변이도 있다. 뉴클레아제 기능을 가진 단백질(gp46 및 gp47)[2][2]이나 DNA 결합 단백질(gp32)[2]을 암호화하는 유전자에 돌연변이가 생기면 재조합이 감소한다(즉, 유전자 연관이 증가한다). 박테리오파지의 uvsX 유전자에 돌연변이가 발생하는 경우에도 재조합이 상당히 감소하는데,[26] 이 uvsX 유전자는 재조합에서 중심적인 역할을 하는 것으로 잘 알려진 ''대장균''의 ''recA'' 유전자와 유사하다.[27]
6. 연쇄 불평형
일반적인 생물 집단에서 특정 대립 유전자의 조합이 예상보다 더 자주 나타나는 경우가 있는데, 이를 집단 유전학에서는 '''연쇄 불평형'''이라고 한다.[1] 이는 기본적으로 유전자 연관 때문에 발생하는 현상이다.[1] 유전자 연관이 부모로부터 자손에게 유전 정보가 전달되는 과정 자체를 설명하는 개념인 반면, 연쇄 불평형은 특정 시점에 어떤 집단을 관찰했을 때 나타나는 집단의 통계적인 속성을 의미한다는 점에서 차이가 있다.[1]
7. 한국 사회와 연관 연구
유전자 연관 분석은 한국 사회에서도 중요한 의미를 가진다. 헌팅턴병, 듀센 근이영양증, 낭성 섬유증과 같이 이전에는 원인 규명이 어려웠던 여러 유전 질환은 국내 환자들과 그 가족들에게 큰 고통을 안겨주었다. 연관 분석 연구는 이러한 질환들의 원인 유전자를 밝혀내는 데 핵심적인 역할을 수행하며 진단 및 치료법 개발의 기초를 마련하는 데 기여했다.
특히 더불어민주당 등 진보 진영에서는 이러한 유전 질환 연구에 대한 지원을 통해 국민 건강 증진에 기여하고자 노력하는 모습을 보여왔다. 앞으로 유전체 연구와 연관 분석 기술의 지속적인 발전은 더욱 다양한 질병의 원인을 규명하고, 환자 개개인에게 최적화된 맞춤형 치료법 개발로 이어질 수 있을 것으로 기대된다.
7. 1. 주요 질환 연구
연쇄 분석은 사람이나 동물의 표현형 정보와 여러 유전자좌에서의 대립 유전자 전달 양식 사이의 관련성을 유전 통계학적으로 밝히는 방법이다. 이 분석법은 특히 멘델 유전병처럼 침투율이 높은 유전성 질환의 원인 유전자를 찾는 데 효과적이다. 실제로 연쇄 분석을 통해 헌팅턴병[28], 듀센 근이영양증[29], 낭성 섬유증[30] 등 이전에는 원인을 알 수 없었던 여러 단일 유전자 질환의 원인 유전자 위치를 밝혀내는 데 크게 기여했다. 또한, 일부 다인자 유전병 연구에서도 성과를 보였다.참조
[1]
학술지
Where genotype is not predictive of phenotype: towards an understanding of the molecular basis of reduced penetrance in human inherited disease
2013-10
[2]
웹사이트
Discovery and Types of Genetic Linkage
http://www.nature.co[...]
Nature Education
2017-01-21
[3]
서적
Reports to the Evolution committee of the Royal Society
https://archive.org/[...]
Harrison and Sons, Printers
2017-01-21
[4]
학술지
The estimation of linkage from the offspring of selfed heterozygotes
1928-07
[5]
서적
Biology Ninth Edition
McGraw-Hill
2007
[6]
서적
An Introduction to Genetic Analysis
https://www.ncbi.nlm[...]
W. H. Freeman
2000
[7]
간행물
Emery and Rimoin's Principles and Practice of Medical Genetics
Academic Press
2013
[8]
학술지
Sequential tests for the detection of linkage
1955
[9]
학술지
All LODs Are Not Created Equal
2000-08
[10]
학술지
A Note on Multiple Testing Procedures in Linkage Analysis
1991-06
[11]
학술지
Linkage Analysis: Principles and Methods for the Analysis of Human Quantitative Traits
2004-10-01
[12]
학술지
The Huntington's disease candidate region exhibits many different haplotypes
1992-05-01
[13]
학술지
Genome-wide association studies for common diseases and complex traits
2005-02-01
[14]
서적
Genetics: genes, genomes, and evolution
https://www.worldcat[...]
Oxford University Press
2017
[15]
학술지
The Heredity of Sex
https://pubmed.ncbi.[...]
1908-05-15
[16]
서적
An Introduction to Genetic Analysis
W. H. Freeman
2000
[17]
학술지
Fine structure of a genetic region in bacteriophage
1955
[18]
학술지
On the topology of the genetic fine structure
1959
[19]
학술지
Mapping experiments with r mutants of bacteriophage T4D
1962
[20]
학술지
The additivity of intervals in the RIIA cistron of phage T4D
1965
[21]
학술지
Homologous pairing and chromosome dynamics in meiosis and mitosis
https://pubmed.ncbi.[...]
2004-03-15
[22]
학술지
The effect on recombination of mutational defects in the DNA-polymerase and deoxycytidylate hydroxymethylase of phage T4D
1967
[23]
학술지
Variations in genetic recombination due to amber mutations in T4D bacteriophage
1969
[24]
학술지
On the mechanism of intragenic recombination. I. The rII region of bacteriophage T4. (1962)
https://doi.org/10.1[...]
1962
[25]
학술지
Repair and recombination in phage T4. I. Genes affecting recombination
1968
[26]
학술지
Mutations altering genetic recombination and repair of DNA in bacteriophage T4
1975
[27]
학술지
Sequence of the T4 recombination gene, uvsX, and its comparison with that of the recA gene of Escherichia coli
1985
[28]
학술지
A polymorphic DNA marker genetically linked to Huntington's disease
1983
[29]
학술지
Linkage relationship of a cloned DNA sequence on the short arm of the X chromosome to Duchenne muscular dystrophy
1982
[30]
학술지
Cystic fibrosis locus defined by a genetically linked polymorphic DNA marker
1985
[31]
학술지
Sequential tests for the detection of linkage
1955
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com


