피루브산 탈수소효소 복합체
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
피루브산 탈수소효소 복합체는 피루브산, 조효소 A(CoA), NAD+를 아세틸-CoA, CO2, NADH, H+로 전환하는 반응을 촉매하는 효소 복합체이다. 이 복합체는 여러 효소 소단위체로 구성되며, 진핵생물의 미토콘드리아 기질에 위치한다. 피루브산 탈수소효소 복합체는 E1, E2, E3의 세 가지 주요 효소와 E3 결합 단백질(E3BP)로 구성되며, 생물종에 따라 구조적 차이가 있다. 이 복합체는 ATP/ADP, NADH/NAD+, 아세틸-CoA/CoA 비율에 의해 조절되며, 피루브산 탈수소효소 키네이스(PDK)와 피루브산 탈수소효소 인산가수분해효소(PDP)에 의해 조절된다. 피루브산 탈수소효소 복합체의 결핍은 젖산산증을 유발하며, 다양한 유전자의 돌연변이와 관련이 있다. 진화적으로는 그람양성세균 효소와 유사성을 가지며, 가지사슬 α-케토산 탈수소효소 복합체와도 유사한 점이 있다.
더 읽어볼만한 페이지
- EC 1.2.1 - 수소화효소
수소화효소는 활성 부위 금속 성분에 따라 니켈-철, 철-황, 철 수소화효소 등으로 나뉘며 미생물에서 발견되는 효소로, 수소 산화 환원 반응 촉매, 에너지 생성, 생물 정화 등 생물학적 기능 수행 및 재생 에너지 분야 응용 가능성으로 주목받는다. - EC 1.2.1 - 폼산 탈수소효소
폼산 탈수소효소는 폼산을 이산화탄소로 산화시키는 효소로, NAD 의존성과 사이토크롬 의존성 효소로 나뉘며 메틸영양생물과 혐기성 대사에서 중요하고 몰리브도프테린, 몰리브데넘, 셀레늄을 필요로 하며 전자 전달을 수행한다. - 해당 - 유비퀴논
유비퀴논(CoQ10)은 미토콘드리아 전자 전달계의 주요 구성 요소인 지용성 유기 화합물로, 세포 호흡과 에너지 생산에 필수적이며 항산화 작용을 통해 세포를 보호하고 면역 반응에도 관여한다. - 해당 - 글루코스
글루코스는 6개의 탄소 원자를 가진 단당류로, D-글루코스 이성질체는 덱스트로스라고도 불리며 생물체의 에너지원, 식물의 광합성 생산물, 그리고 다양한 탄수화물의 구성 성분으로서 중요한 역할을 한다. - 세포 호흡 - 유비퀴논
유비퀴논(CoQ10)은 미토콘드리아 전자 전달계의 주요 구성 요소인 지용성 유기 화합물로, 세포 호흡과 에너지 생산에 필수적이며 항산화 작용을 통해 세포를 보호하고 면역 반응에도 관여한다. - 세포 호흡 - 광합성
광합성은 생물이 빛에너지를 이용하여 이산화탄소와 물로부터 유기물을 합성하고 산소를 방출하는 과정으로, 엽록체 내 틸라코이드 막에서 일어나는 명반응과 스트로마에서 일어나는 암반응으로 구성되며, 환경에 따라 탄소 농축 메커니즘을 통해 효율을 높이기도 하고, 지구 대기의 산소를 생성하는 주요 원천이다.
2. 반응
피루브산 탈수소효소 복합체는 피루브산을 아세틸-CoA로 전환하는 반응을 촉매한다. 이 과정에서 CoA-SH와 NAD+가 추가적으로 필요하며, CO2, NADH, H+가 생성된다.
피루브산 탈수소효소 복합체는 여러 개의 효소 소단위체로 구성된 거대한 복합체이다. 생물종에 따라 구성에 약간의 차이가 있을 수 있다.
3. 구조
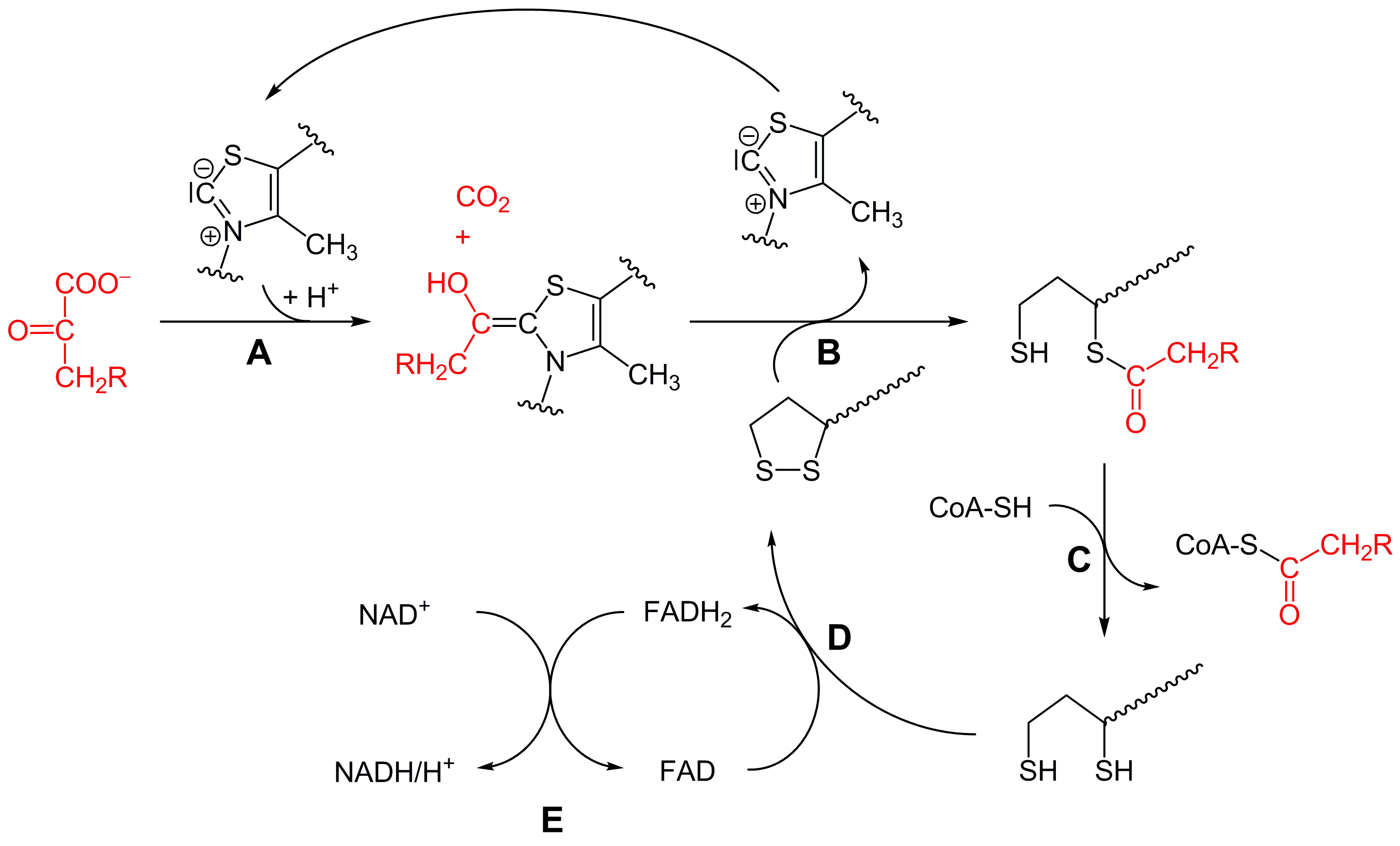
진핵생물의 미토콘드리아 기질에 위치하는 피루브산 탈수소효소 복합체는 3가지 기능성 단백질로 구성된다. 원핵생물과 진핵생물의 피루브산 탈수소효소 복합체 구성에는 차이가 있는데, 진핵생물에는 E3를 복합체에 결합시키는 추가적인 단백질인 E3 결합 단백질(E3BP)이 존재한다.
3. 1. 피루브산 탈수소효소 (E1)
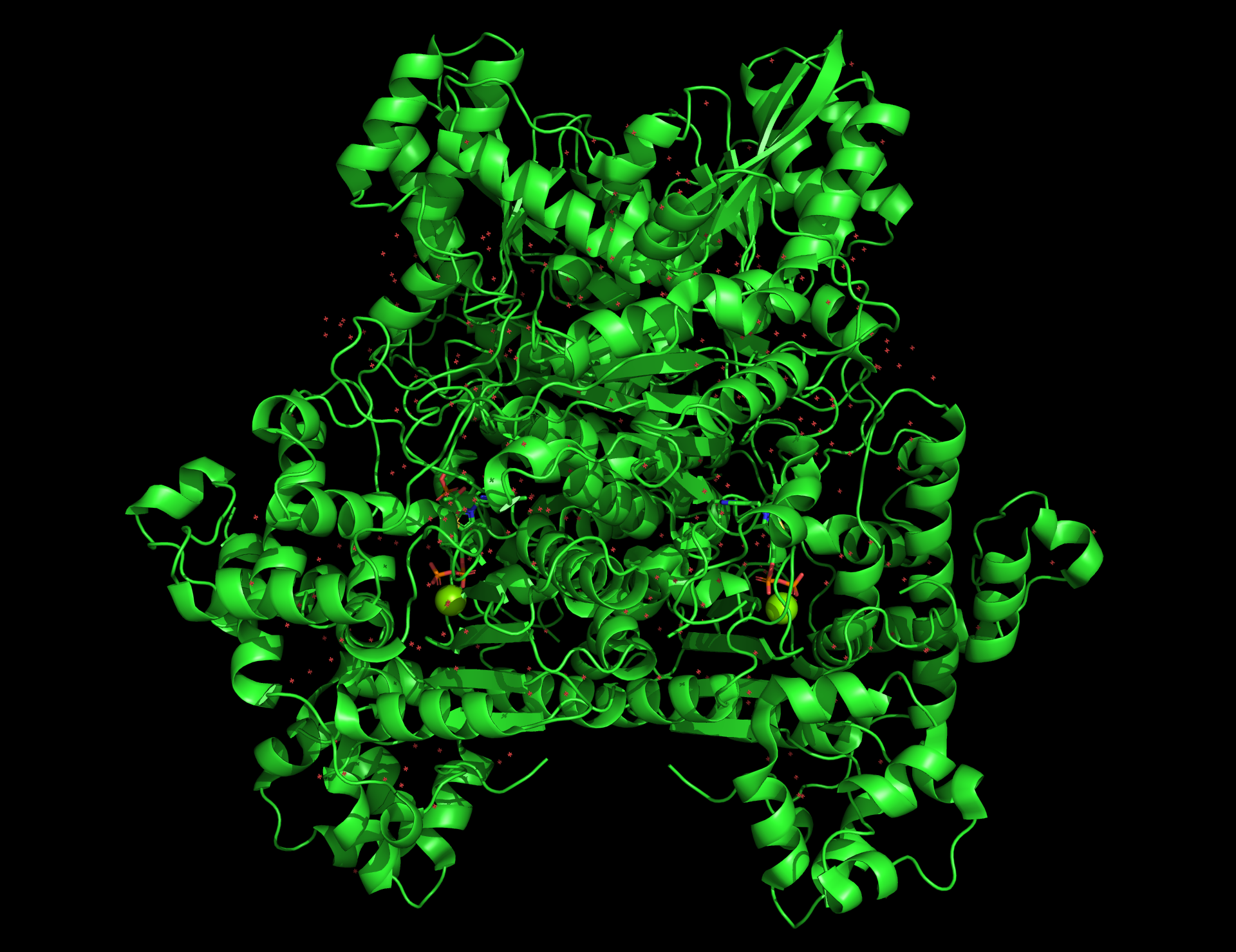
피루브산 탈수소효소(E1)는 피루브산 탈수소효소 복합체의 소단위체 중 하나로, 동종이량체(예: 대장균에서 두 개의 "α" 사슬) 또는 이종사량체(두 개의 "α" 사슬 및 두 개의 "β" 사슬) 구조를 갖는다. 마그네슘 이온은 α 사슬에 있는 세 개의 극성 아미노산 잔기(Asp, Asn, Tyr)와 티아민 피로인산(TPP)에 4배위 복합체를 형성하며, TPP는 피루브산의 탈카복실화에 직접 관여하는 보조 인자이다.[27][28]
피루브산과 티아민 피로인산(TPP, 비타민 B1)이 피루브산 탈수소효소 소단위체에 결합하면, TPP의 티아졸륨 고리가 양쪽성 이온 형태로 존재하며, 음이온성 C2 탄소가 피루브산의 C2 카보닐기(케톤)를 친핵성 공격한다. 생성된 헤미티오아세탈은 탈카복실화를 거쳐 아실 음이온 등가물을 생성한다. 이 음이온은 리신 잔기에 부착된 산화된 리포산의 S1을 공격하고, 개환 SN2 유사 메커니즘에 의해 S2는 황화물 또는 설프하이드릴 부분으로 대체된다. 이후 사면체 헤미티오아세탈이 붕괴되면서 티아졸이 방출되고 TPP 보조 인자가 떨어져 나가면, 리포산의 S1에 티오아세트산이 생성된다. E1의 촉매 반응은 전체 피루브산 탈수소효소 복합체의 속도 제한 단계이다.
3. 2. 다이하이드로리포일 아세틸기전이효소 (E2)
다이하이드로리포일 아세틸기전이효소(E2)는 피루브산 탈수소효소 복합체의 구성 요소 중 하나로, E1에서 생성된 아세틸기를 조효소 A(CoA)로 전달하여 아세틸-CoA를 생성하는 반응을 촉매한다.[29] 이 과정에서 α-리포산이 보조 인자로 작용한다.[29] E2는 리포아마이드 환원효소-아세틸기전이효소로도 알려져 있다.
E2 소단위체는 N-말단 도메인(리포일 도메인), 말초 소단위체 결합 도메인(PSBD), C-말단 도메인(촉매 도메인)의 세 가지 도메인으로 구성된다.[29] 각 도메인의 기능은 다음과 같다.
감마프로테오박테리아의 E2는 24개의 사본이 모여 입방체 코어를 형성하며, 8개의 E2 동종삼량체가 입방체 코어의 각 꼭짓점에 위치한다.[29]
E2의 활성 부위에서 리포산-싸이오에스터는 조효소 A(CoA)의 싸이올로 아세틸기를 전달하는 트랜스 아실화 반응을 촉매한다.[25] 이 반응을 통해 생성된 아세틸-CoA는 피루브산 탈수소효소 복합체에서 방출되어 시트르산 회로로 들어간다.[25]
3. 3. 다이하이드로리포일 탈수소효소 (E3)
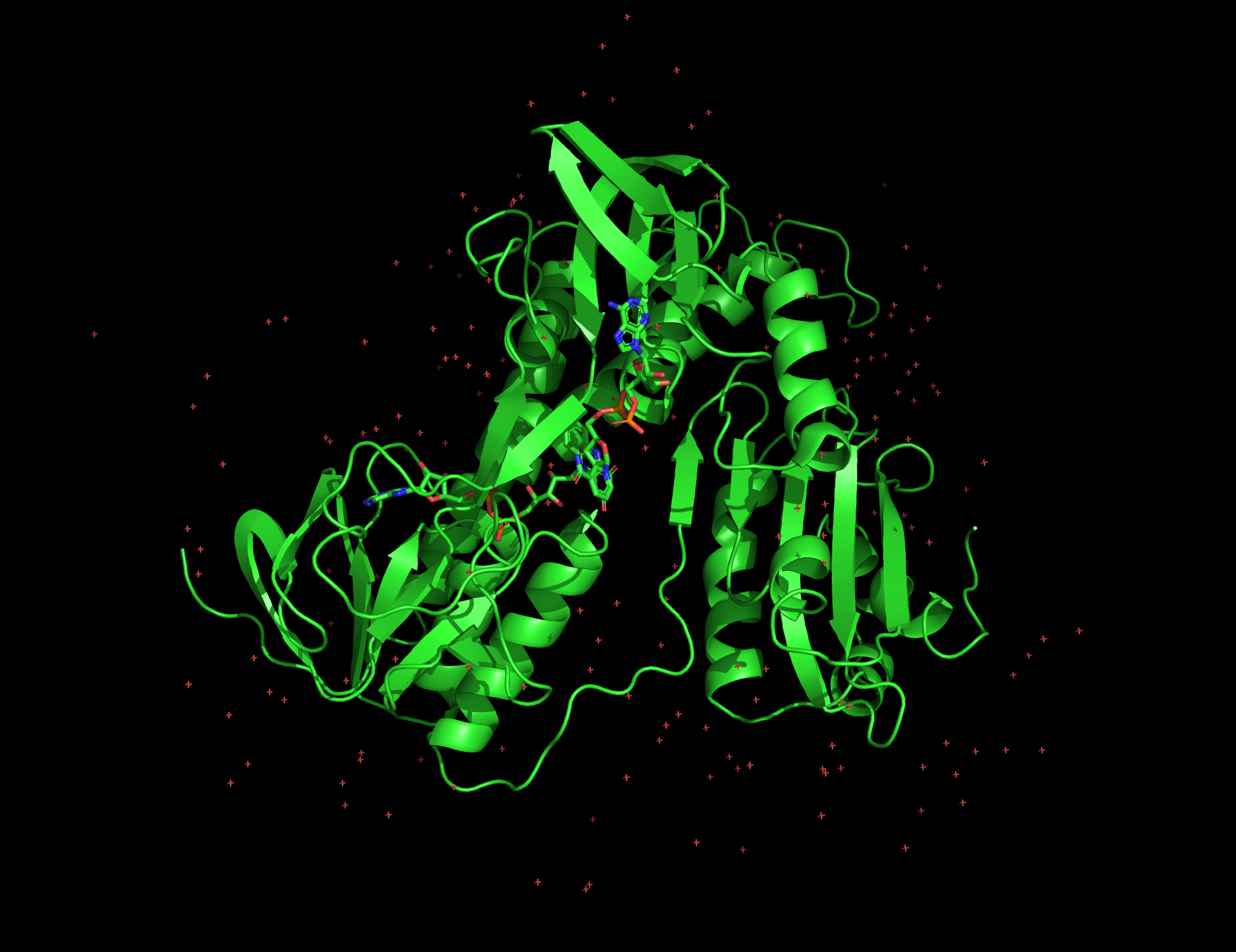
다이하이드로리포일 탈수소효소(E3)는 이황화 결합에 관여하는 2개의 시스테인 잔기와 활성 부위의 FAD 보조 인자가 산화 촉매로서의 주요 기능을 하는 동종이량체 단백질이다. 슈도모나스 푸티다(''Pseudomonas putida'')에서 발견되는 E3 구조는 각 동종이량체 소단위체가 FAD 결합과 NAD 결합을 담당하는 두 개의 결합 도메인, 중앙 도메인, 인터페이스 도메인으로 구성된다.[30][31]
피루브산 탈수소효소 복합체의 리신 잔기에 결합되어 있는 다이하이드로리포산은 다이하이드로리포일 탈수소효소(E3)의 활성 부위로 이동하여[25] 티오레독신 환원효소와 유사한 플라빈 매개 산화를 겪는다. 먼저, FAD는 다이하이드로리포산을 리포산으로 산화시켜 FADH2를 생성한다. 그런 다음, NAD+ 보조 인자는 FADH2를 FAD로 산화시켜 NADH와 H+를 생성한다.
3. 4. 다이하이드로리포일 탈수소효소 결합 단백질 (E3BP)
E3 결합 단백질(E3BP)은 대부분의 진핵생물에 있는 보조 단백질로, E3 소단위체를 피루브산 탈수소효소 복합체(PDC)에 결합시키는 역할을 한다. 사람의 E3 결합 단백질의 경우, 결합 단백질의 소수성 프롤린과 류신 잔기는 두 개의 동일한 E3 단량체가 결합하여 형성된 표면 인식 부위와 상호작용을 한다.[32]
4. 생물종 간의 구조적 차이
피루브산 탈수소효소 복합체(PDC)는 생물종에 따라 구성에 차이가 있다. 모든 생물에서 PDC는 E1, E2, E3 세 가지 촉매 서브유닛의 여러 복사본으로 구성된 큰 복합체이다. 모든 PDC의 공통적인 특징은 E2 서브유닛이 복합체의 핵심을 형성하고, 주변 서브유닛 E1과 E3가 이에 결합한다는 것이다. 진핵생물의 PDC는 E3 결합 단백질(E3BP, "단백질 X"라고도 함)이라는 추가적인 비촉매 서브유닛을 핵심에 포함한다. 이종 올리고머 코어를 가진 진핵생물 PDC에서 E1은 E2에만 결합하고, E3는 E3BP에만 결합한다. 반면에, 동종 올리고머 E2 코어를 가진 박테리아 PDC에서는 E1과 E3가 E2에 결합하기 위해 경쟁한다.
주변 효소 E3는 모든 유기체에서 이량체이지만, 주변 효소 E1은 진핵생물에서 alpha2beta2 이종사량체이다. 피루브산 탈수소효소 복합체는 미토콘드리아 기질에 위치하며, 총 60개의 서브 유닛을 포함하고 3개의 기능성 단백질로 구성된다.
4. 1. 그람음성세균
대장균(Escherichia coli)과 같은 그람음성세균의 피루브산 탈수소효소 복합체는 24개의 다이하이드로리포일 아세틸기전이효소(E2)로 구성된 중앙 입방체 코어를 가진다. E2 코어 주변에는 최대 16개의 피루브산 탈수소효소(E1) 동종이량체와 8개의 다이하이드로리포일 탈수소효소(E3) 동종이량체가 결합한다.[33] 감마프로테오박테리아에서 E1 또는 E3 결합에 대한 PSBD의 특이성은 PSBD의 올리고머 상태에 의해 결정된다. 각 E2 동종삼량체에서는 PSBD 3개 중 2개가 이량체화된다. 두 개의 E1 동종이량체는 이량체 PSBD에 협력적으로 결합하는 반면, 짝을 이루지 않은 나머지 PSBD는 하나의 E3 동종이량체와 특이적으로 상호작용한다. 따라서 PSBD 이량체화는 E1:E2:E3(단량체) = 32:24:16의 화학량론을 갖는 주변 소단위체 E1 및 E3으로 완전히 포화될 때 피루브산 탈수소효소 복합체의 소단위체 구성을 결정한다.[33]4. 2. 그람양성세균 및 진핵생물
그람 양성균(예: 바실루스 스테아로테르모필루스)과 진핵생물에서 중심 PDC 코어는 60개의 E2 분자가 정이십면체로 배열되어 있다. 이 E2 소단위체 "코어"는 30개의 E1 소단위체와 12개의 E3 복사본과 결합한다.[34]진핵생물은 또한 E3 소단위체를 E2 코어에 결합시키는 추가적인 코어 단백질인 E3 결합 단백질(E3BP) 12개의 복사본을 포함한다.[34] E3BP의 정확한 위치는 완전히 명확하지 않다. 저온 전자 현미경을 통해 E3BP가 효모의 각 정이십면체 면에 결합한다는 사실이 확인되었다.[35] 그러나 이는 소의 PDC 코어에서 동일한 수의 E2 분자를 대체하는 것으로 제안되었다.
그람 양성균의 경우 최대 60개의 E1 또는 E3 분자가 E2 코어와 결합할 수 있으며, 결합은 상호 배타적이다. 진핵생물에서는 E1이 E2에 특이적으로 결합하는 반면 E3는 E3BP와 결합한다. 최대 30개의 E1 및 6개의 E3 효소가 존재한다고 생각되지만, 분자의 정확한 수는 ''생체 내''에서 다양할 수 있으며 종종 해당 조직의 대사 요구 사항을 반영한다.
5. 메커니즘
피루브산 탈수소효소 소단위체(E1)에 의한 첫 번째 단계는 피루브산과 티아민 피로인산(TPP)의 결합이다.[25] TPP의 티아졸륨 고리는 양쪽성 이온 형태이며, 음이온성 C2 탄소가 피루브산의 C2 카보닐기(케톤)를 친핵성 공격한다. 생성된 헤미티오아세탈은 탈카복실화를 거쳐 아실 음이온 등가물을 생성한다. 이 음이온은 리신 잔기에 부착된 산화된 리포산의 S1을 공격한다. 개환 SN2 유사 메커니즘에서 S2는 황화물 또는 설프하이드릴 부분으로 대체된다. 이후 사면체 헤미티오아세탈이 붕괴되면 티아졸이 방출되고 TPP 보조 인자가 방출되며 리포산의 S1에 티오아세트산이 생성된다. E1 촉매 과정은 전체 피루브산 탈수소효소 복합체의 속도 제한 단계이다.
다음으로, 다이하이드로리포일 아세틸기전이효소(E2)는 리포산-싸이오에스터 기능기를 활성 부위로 전위시킨다.[25] 여기서 트랜스 아실화 반응은 리포일의 "스윙잉 암(swinging arm)"으로부터 조효소 A(CoA)의 싸이올로 아세틸기를 전달한다. 이는 아세틸-CoA를 생성하고, 아세틸-CoA는 피루브산 탈수소효소 복합체로부터 방출되며 이어서 시트르산 회로로 들어간다. E2는 리포아마이드 환원효소-아세틸기전이효소로도 알려져 있다.
마지막으로, 다이하이드로리포산은 다이하이드로리포일 탈수소효소(E3)의 활성 부위로 이동하여[25] 플라빈 매개 산화를 겪는다. 먼저, FAD는 다이하이드로리포산을 리포산으로 다시 산화시켜 FADH2를 생성한다. 그런 다음, NAD+ 보조 인자는 FADH2를 FAD로 다시 산화시켜 NADH와 H+를 생성한다.
6. 조절
ATP/ADP, NADH/NAD+ 및 아세틸-CoA/CoA 비율 중 하나 이상이 증가하면 피루브산 탈수소효소는 억제된다.
진핵생물에서 피루브산 탈수소효소 복합체(PDC)는 고유의 특정 피루브산 탈수소효소 키네이스(PDK)와 피루브산 탈수소효소 인산가수분해효소(PDP)에 의해 각각 엄격히 조절되어 비활성화되거나 활성화된다.[36]
- 피루브산 탈수소효소 키네이스(PDK)는 E1의 세 가지 특정 세린 잔기를 서로 다른 친화도로 인산화한다. 이들 중 어느 하나의 인산화(ATP 사용)는 E1(결과적으로 전체 복합체)을 비활성화시킨다.[36]
- 피루브산 탈수소효소 인산가수분해효소(PDP)에 의한 E1의 탈인산화는 피루브산 탈수소효소 복합체의 활성을 회복시킨다.[36]
반응 생성물은 피루브산 탈수소효소 키네이스(PDK)를 활성화시키기 때문에 피루브산 탈수소효소 복합체(PDC)의 다른 자리 입체성 저해제 역할을 한다. 기질은 차례로 피루브산 탈수소효소 키네이스(PDK)를 저해하여 피루브산 탈수소효소 복합체를 재활성화한다.
기아 상태에서 피루브산 탈수소효소 키네이스(PDK)는 유전자 전사 증가를 통해 골격근을 포함한 대부분의 조직에서 양이 증가한다. 동일한 조건에서는 피루브산 탈수소효소 인산가수분해효소(PDP)의 양이 감소한다. 피루브산 탈수소효소 복합체(PDC)의 결과적인 저해는 근육 및 다른 조직이 포도당 및 포도당신생합성 전구체를 이화대사하는 것을 방지한다. 대사는 지방 활용 쪽으로 전환되고, 포도당신생합성 전구체를 공급하기 위한 근육 단백질 분해는 최소화되며, 뇌에서 사용하기 위한 이용 가능한 포도당은 절약된다.
칼슘 이온은 근육 수축 중에 피루브산 탈수소효소 인산가수분해효소(PDP)를 활성화하여 세포질에서 일어나는 해당과정을 자극하기 때문에 근육 조직에서 피루브산 탈수소효소 복합체(PDC)를 조절하는 역할을 한다. 이러한 전사의 일부 생성물은 H2를 근육으로 방출한다. 이로 인해 시간이 지남에 따라 칼슘 이온이 감소할 수 있다.
7. 임상적 관련성
피루브산 탈수소효소 결핍증(PDCD)은 피루브산 탈수소효소 복합체를 구성하는 효소나 보조 인자의 돌연변이로 인해 발생할 수 있다. 피루브산 탈수소효소 결핍증의 주요 임상적 소견은 젖산산증이다.[42] 이러한 피루브산 탈수소효소 결핍증 돌연변이는 산화적 인산화 과정에 필요한 NAD+ 및 FAD 생성을 저해한다. 그 결과 아세틸-CoA는 혐기성 대사 과정을 통해 젖산으로 전환되어 체내에 젖산이 과도하게 축적되고 관련 신경학적 병증을 유발한다.[43]
피루브산 탈수소효소 결핍증은 드물지만, 돌연변이가 발생하거나 기능하지 않는 경우 이러한 결핍을 유발할 수 있는 다양한 유전자가 있다. 피루브산 탈수소효소의 E1 소단위체는 4개의 소단위체(E1-α로 지정된 2개의 α 소단위체와 E1-β로 지정된 2개의 β 소단위체)로 구성된다. 피루브산 탈수소효소 결핍증의 약 80%는 E1-α 소단위체에 있는 PDHA1 유전자의 돌연변이 때문에 발생한다. 이 돌연변이는 E1-α 단백질을 짧게 만들어, 기능하는 E1-α를 감소시키고 피루브산 탈수소효소가 피루브산과 충분히 결합하는 것을 방해하여 전체 복합체의 활성을 감소시킨다.[44] 피루브산 탈수소효소 복합체의 E1-β 소단위체에 있는 PDHB 유전자의 돌연변이도 피루브산 탈수소효소 결핍증을 일으킨다.[45] E2 소단위체의 DLAT 유전자, E3 소단위체의 PDHX 유전자, 피루브산 탈수소효소 인산가수분해효소 유전자인 PDP1의 돌연변이도 피루브산 탈수소효소 결핍증과 관련이 있지만, 질병에 대한 정확한 기여 정도는 알려져 있지 않다.[46][47][48]
8. 진화
진핵생물의 미토콘드리아에서 발견되는 피루브산 탈수소효소는 그람양성세균인 게오바실루스 스테아로테르모필루스(''Geobacillus stearothermophilus'')의 효소와 매우 유사하다.[39] 그람음성세균과는 유사성이 거의 없다. 피루브산 탈수소효소와 그람양성세균 효소의 4차 구조 유사성은 이들이 진화적으로 연관되어 있음을 보여준다. 세포 내 공생을 통해 진핵생물의 미토콘드리아에 있는 피루브산 탈수소효소는 그람양성세균에서 기원한 것으로 보인다.[39]
피루브산 탈수소효소 복합체는 가지사슬 α-케토산 탈수소효소 복합체와 많은 유사점을 공유하는데, 특히 α-케토산에 대한 기질 특이성이 비슷하다.[40] 가지사슬 α-케토산 탈수소효소 복합체는 아미노산 분해를 촉진하며, 원시 지구 환경에서 널리 퍼져 있었을 것으로 추정된다. 피루브산 탈수소효소 복합체의 E2 소단위체는 가지사슬 α-케토산 탈수소효소 복합체의 E2 유전자에서 진화했고, 두 효소 복합체는 동일한 E3 소단위체를 가진다. E1 소단위체는 기질 특이성이 다르지만, 피루브산 탈수소효소 복합체와 가지사슬 α-케토산 탈수소효소 복합체의 E1 소단위체는 유전적 유사성을 공유한다. 그람양성세균과 남세균은 가지사슬 α-케토산 탈수소효소 복합체와 유전적으로 관련된 E1 소단위체를 가지는데, 이들은 진핵세포의 미토콘드리아와 엽록체의 조상으로 추정된다.[40][41]
참조
[1]
간행물
Chapter 12 - Pyruvate Dehydrogenase Complex Deficiency
http://www.sciencedi[...]
Academic Press
2016-01-01
[2]
서적
Biochemistry
Freeman
[3]
논문
How phosphorylation influences E1 subunit pyruvate dehydrogenase: A computational study
2018
[4]
논문
A dynamic loop at the active center of the Escherichia coli pyruvate dehydrogenase COMPLEX E1 component Modulates SUBSTRATE utilization and CHEMICAL communication with the E2 component
2007
[5]
논문
The pyruvate dehydrogenase complexes: structure-based function and regulation
2014
[6]
논문
Lipoic Acid Biosynthesis and Enzymology
2010
[7]
논문
The refined crystal STRUCTURE of pseudomonas Putida LIPOAMIDE DEHYDROGENASE complexed with NAD+ at 2.45 Å resolution
1992
[8]
논문
How dihydrolipoamide dehydrogenase-binding protein binds dihydrolipoamide dehydrogenase in the human pyruvate dehydrogenase complex
2006
[9]
논문
Dimerization of a 5-kDa domain defines the architecture of the 5-MDa gammaproteobacterial pyruvate dehydrogenase complex
[10]
논문
Structural insight into interactions between Dihydrolipoamide Dehydrogenase (E3) and E3 binding protein of Human pyruvate dehydrogenase complex
2006
[11]
문서
On the unique structural organization of the Saccharomyces cerevisiae pyruvate dehydrogenase complex
[12]
간행물
6 - Glycolysis and Pyruvate Oxidation
http://www.sciencedi[...]
W.B. Saunders
2012-01-01
[13]
논문
The transport of pyruvate in rat liver mitochondria
1971-01-30
[14]
논문
The long and winding road to the mitochondrial pyruvate carrier
2013-01-23
[15]
논문
Structure and symmetry of B. stearothermophilus pyruvate dehydrogenase multienzyme complex and implications for eucaryote evolution
http://dx.doi.org/10[...]
1979-05
[16]
논문
E1 Enzyme of the Pyruvate Dehydrogenase Complex in Corynebacterium glutamicum: Molecular Analysis of the Gene and Phylogenetic Aspects
2005-09-01
[17]
논문
Evolution of the enzymes of the citric acid cycle and the glyoxylate cycle of higher plants
2002-02-01
[18]
웹사이트
Pyruvate dehydrogenase deficiency
http://ghr.nlm.nih.g[...]
2013-03-17
[19]
논문
Pyruvate Dehydrogenase Complex Deficiency: An Unusual Cause of Recurrent Lactic Acidosis in a Paediatric Critical Care Unit
[20]
논문
Mutations in the X-linked pyruvate dehydrogenase (E1) ? subunit gene (PDHA1) in patients with a pyruvate dehydrogenase complex deficiency
2000-03
[21]
논문
Mutations of the E1β subunit gene (PDHB) in four families with pyruvate dehydrogenase deficiency
http://dx.doi.org/10[...]
2008-04
[22]
논문
Clinical and genetic spectrum of pyruvate dehydrogenase deficiency: Dihydrolipoamide acetyltransferase (E2) deficiency
http://dx.doi.org/10[...]
2005-07-27
[23]
논문
Pyruvate dehydrogenase complex deficiency: updating the clinical, metabolic and mutational landscapes in a cohort of Portuguese patients
2020-12
[24]
논문
Mitochondrial pyruvate dehydrogenase phosphatase 1 regulates the early differentiation of cardiomyocytes from mouse embryonic stem cells
2016-08
[25]
간행물
Chapter 12 - Pyruvate Dehydrogenase Complex Deficiency
http://www.sciencedi[...]
Academic Press
2016-01-01
[26]
서적
Biochemistry
Freeman
[27]
논문
How phosphorylation influences E1 subunit pyruvate dehydrogenase: A computational study
2018
[28]
논문
A dynamic loop at the active center of the Escherichia coli pyruvate dehydrogenase COMPLEX E1 component Modulates SUBSTRATE utilization and CHEMICAL communication with the E2 component
2007
[29]
논문
The pyruvate dehydrogenase complexes: structure-based function and regulation
2014
[30]
논문
Lipoic Acid Biosynthesis and Enzymology
2010
[31]
논문
The refined crystal STRUCTURE of pseudomonas Putida LIPOAMIDE DEHYDROGENASE complexed with NAD+ at 2.45 Å resolution
1992
[32]
논문
How dihydrolipoamide dehydrogenase-binding protein binds dihydrolipoamide dehydrogenase in the human pyruvate dehydrogenase complex
2006
[33]
논문
Dimerization of a 5-kDa domain defines the architecture of the 5-MDa gammaproteobacterial pyruvate dehydrogenase complex
[34]
논문
Structural insight into interactions between Dihydrolipoamide Dehydrogenase (E3) and E3 binding protein of Human pyruvate dehydrogenase complex
2006
[35]
문서
On the unique structural organization of the Saccharomyces cerevisiae pyruvate dehydrogenase complex
[36]
서적
6 - Glycolysis and Pyruvate Oxidation
http://www.sciencedi[...]
W.B. Saunders
2012-01-01
[37]
논문
The transport of pyruvate in rat liver mitochondria
1971-01-30
[38]
논문
The long and winding road to the mitochondrial pyruvate carrier
2013-01-23
[39]
논문
Structure and symmetry of B. stearothermophilus pyruvate dehydrogenase multienzyme complex and implications for eucaryote evolution
http://dx.doi.org/10[...]
1979-05
[40]
논문
E1 Enzyme of the Pyruvate Dehydrogenase Complex in Corynebacterium glutamicum: Molecular Analysis of the Gene and Phylogenetic Aspects
2005-09-01
[41]
논문
Evolution of the enzymes of the citric acid cycle and the glyoxylate cycle of higher plants
2002-02-01
[42]
웹인용
Pyruvate dehydrogenase deficiency
http://ghr.nlm.nih.g[...]
2013-03-17
[43]
논문
Pyruvate Dehydrogenase Complex Deficiency: An Unusual Cause of Recurrent Lactic Acidosis in a Paediatric Critical Care Unit
[44]
논문
Mutations in the X-linked pyruvate dehydrogenase (E1) ? subunit gene (PDHA1) in patients with a pyruvate dehydrogenase complex deficiency
2000-03
[45]
논문
Mutations of the E1β subunit gene (PDHB) in four families with pyruvate dehydrogenase deficiency
http://dx.doi.org/10[...]
2008-04
[46]
논문
Clinical and genetic spectrum of pyruvate dehydrogenase deficiency: Dihydrolipoamide acetyltransferase (E2) deficiency
http://dx.doi.org/10[...]
2005-07-27
[47]
논문
Pyruvate dehydrogenase complex deficiency: updating the clinical, metabolic and mutational landscapes in a cohort of Portuguese patients
2020-12
[48]
논문
Mitochondrial pyruvate dehydrogenase phosphatase 1 regulates the early differentiation of cardiomyocytes from mouse embryonic stem cells
2016-08
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com
