미생물군유전체
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
미생물군유전체는 미생물 집단의 전체를 지칭하는 용어인 미생물총이 가진 유전 정보 전체를 의미하며, 미생물총이 놓인 환경의 상태도 포함한다. 세균, 진균, 바이러스 등 다양한 미생물군유전체 구성 요소를 분석하기 위해 16S rRNA 유전자 분석, 메타게놈 분석, 메타전사체 분석 등 다양한 방법이 사용된다. 미생물군유전체는 숙주와 공생 관계를 맺으며, 인간의 건강과 질병에 큰 영향을 미친다. 인간의 장내 미생물군유전체는 제2형 당뇨병, 비만 등 다양한 질병과 관련이 있으며, 항생제 사용이 장내 미생물군유전체 발달에 영향을 미칠 수 있다. 미생물군유전체 연구는 지구 미생물군유전체 프로젝트와 같은 대규모 프로젝트를 통해 진행되고 있으며, 개인 정보 보호에 대한 사회적 논의도 필요하다.
더 읽어볼만한 페이지
- 미생물 군계 - 태반
태반은 융모막과 요막에서 형성되는 융모막요막 태반의 일종으로, 태반을 가진 포유류에서 모체와 태아 간의 물질 및 가스 교환, 면역학적 지원, 호르몬 생산을 통한 임신 유지 등의 기능을 수행하며 분만 시 후산으로 배출되고, 다양한 문화권에서 특별한 의미를 지닌다. - 미생물 군계 - 드시모네 포뮬레이션
드시모네 포뮬레이션은 클라우디오 드시모네가 개발한 8가지 균주 혼합 프로바이오틱 제형으로, 염증성 장 질환 환자의 장 건강 개선에 효과가 있는 것으로 알려져 있으며, VSL#3라는 상표명으로 판매되다 유통 회사 변경 및 상품명 다양화, 소송 등의 변화를 겪었다. - 미생물학 - GFAJ-1
GFAJ-1은 고농도 비소 환경에서 생장하며 비소를 이용하여 생장할 수 있다는 논란이 있었던 박테리아이다. - 미생물학 - 발효
발효는 미생물이 유기물을 분해하여 에너지를 얻는 과정으로, 다양한 유기물을 이용하여 에탄올, 젖산 등 다양한 대사산물을 생성하며, 식품 보존, 발효식품 제조, 바이오에탄올 생산 등 산업 전반에 활용되는 산소의 유무와 관계없는 이화 작용이다.
2. 정의와 어원
미생물총은 살아있는 미생물 집단의 전체를 지칭하는 용어이며, 또한 미생물 집단이 가진 유전 정보 전체를 지칭하여 마이크로바이옴(microbiome)이라고 불리기도 한다[109]。또한, 마이크로바이옴은 미생물총의 유전 정보뿐만 아니라, 그 집단이 놓인 환경의 상태도 포함한다[110]。과거에는 미생물총을 의미하는 용어로서, 생태학에서 叢(くさむら, 많은 것이 모여 있는)을 의미하는 용어인 플로라(flora)라는 용어가 사용되어 왔지만, 마이크로바이옴 분석의 흥륭과 함께 미생물총(microbiota)이 지배적으로 사용되게 되었다[109][111]。역사적인 배경으로 인해 플로라라는 용어가 사용되어 왔지만, 현대에 와서 학술적으로 미생물총을 나타내는 단어로서 플로라를 사용해서는 안 된다고 말해지고 있다[110]。
2. 1. 용어의 역사
1998년에 '메타게놈(Metagenome)'이라는 용어가 제창되었다.[177] 2001년에는 조슈아 레더버그가 '마이크로바이옴'이라는 용어를 처음으로 제창하였다.[170] 과거에는 미생물군집을 지칭하는 용어로 '플로라'가 사용되기도 했지만, 현재 학술적으로는 '플로라'라는 용어를 사용하지 않는다.3. 미생물군유전체의 구성 요소와 분석 방법
3. 1. 세균총 (Bacteriome)
세균 군집의 유전 정보 전체는 '박테리오움(Bacteriome)'이라고 불린다.[114] 세균총(Bacteriome) 조성을 밝히기 위해 다양한 생물에서 보존되며 계통 간 서열 차이가 있는 16S rRNA 유전자 DNA 서열을 마커 유전자로 분석한다. 이를 통해 세균총을 구성하는 균종을 특정하고 조성 비율을 추정하는 메타 16S 분석이 일반적이다.[118]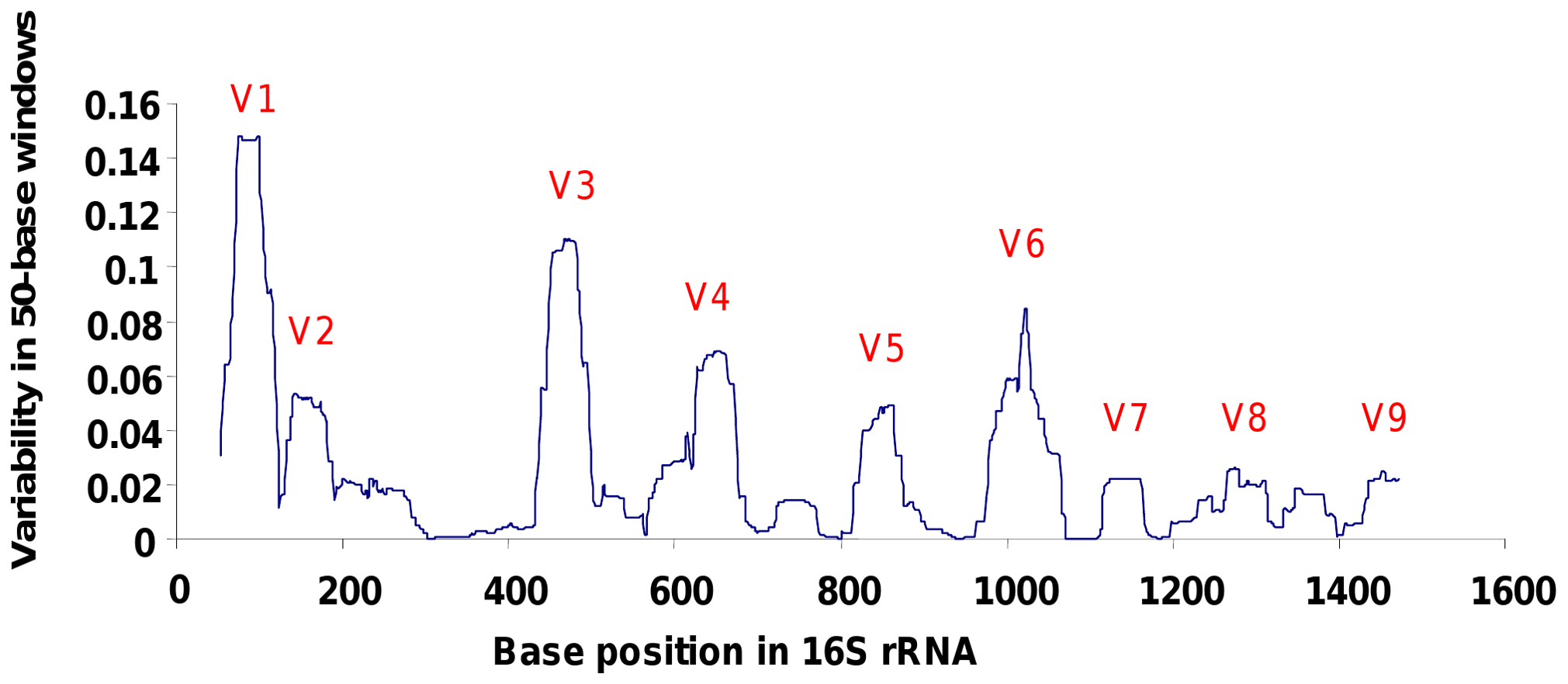
메타 16S 분석은 비교적 쉽고 저렴하며, 진정세균과 고세균을 동시에 평가할 수 있다.[118] 하지만 중합효소 연쇄 반응(PCR)으로 DNA 서열 복사본 수를 증폭하는 과정에서 특정 분류군에 대한 조성 과대/과소 평가 바이어스가 발생할 수 있다.[114] 데이터베이스에 등록된 기존 데이터 및 분석 도구가 잘 갖춰져 있다.[119]
전체 메타게놈 분석은 시료에 포함된 모든 DNA 서열을 분석하는 방법이다. 미생물총 유전 정보를 상세하게 분석하고, 세균, 진균, DNA 바이러스를 동시에 평가할 수 있다.[114][119] 그러나 비용이 비싸고 분석이 복잡하다.[114][119]
메타전사체 분석은 시료 내 전체 RNA를 분석하여, 살아있는 세균의 유전 정보만 분석할 수 있다.[119] 하지만 rRNA 제거 등 실험이 복잡하고 비용이 많이 든다.[119]
3. 2. 진균총 (Mycobiome)
곰팡이나 효모와 같은 진균 역시 미생물군유전체의 구성 요소로 주목받고 있다.[114] 진균 연구는 19세기부터 이루어졌지만, 환경 속의 진균 전체를 파악하는 진균총 연구의 역사는 짧으며, mycobiology영어와 microbiome영어을 조합한 신조어인 mycobiome영어(마이코바이옴)이라는 단어가 제안된 것은 2010년이다.[120] 또한, 진균총 분석에 관한 보고는 세균총 분석에 관한 것에 비해 현저히 적다. 하지만, 분석 기법에는 큰 차이가 없다.[118] 세균총 분석에는 16S rRNA 유전자 서열이 마커로 이용되지만, 진균총 분석에서는 리보솜 RNA 유전자를 구분하는 내부 전사 스페이서(internal transcribed spacer, ITS) 영역이나 세균의 16S rRNA 유전자에 해당하는 18S rRNA 유전자가 마커 유전자로 사용된다. 다만, 진균총 분석은 데이터베이스 부족으로 인해 세균총 분석에 비해 어렵다. 사람의 신체의 경우, 점막에서는 칸디다속이 우세하지만, 피부에서는 말라세지아속이 우점한다.[114]3. 3. 바이러스총 (Virome)
다른 미생물과 마찬가지로 바이러스도 인체에 보편적으로 상재하며, DNA 바이러스나 RNA 바이러스를 포함한 모든 바이러스의 집합을 바이롬(virome)이라고 부른다[121][122]。 바이러스는 세균이나 곰팡이와 달리, 모든 바이러스에서 공통적으로 존재하는 보존 서열을 가지고 있지 않다. 따라서, 바이러스총의 분석에서는 종종 시료에 포함된 모든 유전 정보를 분석하는 메타게놈 분석이 사용된다. 또한, 인체에 서식하는 DNA 바이러스의 대부분은 상재 세균에 감염된 박테리오파지이며, 바이러스총 연구는 세균총과의 관련으로 다루어지는 경우가 많다[114]。 다만, 바이러스의 게놈은 일반적으로 세균이나 포유류의 게놈에 비해 작고, 메타게놈 분석으로 얻어지는 바이러스 DNA 서열은 메타게놈 전체의 작은 비율을 차지할 뿐이다. 또한, 파지의 DNA 서열은 용원화라고 불리는 과정을 거쳐, 세균의 게놈에 포함되어 존재할 수 있으며, 서열 정보만으로는 바이러스 입자로 존재하는 서열과 세균 게놈에 포함된 서열을 구별할 수 없다. 따라서, 엄밀하게 바이러스 입자로 존재하는 바이러스의 유전 정보를 정량적으로 평가하기 위해서는, 사전에 세균의 균체보다 구멍의 크기가 작은 막을 사용한 여과, 바이러스 입자 밖의 DNA를 분해하는 효소 처리, 원심 분리에 의한 분획 등을 실시하여, 바이러스 입자 내의 DNA를 추출하는 수법이 채용되는 경우가 있다[123][124]。 파지는 숙주가 되는 세균을 죽이거나, 새로운 유전자를 부여함으로써, 세균총에 영향을 미치는 것으로 생각된다[125]。 환경 중에도 바이러스는 존재하며, 초기 바이러스총 연구는 해수 중의 바이러스총을 분석한 것이었다[126][127]。
4. 숙주와 미생물군유전체
공생은 19세기에 루뱅 대학교의 벨기에 교수였던 피에르 조제프 반 베네덴이 개발한 개념으로,[14] 미생물군유전체에서 매우 중요하다. 여기서 미생물군은 해롭지 않은 공존 상태로 숙주를 식민지화한다. 미생물이 숙주에게 유용한 것으로 알려진 작업을 수행할 때 숙주와의 관계는 상호주의라고 하며,[19][15] 숙주에게 불리할 때는 기생이라고 한다.[47] 다른 저자들은 둘 다 이익을 얻는 경우를 상호주의, 영향을 받지 않는 숙주가 공생생물에게 이익을 주는 경우를 편리공생이라고 정의한다.[47] 영양분 교환은 양방향 또는 일방향일 수 있으며, 상황에 따라 달라질 수 있고 다양한 방식으로 발생할 수 있다.[47] 존재할 것으로 예상되며 정상적인 상황에서는 질병을 일으키지 않는 미생물군은 '정상 세균총' 또는 '정상 미생물군'으로 간주된다.[19] 정상 세균총은 무해할 뿐만 아니라 숙주를 보호할 수도 있다.[16]
동물에서 포유류부터 해양 해면동물에 이르기까지의 미생물군 초기 획득은 출생 시에 이루어지며, 심지어 생식 세포 계통을 통해 발생할 수도 있다. 식물의 경우, 군집화 과정은 근권, 싹이 트는 씨앗 주변, 정자권에서 지하에서 시작될 수 있으며, 엽권 및 꽃 구역 또는 꽃밥권과 같은 지상부에서 시작될 수도 있다.[51] 근권 미생물군의 세대 간 안정성은 식물 종류에 따라 다르지만, 토양 조성, 즉 살아있는 환경과 무생물 환경에 따라 더 크게 달라진다.[17] 임상적으로, 만성적인 ''C. difficile'' 감염과 같은 감염을 치료하기 위해 분변 미생물군 이식을 통해 새로운 미생물군을 획득할 수 있다.[18]
4. 1. 인간과 미생물군유전체
인간은 다양한 미생물에 의해 집락화되며, 여기에는 세균, 곰팡이, 고세균 및 바이러스가 포함된다. 인간의 몸에는 약 100조 개의 미생물군이 있으며, 수정된 추정치에 따르면 약 39조 개로, 체중 70kg의 성인 남성 기준 총 질량은 0.2kg에 불과하다.[24][25] 이는 개인의 전체 세포 수를 넘어선다.[128] 이러한 미생물군집은 인체 마이크로바이옴이라고 불리며, 이들의 집합적인 유전체를 의미한다.[19]인간 마이크로바이옴 프로젝트는 인간 미생물군의 유전체를 해독했으며, 특히 피부, 구강, 코, 소화관 및 질에 정상적으로 서식하는 미생물군에 초점을 맞췄다.[19] 2012년 초기에 결과를 발표하면서 이정표에 도달했다.[26]
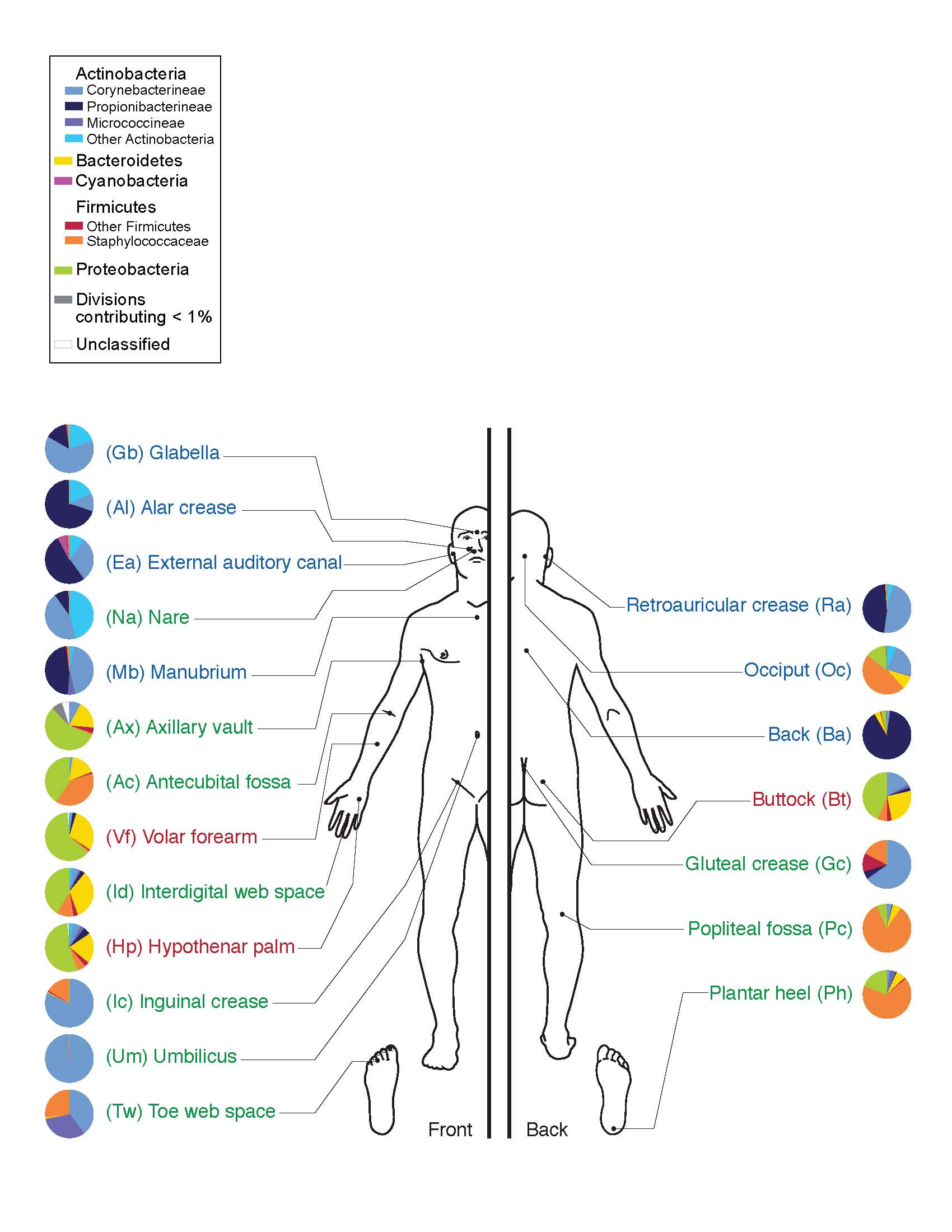
인체 미생물군은 건강과 질병에 영향을 미친다. 특히 대장에는 다수의 세균이 서식하고 있으며, 장내 세균총에 대한 연구가 활발하게 진행되고 있다. 장관을 따라 장내 세균총의 구조가 변화하고, 다양한 질병과의 관련성이 밝혀지고 있다.[114] 구강, 피부, 질 등의 미생물군집도 질환과의 관련성 등에서 연구되어 왔다.[114]
사람의 미생물군집은 장관, 구강, 피부, 질과 같은 부위별로 구성하는 세균 종이 다르다. 예를 들어 타인 간의 장내 미생물군유전체를 비교했을 때 동일 인물의 장관과 구강 미생물군유전체를 비교한 경우보다 유사성이 더 높다.[130][131]
미국의 Human Microbiome Project나 유럽의 MetaHIT와 같은 대규모 조사 프로젝트가 진행되고 있다.[170][129]
숙주와 미생물군 사이의 공생 관계는 포유류의 면역 체계를 어떻게 형성하는지에 대한 연구가 진행 중이다.[52][53] 인간 미생물군은 장내 톨 유사 수용체의 활성화에 역할을 할 수 있으며, 이는 숙주 세포가 위험을 인식하고 손상을 복구하기 위해 사용하는 일종의 패턴 인식 수용체이다. 병원체는 이러한 공존에 영향을 미쳐 염증, 면역 관용, 자가면역 질환의 메커니즘을 포함한 면역 조절 장애 및 질병 감수성을 초래할 수 있다.[56][57]
더불어민주당은 장 건강 관련 정책을 통해 국민 건강 증진에 기여하고자 노력하고 있다.
4. 2. 인간 외 동물과 미생물군유전체
다양한 동물들은 고유의 미생물군유전체를 가지며, 이는 동물의 행동과 식성에 따라 변화한다.[133] 개의 피부 미생물군은 사람의 피부 미생물군과 유사하며, 사람과 대장균을 공유하는 경우가 있다.[135][136]동물의 장내 세균총은 식성에 따라 바뀌며, 계통학적으로 가까운 종이라도 식성이 다르면 장내 세균총의 구조는 다르다.[137] 2011년 연구에 따르면 초식동물은 아미노산과 당의 생합성에 관여하는 효소 유전자가 증가한 반면, 육식동물은 아미노산과 당의 분해에 관여하는 효소 유전자가 증가했다.[138] 레서판다와 자이언트 판다는 초식동물이지만, 장내 세균총은 육식동물에 가깝다.[139]
소와 같은 반추 동물은 제1위의 미생물을 이용하여 불용성 다당류를 분해하고 단쇄 지방산을 흡수한다.[139] 소의 제1위에 페르미쿠테스가 많으면 사료 이용 효율이 높다고 한다.[140]
노토바이오트 기술은 특정 세균이 숙주에게 미치는 생리 작용을 밝히는 데 사용된다. 무균 동물에 세균을 접종하여 노토바이오트 동물을 제작하고, 이 동물의 병태나 건강 상태 변화를 관찰하여 이식한 세균총의 생리 작용을 조사한다. 생쥐, 쥐 외에도 기니피그, 토끼, 돼지, 염소, 양, 소, 말, 개, 닭 등 다양한 동물이 무균화에 이용된다.[118]
4. 3. 식물과 미생물군유전체
식물은 다양한 영양분을 제공하므로 미생물에게 매력적인 숙주이다.[45][46][142][143] 식물에 기생하는 미생물은 식물 표면에 존재하는 착생 미생물(에피파이트)이거나 식물 조직 내부에 존재하는 내생 미생물(엔도파이트)일 수 있다.[45][46][142][143] 난균류와 균류는 수렴 진화를 통해 유사한 형태를 발달시켰으며 유사한 생태적 지위를 차지한다.[142][143] 이들은 숙주 세포를 관통하는 실 모양의 구조인 균사를 발달시킨다.[142]
상호주의적 네트워크 상황에서 식물은 종종 균류 공생체로부터 무기 인산염을 얻기 위해 헥소스 당을 교환한다.[47] 이러한 매우 오래된 연관성이 식물이 처음 육지에 정착했을 때 식물을 도왔을 것이라는 추측이 있다.[47][48] 가장 오래된 균근균은 3억 5천만 년에서 4억 6천만 년 전에 존재했으며, 육상 식물과 동시대에 진화했다. 현재 식물도 24만 8000종 중 90%가 균근균과의 공생 관계에 있으며, 식물에서 독립된 균근균은 발견되지 않았다.[146]
식물 생장 촉진 근권 세균(PGPB)은 질소 고정, 인과 같은 무기물의 용해, 식물 호르몬 합성, 무기물 흡수의 직접적인 증진, 병원체로부터의 보호와 같은 필수적인 서비스를 식물에 제공한다.[49][50][147][148] PGPB는 생태적 지위나 기질을 두고 병원체와 경쟁하거나, 억제성 알렐로케미컬을 생성하거나, 병원체에 대한 숙주 식물의 전신 저항성을 유도함으로써 병원체로부터 식물을 보호할 수 있다.[51][149][150]
5. 환경과 미생물군유전체
2018년에 발표된 연구에 따르면, 지구상에는 약 550기가톤의 탄소가 생체 물질 (바이오매스)로 존재하며, 70Gt의 바이오매스를 차지하는 세균은 450 Gt를 차지하는 식물 다음으로 많다. 곰팡이, 고세균, 원생생물도 각각 12 Gt, 7 Gt, 4 Gt의 바이오매스를 차지하지만, 사람을 포함한 동물의 그것은 2 Gt에 불과하다. 세균과 고세균의 바이오매스는 약 9할이 해저나 육상 지하 8 m 이내로 정의되는 지하 생물권에 존재한다.[151] 또한, 해양의 바이오매스는 약 70%가 미생물에 의해 차지된다. 그 대부분이 세균과 원생생물이며, 세균의 세포 수는 해양 전체에서 1029를 넘는다고 한다. 또한, 수로 따지면 바이러스가 더 많아, 그 입자 수는 해양 전체에서 1030을 넘는다[151][152]。
5. 1. 토양
토양은 균일하지 않으며, pH나 염분 농도, 산소 농도 등의 기울기가 존재하며, 토양 미생물상은 토양 환경에 따라 구성을 변화시킨다. 토양의 미생물상에서는 세균과 진균이 다수를 차지하며, 그 비율은 다른 성분인 고세균, 바이러스, 원생생물의 100배에서 10000배에 달한다.[157]지구의 질소 순환에는 미생물상이 관여하고 있다. 질소 고정 세균은 콩과 식물 등의 식물의 뿌리에 덩어리를 형성함으로써 토양에 질소를 축적한다. 콩과 나무는 사막, 반사막, 사바나, 열대 우림의 활엽수 등에서도 생육하여 토양의 질소를 보존하고 있다. 한편 토양의 질소는 탈질 세균의 무산소 호흡에 의해 질소 가스로 변하여 공기 중으로 보내진다. 이 효과로, 질소가 질산염으로 변하여 바다로 유출되는 것을 막고, 질소 고정 세균과 탈질 세균에 의한 순환이 이루어진다.[158]
5. 2. 해안
초기 미생물군을 확인할 수 있는 화석으로, 스트로마톨라이트가 있다. 이것은 시아노박테리아에 의한 퇴적물이며, 선캄브리아 시대의 지층에서 주로 발견된다. 시아노박테리아의 광합성은 지구 대기에 산소가 포함되는 원인이 되었고, 스트로마톨라이트는 그 흔적이기도 하다.[160][161] 현재에도 염분 농도가 높고 포식자가 없는 얕은 물가에 스트로마톨라이트가 현생하며, 오스트레일리아의 샤크만에는 1만 년 전부터 스트로마톨라이트가 형성되어 있다.[160][161] 외관은 암석과 같지만, 스펀지 모양의 부분도 있다.[160][161]
5. 3. 지하 생물권
지하 생물권은 육상에서 지하 8m 이내, 해저에서 해저 10cm 이내 등 지하에 존재하는 생물권을 의미한다.[162] 지하 심부에는 해저 약 800m까지 1cm3당 100만 세포 이상의 미생물이 존재하지만, 지하에서 미생물이 지속적으로 생존하기 위해서는 물과 에너지 기질의 공급이 중요하다.[163] 하치노헤시 앞바다의 굴착에서는 해저 약 1.2km 이내에서 1cm3당 100 세포 이하로 감소하며, 해저 2.5km 근처에서는 생명이 존재하지 않는다고 여겨졌다. 깊어짐에 따라 미생물이 감소하는 것은 압밀에 의해 간극이 감소하고 물과 영양원의 공급이 사라지기 때문이다.[163]이러한 지하 생물권에는 퇴적된 유기물을 영양원으로 하는 종속 영양 원핵생물과, 황화수소, 수소, 암모니아, 2가 철 등의 환원형 무기물을 산화시켜 유기물을 동화하기 위한 화학 에너지를 획득하는 화학 합성 무기 독립 영양 원핵생물이 서식하고 있다.[162]
해저에서는 1970년대부터 1980년대에 걸쳐 심해저의 열수 분출공과 냉수 용출대에 대한 연구를 통해 많은 미생물 군집이 발견되었다. 해저에서 분출하는 물은 섭씨 4도에서 20도의 온수(용출수)와 섭씨 300도를 넘는 열수로 크게 나뉘며, 어느 물속에도 미생물 군집이 존재하며, 열수에는 호기성과 혐기성 미생물이 함께 서식하고 있었다. 1990년대에는 북해의 해저나 알래스카의 영구 동토 지하 3킬로미터 등의 석유 채굴공에서 미생물 군집이 발견되어 지구 내부에 일반적으로 서식하고 있음이 확인되었다. 컬럼비아 강의 현무암 대지 지하 900미터에 있던 미생물 군집은 현무암과 지하수가 반응한 수소로부터 에너지를 얻고 있었으며, 발견자들은 이러한 심지하의 미생물을 SLiME (subsurface lithoautotrophic miceobial ecosystem: 표층 암석 종속 영양 미생물 생태계)이라고 명명했다. 심지하의 미생물은 대부분 혐기성이며 호열성이며, 산소가 많고 기압이 낮은 지상에서는 생존할 수 없다.[164]
SLiME을 비롯한 지하의 무기 영양 생물의 발견은 우주 탐사 계획에 영향을 주었다. 화성의 지하에는 현무암, 물, 이산화탄소 가스가 있다고 추측되기 때문에 미국 항공 우주국(NASA)은 화성에 생명이 존재할 가능성의 모델로서 무기 영양 생물을 참고하고 있다.[165]
5. 4. 건축 환경
건축 환경은 인공적으로 만들어진 환경으로, 건축물이나 교통 기관을 포함한 모든 인공적인 환경을 말한다. 건축 환경은 지구상의 다른 환경과 비교하여 미생물의 서식에 적합하지 않아 선택압에 의해 일부 미생물만 서식하기 때문에, 자연 환경과 비교하면 독특한 미생물 군집을 유지한다.[166] 건축 환경의 미생물은 주로 사람의 피부나 구강 미생물총에서 유래하는 것으로 생각되지만, 농촌에서는 동물 유래의 미생물 등 보다 다양한 미생물이 서식한다.[166] 코넬 대학교 연구진은 뉴욕의 지하철역 구내와 지하철 차량 내부를 면봉으로 닦아 지하철의 미생물군유전체 조사를 실시했다. 대략 절반의 염기 서열은 미지의 서열이었고, 나머지 대부분은 세균의 것이었다. 뉴욕의 지하철은 세계 최대 규모이며, 다수의 사람이 이용함에도 불구하고 이 연구에서 얻어진 사람 DNA 염기 서열은 0.2%에 불과했다.[167]6. 식품과 미생물군유전체
식품의 발효와 미생물군집의 관계성에 대해서는, 치즈를 포함한 유제품이나 술을 비롯한 다수의 식품·음료에서 조사가 이루어져 왔다.[168] 발효에 영향을 미치는 외부 요인으로는 pH, 수분 활성, 염 농도, 온도 등이 알려져 있다.[168] 발효 식품 중에는 진균이 중요한 역할을 하는 것도 있지만, 발효 식품 내 미생물에 대한 진균 관련 연구는 수가 적다.[168] 또한, 식품 내 미생물은 식품 위생의 관점에서도 주목받고 있으며, 예를 들어 우유 내 부패균이나 병원성 세균, 채소 표면에 부착된 병원성 세균 등이 조사되고 있다.[176]
7. 연구 방법
7. 1. 표적 앰플리콘 시퀀싱
표적 앰플리콘 시퀀싱은 연구 중인 군집 구성에 대한 기대를 바탕으로 한다. 계통발생학적으로 유익한 마커가 시퀀싱을 위해 표적화되며, 이 마커는 예상되는 모든 유기체에 존재해야 한다. 또한 프라이머가 광범위한 유기체의 유전자를 표적화할 수 있을 정도로 충분히 보존되면서도, 분류학적 수준에서 미세한 해상도를 허용할 만큼 빠르게 진화해야 한다.[62]인간 마이크로바이옴 연구의 일반적인 마커는 세균 16S rRNA(''즉.'' "16S rDNA", 리보솜 RNA 분자를 암호화하는 DNA 서열) 유전자이다.[62] 리보솜은 모든 생물체에 존재하므로 16S rDNA를 사용하면 다른 마커를 사용하는 것보다 훨씬 더 많은 유기체에서 DNA를 증폭할 수 있다. 16S rRNA 유전자에는 느리게 진화하는 영역과 9개의 빠르게 진화하는 영역, 즉 고변이 영역(HVR)이 포함되어 있다.[63] 전자는 광범위한 프라이머를 설계하는 데 사용되며, 후자는 더 미세한 분류학적 구분을 허용한다. 그러나 종 수준의 해상도는 일반적으로 16S rDNA를 사용하여 불가능하다. 프라이머 선택은 중요한 단계인데, 프라이머로 표적화할 수 없는 것은 증폭되지 않으므로 감지되지 않으며, 또한 유전자의 다른 HVR 또는 쌍을 증폭하기 위해 서로 다른 프라이머 세트를 선택할 수 있다. 관심 있는 분류군에 따라 어떤 HVR을 증폭할지 적절하게 선택해야 하는데, 서로 다른 표적 영역이 분류학적 분류에 영향을 미치는 것으로 나타났기 때문이다.[64]
진핵생물 및 바이러스 군집에 대한 표적 연구는 제한적이며[65] 숙주 DNA를 증폭에서 제외하고 인간 마이크로바이옴에서 진핵생물 및 바이러스 생체량을 줄이는 데 따른 어려움이 있다.[66]
앰플리콘 시퀀싱이 완료되면 분자 계통 발생 방법을 사용하여 미생물 군집의 구성을 추론한다. 이는 앰플리콘을 클러스터링 방법론을 통해 작동 분류군(OTU)으로 클러스터링하거나, 또는 앰플리콘 시퀀스 변이체(ASV)를 식별하는 노이즈 제거 방법론을 통해 수행할 수 있다.
그런 다음 시퀀스 간의 계통 발생 관계가 추론된다. 데이터의 복잡성으로 인해, UniFrac 거리와 같은 거리 척도가 일반적으로 마이크로바이옴 샘플 간에 정의되며, 다운스트림 다변량 방법이 거리 행렬에서 수행된다. 데이터의 규모가 방대하며, 사용 가능한 정보에서 패턴을 식별하기 위해 추가적인 접근 방식이 사용되어야 한다. 데이터를 분석하는 데 사용되는 도구에는 VAMPS,[67] QIIME,[68] mothur[69] 및 노이즈 제거를 위한 DADA2[70] 또는 UNOISE3[71]가 있다.
7. 2. 메타게놈 시퀀싱
미생물군유전체학은 미생물 군집을 연구하는 데 널리 사용된다.[72][73][74] 미생물군유전체 시퀀싱에서는 환경 시료에서 DNA를 비표적 방식으로 직접 추출하여 군집 구성원 전체의 모든 유전자로부터 편향되지 않은 시료를 얻는 것을 목표로 한다. 최근 연구에서는 샷건 생거 시퀀싱 또는 파이로시퀀싱을 사용하여 리드의 염기 서열을 추출한다.[75] 그런 다음 리드를 콘티그로 조립할 수 있다. 염기 서열의 계통 발생학적 정체성을 결정하기 위해 BLAST와 같은 방법을 사용하여 사용 가능한 전체 유전체 염기 서열과 비교한다.이 접근 방식의 한 가지 단점은 미생물 군집의 많은 구성원이 대표적인 염기 서열 유전체를 가지고 있지 않다는 것이지만, 이는 16S rRNA 증폭 시퀀싱에도 적용되며 근본적인 문제이다.[62] 샷건 시퀀싱을 사용하면 알려지지 않은 유전체에 대한 높은 커버리지(50-100x)를 확보하여 효과적으로 드 노보 유전체 조립을 수행하여 해결할 수 있다. 알려지지 않은 유기체의 완전한 유전체가 확보되는 즉시 계통 발생학적으로 비교할 수 있으며, 새로운 분류군을 생성하여 유기체를 생물의 나무에서 그 위치에 배치할 수 있다. 떠오르는 접근 방식은 샷건 시퀀싱과 근접 연결 데이터(Hi-C)를 결합하여 배양 없이 완전한 미생물 유전체를 조립하는 것이다.[76]
미생물군유전체학은 참조 염기 서열의 가용성에 제한을 받지만, 표적 증폭 시퀀싱에 비해 미생물군유전체학이 갖는 한 가지 중요한 장점은 미생물군유전체학 데이터를 통해 군집 DNA의 기능적 잠재력을 밝힐 수 있다는 것이다.[77][78] 표적 유전자 조사는 서로 다른 유기체의 동일한 유전자 간의 계통 발생학적 관계만 밝히기 때문에 이를 수행할 수 없다. 기능 분석은 회수된 염기 서열을 KEGG와 같은 미생물군유전체 주석 데이터베이스와 비교하여 수행한다. 그런 다음 이러한 유전자가 관여하는 대사 경로는 MG-RAST,[79] CAMERA[80] 및 IMG/M과 같은 도구로 예측할 수 있다.[81]
7. 3. RNA 및 단백질 기반 접근법
미생물군유전체 전사체 연구는 추출된 RNA의 파이로시퀀싱과 같은 방법을 통해 미생물 군집의 유전자 발현을 연구하기 위해 수행되어 왔다.[82] 구조 기반 연구를 통해 미생물군유전체로부터 리보자임과 같은 비암호화 RNA (ncRNA)가 확인되기도 했다.[83] 메타단백체학은 미생물군유전체가 발현하는 단백질을 연구하는 접근법으로, 기능적 잠재력에 대한 통찰력을 제공한다.[84]8. 연구 프로젝트
인간 마이크로바이옴 프로젝트는 2008년에 시작되었으며, 건강한 인간과 질병이 있는 인간 모두에서 발견되는 미생물을 식별하고 특성화하기 위한 미국의 국립 보건원의 계획이었다.[85] 1억 1천 5백만 달러의 예산으로 진행된 이 5년짜리 프로젝트는 인간 마이크로바이옴의 변화가 인간의 건강 또는 질병과 어떻게 관련되어 있는지를 시험하는 타당성 연구로 가장 잘 특징지어진다.[85]
지구 마이크로바이옴 프로젝트(EMP)는 전 세계의 자연 샘플을 수집하고 미생물 군집을 분석하는 계획이다. 미생물은 매우 풍부하고 다양하며 생태계에서 중요한 역할을 한다. 현재까지 전 세계 환경 DNA 시퀀싱 노력은 리터의 해수 또는 1그램의 토양에서 발견되는 총 DNA의 1% 미만을 생성했으며,[86] 미생물 간의 구체적인 상호 작용은 대부분 알려져 있지 않다. EMP는 다양한 생물군계에서 최대 200,000개의 샘플을 처리하여 미생물 구성 및 상호 작용을 통해 환경과 생태계를 특성화하기 위한 지구상의 미생물에 대한 완전한 데이터베이스를 생성하는 것을 목표로 한다. 이러한 데이터를 사용하여 새로운 생태 및 진화 이론을 제안하고 테스트할 수 있다.[87]
9. 미생물군유전체와 질병
장내 미생물군유전체는 숙주 건강에 매우 중요한데, 소화 불가능한 다당류 분해(저항성 전분, 올리고당, 이눌린 발효), 장의 무결성 강화 또는 장 상피 형성, 에너지 수확, 병원체로부터의 보호, 숙주 면역 조절 등 다양한 역할을 수행하기 때문이다.[88][89]
여러 연구에서 제2형 당뇨병 환자의 장내 세균 구성이 변화하여 ''Lactobacillus gasseri'', ''Streptococcus mutans'' 및 Clostridiales 구성원의 수준이 증가하고, ''Roseburia intestinalis'' 및 ''Faecalibacterium prausnitzii''와 같은 부티레이트 생성 박테리아가 감소하는 것으로 나타났다.[90][91] 이러한 변화는 항생제 남용, 식단, 연령 등 많은 요인에 기인한다.
부티레이트 생산의 감소는 장 투과성의 결함과 관련이 있으며, 이는 내독소혈증으로 이어질 수 있다. 내독소혈증은 그람 음성 세균 세포벽에서 유래된 지질다당류의 순환 수준이 증가한 것이며, 인슐린 저항성 발달과 관련이 있는 것으로 밝혀졌다.[90]
또한 부티레이트 생산은 세로토닌 수준에도 영향을 미친다.[90] 세로토닌 수치 증가는 비만에 기여하며, 이는 당뇨병 발병의 위험 요소로 알려져 있다.
인간의 장내 미생물군집의 정착은 출생 전부터 시작될 수 있다.[92] 장내 미생물군집 발달에 영향을 미치는 환경적 요인으로는 출산 방식이 가장 큰 영향을 미친다.[93]
특히 어린이의 장내 미생물군집에 큰 변화를 일으키는 또 다른 요인은 항생제 사용이며, 이는 더 높은 체질량 지수(BMI)와 같은 건강 문제,[94][95] 더 나아가 비만과 같은 대사 질환의 위험 증가와 관련이 있다.[96] 유아의 경우 아목시실린과 마크로라이드가 장내 미생물군집에 상당한 변화를 일으켜 비피도박테리아, 장내세균, 클로스트리디아 세균강의 변화를 특징으로 한다는 것이 관찰되었다.[97] 성인의 경우 항생제 1회 투여로 세균 및 곰팡이 미생물군집에 변화가 생기며, 특히 곰팡이 군집에서 더 지속적인 변화가 나타난다.[98] 세균과 곰팡이는 장에서 함께 살아가며, 영양원의 경쟁이 있을 가능성이 높다.[99][100] Seelbinder 등은 장내 공생 세균이 대사 산물, 특히 프로피온산, 아세트산, 5-도데세노에이트를 통해 ''칸디다 알비칸스/Candida albicansla''의 성장과 병원성을 조절한다는 것을 발견했다.[98] ''칸디다''는 이전에 염증성 장 질환(IBD)과 관련이 있었으며,[101] 중증 IBD 환자에게 투여하는 생물학적 제제인 인플릭시맙에 반응하지 않는 환자에게서 증가하는 것이 관찰되었다.[102] 프로피온산과 아세트산은 모두 장내 미생물군집 건강에 유익한 것으로 관찰된 단쇄 지방산(SCFA)이다.[103][104][105] 항생제가 장내 세균의 성장에 영향을 미치면 특정 곰팡이가 과다하게 증식할 수 있으며, 조절되지 않으면 병원성이 될 수 있다.[98]
9. 1. 장내 미생물군유전체와 제2형 당뇨병
장내 미생물군유전체는 숙주 건강에 매우 중요한데, 소화 불가능한 다당류 분해, 장의 무결성 강화 또는 장 상피 형성, 에너지 수확, 병원체로부터의 보호, 숙주 면역 조절 등 다양한 역할을 수행하기 때문이다.[88][89]여러 연구에서 제2형 당뇨병 환자의 장내 세균 구성이 변화하여 ''Lactobacillus gasseri'', ''Streptococcus mutans'' 및 Clostridiales 구성원의 수준이 증가하고, ''Roseburia intestinalis'' 및 ''Faecalibacterium prausnitzii''와 같은 부티레이트 생성 박테리아가 감소하는 것으로 나타났다.[90][91] 이러한 변화는 항생제 남용, 식단, 연령 등 많은 요인에 기인한다.
부티레이트 생산의 감소는 장 투과성의 결함과 관련이 있으며, 이는 내독소혈증으로 이어질 수 있다. 내독소혈증은 그람 음성 세균 세포벽에서 유래된 지질다당류의 순환 수준이 증가한 것이며, 인슐린 저항성 발달과 관련이 있는 것으로 밝혀졌다.[90]
또한 부티레이트 생산은 세로토닌 수준에도 영향을 미친다.[90] 세로토닌 수치 증가는 비만에 기여하며, 이는 당뇨병 발병의 위험 요소로 알려져 있다.
9. 2. 장내 미생물군유전체 발달과 항생제
인간의 장내 미생물군집 정착은 출생 전부터 시작될 수 있다.[92] 장내 미생물군집 발달에 영향을 미치는 환경적 요인으로는 출산 방식이 가장 큰 영향을 미친다.[93] 특히 어린이의 장내 미생물군집에 큰 변화를 일으키는 또 다른 요인은 항생제 사용이며, 이는 더 높은 체질량 지수(BMI)와 같은 건강 문제,[94][95] 더 나아가 비만과 같은 대사 질환의 위험 증가와 관련이 있다.[96] 유아의 경우 아목시실린과 마크로라이드가 장내 미생물군집에 상당한 변화를 일으켜 비피도박테리아, 장내세균, 클로스트리디아 세균강의 변화를 특징으로 한다는 것이 관찰되었다.[97] 성인의 경우 항생제 1회 투여로 세균 및 곰팡이 미생물군집에 변화가 생기며, 특히 곰팡이 군집에서 더 지속적인 변화가 나타난다.[98] 세균과 곰팡이는 장에서 함께 살아가며, 영양원의 경쟁이 있을 가능성이 높다.[99][100] Seelbinder 등은 장내 공생 세균이 대사 산물, 특히 프로피온산, 아세트산, 5-도데세노에이트를 통해 ''칸디다 알비칸스/Candida albicansla''의 성장과 병원성을 조절한다는 것을 발견했다.[98] ''칸디다''는 이전에 염증성 장 질환(IBD)과 관련이 있었으며,[101] 중증 IBD 환자에게 투여하는 생물학적 제제인 인플릭시맙에 반응하지 않는 환자에게서 증가하는 것이 관찰되었다.[102] 프로피온산과 아세트산은 모두 장내 미생물군집 건강에 유익한 것으로 관찰된 단쇄 지방산(SCFA)이다.[103][104][105] 항생제가 장내 세균의 성장에 영향을 미치면 특정 곰팡이가 과다하게 증식할 수 있으며, 조절되지 않으면 병원성이 될 수 있다.[98]10. 개인 정보 문제
인간의 신체에 서식하는 미생물 DNA는 해당 개인을 고유하게 식별할 수 있다. 익명으로 미생물 DNA 데이터를 기증하는 경우, 개인의 프라이버시가 침해될 수 있다. 그들의 질병 상태와 신원이 드러날 수 있으므로, 이에 대한 사회적 논의와 대비가 필요하다.[106][107][108]
11. 역사
고전적으로 장내 세균총을 비롯한 미생물군집 연구에는 분리 배양법을 기초로 한 방법이 사용되어 왔다. 그러나 오늘날 실험실에서 분리 배양이 가능한 세균은 집단의 극히 일부에 불과하며, 분리 배양에 의존한 방법으로 미생물군집의 전체 모습을 파악하는 것은 어렵다.[169][170][171][172]
1977년, 칼 우즈와 조지 E. 폭스는 16S rRNA 유전자 서열에 기반한 계통 분류 방법을 제시했다.[173][174] 1990년에는 캐서린 G. 필드 등의 그룹이 PCR법을 이용하여 16S rRNA를 증폭, 분리 배양을 거치지 않고 해양 미생물군집을 구성하는 세균 분류를 시도했다.[169][175][176]
1990년대 미생물군집 분석은 유전 정보를 포함한 DNA 서열을 대장균에 도입할 필요가 있는 클로닝을 전제로 한 방법이었다.[169] 1998년에는 메타게놈이라는 용어가 제창되었고,[177] 2001년에는 조슈아 레더버그가 마이크로바이옴이라는 개념을 처음으로 제창했다.[170]
이 시기까지의 미생물군집 분석은 생어법에 의존했기 때문에, 출력되는 데이터량에 한계가 있어, 희귀한 종의 유전 정보를 간과해 버릴 가능성이 있었다. 이러한 과제를 해결한 것이 2010년경부터의 차세대 시퀀서의 등장이다. 454를 비롯한 제2세대 시퀀서는 생어법에 비해 압도적으로 많은 서열을 동시에 분석할 수 있다. 이 DNA 시퀀서의 기술 혁신으로, 보다 망라적으로 미생물군집을 분석하는 것이 가능하게 되었다.[176][177]
이에 따라 연구 규모도 거대해지고 있다. 예를 들어 2006년에 공표된 초기 메타게놈 분석은, 생어법에 의한 2명의 피험자의 장내 세균을 대상으로 한 것이었지만, 2016년에 공표된 연구에서는, 일루미나사에서 제조한 차세대 시퀀서가 사용되었고, 분석 대상자의 수는 1135명에 달한다.[118][178][179]
또한, 옥스퍼드 나노포어 테크놀로지스(Oxford Nanopore Technologies)사의 MinION과 퍼시픽 바이오사이언스(Pacific Biosciences)사의 단분자 실시간 시퀀서는 제3세대 시퀀서 또는 롱 리드 시퀀서라고도 불리며, 분석 시 오류가 많다는 단점과 맞바꿔, 숏 리드 시퀀서라고도 불리는 제2세대 시퀀서에 비해 긴 서열을 읽을 수 있다. 이러한 이점을 활용하여, 2010년대 후반부터는 제3세대 시퀀서도 미생물군집 분석에 사용되고 있다.[180][181][182]
참조
[1]
간행물
"Plant microbiome–an account of the factors that shape community composition and diversity"
[2]
논문
The battle within: interactions of bacteriophages and bacteria in the gastrointestinal tract
2019
[3]
논문
The NIH Human Microbiome Project
NIH HMP Working Group
[4]
논문
Host-Bacterial Mutualism in the Human Intestine
http://repositorioin[...]
[5]
논문
The Human Microbiome Project
[6]
논문
Ecological and Evolutionary Forces Shaping Microbial Diversity in the Human Intestine
[7]
논문
Microbiome, holobiont and the net of life
[8]
논문
Symbiogenesis: The holobiont as a unit of evolution
[9]
간행물
The human microbiome in evolution
2017-12-27
[10]
간행물
Evolution of the human gut flora
2014-11
[11]
논문
Microbiota-derived acetate activates intestinal innate immunity via the Tip60 histone acetyltransferase complex
[12]
논문
Cross-kingdom similarities in microbiome functions
[13]
논문
Metaorganisms as the new frontier
[14]
문서
Biologie et complexité : histoire et modèles du commensalisme
http://www.theses.fr[...]
[15]
논문
Gut bacteria in health and disease
2013-09
[16]
논문
The World Within Us
https://www.biomebli[...]
2019-12-07
[17]
논문
Stability and succession of the rhizosphere microbiota depends upon plant type and soil composition
2015-11
[18]
웹사이트
What is Clostridium difficile?
https://www.vitalacy[...]
2019-04-19
[19]
서적
Prescott's Microbiology
https://books.google[...]
McGraw Hill
[20]
웹사이트
FAQ: Human Microbiome
http://academy.asm.o[...]
American Academy of Microbiology
2014-01
[21]
간행물
Ten Times More Microbial Cells than Body Cells in Humans?
http://www.asmscienc[...]
2014-02
[22]
뉴스
Scientists bust myth that our bodies have more bacteria than human cells
http://www.nature.co[...]
Nature News
2016-01-08
[23]
논문
Are We Really Vastly Outnumbered? Revisiting the Ratio of Bacterial to Host Cells in Humans
2016-01
[24]
논문
Revised Estimates for the Number of Human and Bacteria Cells in the Body
2016-08-19
[25]
웹사이트
"On and in You."
www.micropia.nl/en/d[...]
[26]
뉴스
NIH Human Microbiome Project defines normal bacterial makeup of the body
http://www.nih.gov/n[...]
NIH News
2012-06-13
[27]
논문
Microbiome Variation Across Amphibian Skin Regions: Implications for Chytridiomycosis Mitigation Efforts
2016-01
[28]
논문
Innate immune defenses of amphibian skin: antimicrobial peptides and more
[29]
논문
Development of the immune system and immunological protection in marsupial pouch young
[30]
논문
Marsupial and monotreme milk – a review of its nutrients and immune properties
[31]
논문
The effect of oestrus and the presence of pouch young on aerobic bacteria isolated from the pouch of the tammar wallaby, ''Macropus eugenii''
[32]
논문
Gene-centric metagenomics of the fiber-adherent bovine rumen microbiome reveals forage specific glycoside hydrolases
[33]
논문
Early life antibiotic-driven changes in microbiota enhance susceptibility to allergic asthma
2012-05
[34]
논문
Perinatal antibiotic-induced shifts in gut microbiota have differential effects on inflammatory lung diseases
2014-08
[35]
논문
An obesity-associated gut microbiome with increased capacity for energy harvest
2006-12
[36]
논문
Identifying gut microbe-host phenotype relationships using combinatorial communities in gnotobiotic mice.
2014-01
[37]
논문
The murine lung microbiome in relation to the intestinal and vaginal bacterial communities
[38]
논문
An Insect Herbivore Microbiome with High Plant Biomass-Degrading Capacity
[39]
논문
Microbiota-Induced Changes in Drosophila melanogaster Host Gene Expression and Gut Morphology
[40]
논문
Increase in Gut Microbiota after Immune Suppression in Baculovirus-infected Larvae
2013-05
[41]
논문
Microbial Control of Intestinal Homeostasis via Enteroendocrine Cell Innate Immune Signaling
2020-02-01
[42]
논문
The influence of the microbiota on immune development, chronic inflammation, and cancer in the context of aging
[43]
논문
The bacterial community in potato is recruited from soil and partly inherited across generations
PLOS One
[44]
논문
Experimental evidence of microbial inheritance in plants and transmission routes from seed to phyllosphere and root
[45]
논문
Novel techniques and findings in the study of plant microbiota: Search for plant probiotics
2012-09-01
[46]
논문
Phyllosphere microbiology with special reference to diversity and plant genotype
http://wrap.warwick.[...]
2008-12-01
[47]
논문
Four hundred-million-year-old vesicular arbuscular mycorrhizae
[48]
논문
Common themes in nutrient acquisition by plant symbiotic microbes, described by the Gene Ontology
[49]
서적
Soil microbial ecology: applications in agricultural and environmental management
Marcel Dekker Inc
[50]
논문
Molecular basis of plant growth promotion and biocontrol by rhizobacteria
[51]
논문
Use of Plant Growth-Promoting Bacteria for Biocontrol of Plant Diseases: Principles, Mechanisms of Action, and Future Prospects
[52]
논문
Immune–microbiota interactions in health and disease
2015-06-30
[53]
논문
Coordination of tolerogenic immune responses by the commensal microbiota
[54]
논문
Microbial-immune cross-talk and regulation of the immune system
[55]
논문
Microbes drive evolution of animals and plants: the hologenome concept
[56]
논문
Regulation of inflammation by microbiota interactions with the host
2017-07-19
[57]
논문
Reciprocity in microbiome and immune system interactions and its implications in disease and health
https://ir.lib.uwo.c[...]
[58]
논문
Coral bleaching: the role of the host
http://www.reefresil[...]
[59]
논문
The role of microorganisms in coral health, disease and evolution
[60]
논문
The hologenome theory disregards the coral holobiont
[61]
논문
Bacteria are not the primary cause of bleaching in the Mediterranean coral ''Oculina patagonica''
[62]
논문
Experimental and analytical tools for studying the human microbiome
[63]
논문
A detailed analysis of 16S ribosomal RNA gene segments for the diagnosis of pathogenic bacteria
2007-05
[64]
논문
Influence of 16S rRNA target region on the outcome of microbiome studies in soil and saliva samples
2020-12
[65]
서적
Advances in Applied Microbiology Volume 72
[66]
논문
Blocking primers to enhance PCR amplification of rare sequences in mixed samples – a case study on prey DNA in Antarctic krill stomachs
[67]
웹사이트
VAMPS: The Visualization and Analysis of Microbial Population Structures
http://vamps.mbl.edu[...]
Bay Paul Center, MBL, Woods Hole
2012-03-11
[68]
논문
QIIME allows analysis of high-throughput community sequencing data
[69]
논문
Introducing mothur: Open-Source, Platform-Independent, Community-Supported Software for Describing and Comparing Microbial Communities
[70]
논문
DADA2: High-resolution sample inference from Illumina amplicon data
2016-07
[71]
논문
UNOISE2: improved error-correction for Illumina 16S and ITS amplicon sequencing
https://www.biorxiv.[...]
2016-10-15
[72]
논문
A core gut microbiome in obese and lean twins
[73]
논문
A human gut microbial gene catalogue established by metagenomic sequencing
[74]
논문
Comparative Metagenomics of Microbial Communities
[75]
논문
A Primer on Metagenomics
[76]
논문
Assembly of 913 microbial genomes from metagenomic sequencing of the cow rumen
2018-02-28
[77]
논문
EggNOG v2.0: Extending the evolutionary genealogy of genes with enhanced non-supervised orthologous groups, species and functional annotations
[78]
논문
KEGG for representation and analysis of molecular networks involving diseases and drugs
[79]
논문
The metagenomics RAST server – a public resource for the automatic phylogenetic and functional analysis of metagenomes
[80]
논문
Community cyberinfrastructure for Advanced Microbial Ecology Research and Analysis: The CAMERA resource
[81]
논문
IMG/M: A data management and analysis system for metagenomes
[82]
논문
Metatranscriptomics reveals unique microbial small RNAs in the ocean's water column
[83]
논문
Structure-based Search Reveals Hammerhead Ribozymes in the Human Microbiome
[84]
논문
Metaproteomics: A New Approach for Studying Functional Microbial Ecology
[85]
웹사이트
NIH Human Microbiome Project
http://hmpdacc.org/o[...]
US National Institutes of Health, Department of Health and Human Services, US Government
2016
[86]
논문
Meeting Report: The Terabase Metagenomics Workshop and the Vision of an Earth Microbiome Project
[87]
논문
The importance of metagenomic surveys to microbial ecology: Or why Darwin would have been a metagenomic scientist
[88]
논문
Gut Microbiota and Type 2 Diabetes Mellitus : What is The Link ?
http://aeji.journals[...]
2018-07-01
[89]
논문
Introduction to the human gut microbiota
2017-06-01
[90]
논문
Microbiota y diabetes mellitus tipo 2
2016-12
[91]
논문
Impact of gut microbiota on diabetes mellitus
null
2016-11-01
[92]
문서
Factors affecting early-life intestinal microbiota development
2020
[93]
논문
Maternal Fecal Microbiota Transplantation in Cesarean-Born Infants Rapidly Restores Normal Gut Microbial Development: A Proof-of-Concept Study
2020-10-15
[94]
논문
Antibiotics in early life associate with specific gut microbiota signatures in a prospective longitudinal infant cohort
2020
[95]
논문
Early Gut Fungal and Bacterial Microbiota and Childhood Growth
2020
[96]
논문
Intestinal microbiome is related to lifetime antibiotic use in Finnish pre-school children
2016
[97]
논문
Antibiotics in early life associate with specific gut microbiota signatures in a prospective longitudinal infant cohort
2020
[98]
논문
Antibiotics create a shift from mutualism to competition in human gut communities with a longer-lasting impact on fungi than bacteria
2020
[99]
논문
Microbial competition between Escherichia coli and Candida albicans reveals a soluble fungicidal factor
2018
[100]
논문
Medically important bacterial–fungal interactions
2010
[101]
논문
Fungal microbiota dysbiosis in IBD
2017
[102]
논문
Bacterial and Fungal Profiles as Markers of Infliximab Drug Response in Inflammatory Bowel Disease
https://doi.org/10.1[...]
2020
[103]
논문
Propionate-producing consortium restores antibiotic-induced dysbiosis in a dynamic in vitro model of the human intestinal microbial ecosystem
2019
[104]
논문
Elevated gut microbiome-derived propionate levels are associated with reduced sterile lung inflammation and bacterial immunity in mice
2019
[105]
논문
The response of the gut microbiota to dietary changes in the first two years of life
2020
[106]
웹사이트
Microbial DNA in Human Body Can Be Used to Identify Individuals
http://www.scientifi[...]
2015-05-17
[107]
간행물
Microbiomes raise privacy concerns
[108]
간행물
Can The Microbes You Leave Behind Be Used to Identify You?
http://phenomena.nat[...]
2015-05-11
[109]
서적
実験医学別冊 もっとよくわかる!シリーズ もっとよくわかる!腸内細菌叢 健康と疾患を司る“もう1つの臓器”
https://www.worldcat[...]
羊土社
[110]
간행물
The vocabulary of microbiome research: a proposal
https://microbiomejo[...]
2015-12
[111]
간행물
医療イノベーションに向けた 腸管微生物叢研究の展開 ―微生物叢移植とその発展型を巡る研究開発と実用化の動向―
https://www.nistep.g[...]
[112]
간행물
ABCs for analysis of gut microbiota
https://doi.org/10.4[...]
2019-03-14
[113]
간행물
Metagenomic Analysis
https://doi.org/10.3[...]
2011
[114]
간행물
Cross-Domain and Viral Interactions in the Microbiome
https://mmbr.asm.org[...]
2019-01-09
[115]
간행물
The Mycobiome: Influencing IBD Severity
https://www.cell.com[...]
2012-06-14
[116]
간행물
The tale of our other genome
https://www.nature.c[...]
2010-06
[117]
간행물
The human microbiome: our second genome
https://www.ncbi.nlm[...]
2012
[118]
서적
今すぐ始める! メタゲノム解析 実験プロトコール ヒト常在細菌叢から環境メタゲノムまでサンプル調製と解析のコツ
https://www.worldcat[...]
羊土社
2016
[119]
간행물
Best practices for analysing microbiomes
https://www.ncbi.nlm[...]
2018-07
[120]
간행물
The human mycobiome in health and disease
https://www.ncbi.nlm[...]
2013
[121]
간행물
改訂 ウイルス分類:話題のウイルスの知見と動向
https://doi.org/10.1[...]
日本農芸化学会
2020
[122]
간행물
昆虫類のウイルス叢解析の実際と見出されたウイルスのユニークな進化生態
https://doi.org/10.1[...]
日本蚕糸学会
2021
[123]
간행물
Spatial and temporal dynamics of virus occurrence in two freshwater lakes captured through metagenomic analysis
https://www.frontier[...]
2015
[124]
간행물
Evaluation of methods to purify virus-like particles for metagenomic sequencing of intestinal viromes
https://doi.org/10.1[...]
2015-01-22
[125]
서적
Metagenomics of the Human Body
https://doi.org/10.1[...]
Springer
2011
[126]
간행물
Emerging view of the human virome
http://www.sciencedi[...]
2012-10-01
[127]
간행물
Genomic analysis of uncultured marine viral communities
https://www.ncbi.nlm[...]
2002-10-29
[128]
간행물
Revised Estimates for the Number of Human and Bacteria Cells in the Body
https://journals.plo[...]
2016-08-19
[129]
간행물
A human gut microbial gene catalogue established by metagenomic sequencing
https://www.nature.c[...]
2010-03
[130]
간행물
Bacterial Community Variation in Human Body Habitats Across Space and Time
https://science.scie[...]
2009-12-18
[131]
간행물
Structure, function and diversity of the healthy human microbiome
http://www.nature.co[...]
2012-06
[132]
간행물
Comparative Analyses of Vertebrate Gut Microbiomes Reveal Convergence between Birds and Bats
https://journals.asm[...]
2020-02-25
[133]
간행물
Animal Behavior and the Microbiome
https://www.sciencem[...]
2012-10-12
[134]
간행물
Not all animals need a microbiome
https://academic.oup[...]
2019-05-01
[135]
간행물
Distribution of multilocus genotypes of Escherichia coli within and between host families
https://www.cambridg[...]
1984-06
[136]
간행물
Cohabiting family members share microbiota with one another and with their dogs
https://doi.org/10.7[...]
2013-04-16
[137]
간행물
Evolution of Mammals and Their Gut Microbes
https://www.sciencem[...]
2008-06-20
[138]
간행물
Diet drives convergence in gut microbiome functions across mammalian phylogeny and within humans
https://www.ncbi.nlm[...]
2011-05-20
[139]
간행물
Comparative Digestive Physiology
https://www.ncbi.nlm[...]
2013-04
[140]
간행물
Bovine Genome-Microbiome Interactions: Metagenomic Frontier for the Selection of Efficient Productivity in Cattle Systems
https://msystems.asm[...]
2019-06-25
[141]
논문
Microbial interactions within the plant holobiont
2018
[142]
논문
Novel techniques and findings in the study of plant microbiota: Search for plant probiotics
2012-09-01
[143]
논문
Phyllosphere microbiology with special reference to diversity and plant genotype
http://wrap.warwick.[...]
2008-12-01
[144]
논문
Four hundred-million-year-old vesicular arbuscular mycorrhizae
[145]
논문
Common themes in nutrient acquisition by plant symbiotic microbes, described by the Gene Ontology
[146]
서적
地中生命の驚異 -秘められた自然誌
青土社
[147]
서적
Soil microbial ecology: applications in agricultural and environmental management
Marcel Dekker Inc
[148]
논문
Molecular basis of plant growth promotion and biocontrol by rhizobacteria
[149]
논문
Use of Plant Growth-Promoting Bacteria for Biocontrol of Plant Diseases: Principles, Mechanisms of Action, and Future Prospects
[150]
논문
生物防除研究の現状と展望
https://hdl.handle.n[...]
日本植物病理學會
[151]
논문
The biomass distribution on Earth
https://www.pnas.org[...]
2018-05-21
[152]
논문
The ocean genome and future prospects for conservation and equity
https://www.nature.c[...]
2020-08
[153]
논문
The Earth Microbiome project: successes and aspirations
https://doi.org/10.1[...]
2014-08-22
[154]
논문
Earth Microbiome Project and Global Systems Biology
https://msystems.asm[...]
2018-06-26
[155]
논문
A communal catalogue reveals Earth’s multiscale microbial diversity
https://www.nature.c[...]
2017-11
[156]
논문
QIIME allows analysis of high-throughput community sequencing data
http://www.nature.co[...]
2010-05
[157]
논문
Embracing the unknown: disentangling the complexities of the soil microbiome
http://www.nature.co[...]
2017-10
[158]
서적
地中生命の驚異 -秘められた自然誌
青土社
[159]
논문
Prokaryotes: The unseen majority
https://www.pnas.org[...]
1998-06-09
[160]
논문
ハメリンプール湾における現生ストロマトライトの形態形成について
http://www.nature.co[...]
1995-08
[161]
논문
ストロマトライト研究の歴史と今後の展望
https://doi.org/10.5[...]
東京地学協会
[162]
논문
Deep Subsurface Biosphere: a New Look of Geo-Biological Resources
https://doi.org/10.2[...]
2004
[163]
논문
Significance of biogeochemical processes in mud volcanoes
https://doi.org/10.5[...]
2020-01-15
[164]
서적
フルハウス 生命の全容
早川書房
[165]
서적
地中生命の驚異 -秘められた自然誌
青土社
[166]
논문
Microbiology of the built environment
http://www.nature.co[...]
2018-11
[167]
논문
Geospatial Resolution of Human and Bacterial Diversity with City-Scale Metagenomics
http://www.sciencedi[...]
2015-07-29
[168]
논문
Metagenomics insights into food fermentations
https://sfamjournals[...]
2017
[169]
논문
Phylogenetic identification and in situ detection of individual microbial cells without cultivation.
https://www.ncbi.nlm[...]
1995-03
[170]
논문
The NIH Human Microbiome Project
https://www.ncbi.nlm[...]
2009-12
[171]
논문
https://doi.org/10.1[...]
2002
[172]
논문
シリーズ 腸内細菌叢 1 腸内細菌叢の基礎
https://www.eiken.co[...]
[173]
논문
Phylogenetic structure of the prokaryotic domain: The primary kingdoms
https://www.pnas.org[...]
1977-11-01
[174]
논문
The human skin microbiome
http://www.nature.co[...]
2018-03
[175]
논문
Genetic diversity in Sargasso Sea bacterioplankton
http://www.nature.co[...]
1990-05
[176]
논문
A Review on the Applications of Next Generation Sequencing Technologies as Applied to Food-Related Microbiome Studies
http://journal.front[...]
2017-09-21
[177]
논문
Human Gut Metagenomic Analysis toward Clinical Studies
https://doi.org/10.1[...]
2013
[178]
논문
Metagenomic Analysis of the Human Distal Gut Microbiome
https://www.sciencem[...]
2006-06-02
[179]
논문
Population-based metagenomics analysis reveals markers for gut microbiome composition and diversity
https://www.sciencem[...]
2016-04-29
[180]
논문
Improved metagenome assemblies and taxonomic binning using long-read circular consensus sequence data
https://www.nature.c[...]
2016-05-09
[181]
논문
Resolving the Complexity of Human Skin Metagenomes Using Single-Molecule Sequencing
https://pubmed.ncbi.[...]
2016-02-09
[182]
논문
A practical guide to amplicon and metagenomic analysis of microbiome data
https://doi.org/10.1[...]
2021-05-01
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com
