전사 (생물학)
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
전사(Transcription)는 유전 정보를 담고 있는 DNA를 주형으로 하여 RNA를 합성하는 생명 현상이다. 프랑수아 자코브와 자크 모노는 유전 물질이 단백질로 실현되는 과정을 처음으로 제시했으며, 로저 D. 콘버그는 진핵 세포 전사의 분자적 기초에 대한 연구로 노벨 화학상을 수상했다. 전사는 개시, 프로모터 이탈, 신장, 종결의 단계를 거치며, RNA 중합효소와 전사 인자들이 관여한다. 전사 조절에는 크로마틴 구조 조절, DNA 메틸화, 전사 인자 등이 중요한 역할을 하며, 암과 같은 질병의 발생과도 관련이 있다. 일부 바이러스는 RNA를 DNA로 역전사하는 능력을 가지고 있으며, 텔로머라아제는 텔로미어의 합성을 통해 세포의 불멸화에 기여한다.
더 읽어볼만한 페이지
2. 역사
프랑수아 자코브와 자크 모노는 유전 물질이 단백질로 실현되도록 하는 분자에 대한 가설을 처음으로 세웠다. 세베로 오초아는 폴리뉴클레오티드 인산분해효소를 사용하여 ''생체 외''(in vitro)에서 RNA를 합성하는 과정을 개발하여 유전 암호를 해독하는 데 기여한 공로로 1959년 노벨 생리학·의학상을 수상했다. 1965년까지 여러 연구실에서 ''생체 외'' RNA 중합 효소에 의한 RNA 합성이 확립되었지만, 이러한 효소에 의해 합성된 RNA는 전사를 올바르게 종료하는 데 필요한 추가 인자가 존재함을 시사하는 특성을 가지고 있었다.
DNA의 "비" 주형(센스) 가닥은 코딩 가닥이라고 불리는데, 이는 티민(T) 대신 유라실(U)이 사용되는 것을 제외하면 새로 생성되는 RNA 전사체와 서열이 동일하기 때문이다.[4] 전사는 DNA 복제보다 복사 정확도가 낮은데, 이는 전사가 DNA 복제에 비해 교정 메커니즘이 적고 덜 효과적이기 때문이다.[5]
로저 D. 콘버그는 "진핵 세포 전사의 분자적 기초에 대한 연구"로 2006년 노벨 화학상을 수상했다.[62]
3. 전사의 주요 단계
전사는 일반적으로 '''개시''', '''신장''', '''종결'''의 주요 단계로 나뉜다.[6] 세균은 세포질 내에서 비교적 단순한 전사 기구를 통해 전사를 수행하는 반면, 진핵생물은 세포핵 내에서 다수의 효소가 관여하는 복잡한 기구를 사용한다. 고세균은 세포질 내에서 전사를 수행하지만, 전사 기구는 진핵생물과 유사하다.[68]
RNA 중합 효소는 DNA의 안티센스 가닥을 3' 말단에서 5' 말단(3' → 5')으로 읽고, 상보적인 RNA를 5' → 3' 방향으로 생성한다. 이 방향성은 RNA 중합 효소가 성장하는 mRNA 사슬의 3' 말단에만 뉴클레오티드를 추가할 수 있기 때문이다.[2] DNA의 주형 가닥(template strand)은 전사 가닥 또는 (-)가닥이라고도 불리며, 비주형 가닥(nontemplate strand)은 비전사 가닥 또는 (+)가닥이라고도 불린다. (+)가닥은 유전 정보를 보유하고 있어 코딩 가닥(coding strand) 또는 센스 가닥(sense strand)이라고도 하며, (-)가닥은 안티코돈 가닥 또는 안티센스 가닥(antisense strand)이라고도 한다.[68]3. 1. 개시
DNA는 평상시 히스톤이라는 단백질과 함께 촘촘한 구조를 이루고 있어 전사 인자와 RNA 중합효소가 접근하기 어렵다. 전사가 일어나려면 히스톤에 아세틸기가 붙어 구조가 풀려야 한다.
전사가 일어날 위치에 여러 단백질이 붙어 DNA 두 가닥이 풀린다. 이때 한 가닥이 다시 두 가닥으로 되는 것을 막기 위해 특정 단백질(SSB)이 붙어 한 가닥 상태를 유지한다.
전사는 RNA 중합효소와 하나 이상의 전사 인자가 DNA 프로모터 서열에 결합하여 RNA 중합효소-프로모터 폐쇄 복합체를 형성하면서 시작된다. 폐쇄 복합체에서 프로모터 DNA는 이중 가닥 상태이다.[6]
RNA 중합효소는 전사 인자의 도움을 받아 DNA를 풀어 RNA 중합효소-프로모터 개방 복합체를 형성한다. 개방 복합체에서 프로모터 DNA는 부분적으로 풀려 단일 가닥이 되며, 이를 "전사 버블"이라고 한다.[6]
RNA 중합효소는 전사 버블에서 전사 시작 부위를 선택하고, 상보적인 NTP와 결합하여 초기 RNA 산물을 생성한다.[6]
세균의 RNA 중합효소 홀로효소는 2개의 α 서브유닛, 1개의 β 서브유닛, 1개의 β' 서브유닛, 1개의 ω 서브유닛으로 구성되며, 시그마 인자라는 하나의 일반 RNA 전사 인자가 있다. RNA 중합효소 코어 효소는 시그마 인자에 결합하여 RNA 중합효소 홀로효소를 형성한 다음 프로모터에 결합한다.[6]
고세균과 진핵생물의 RNA 중합효소는 세균의 5개 RNA 중합효소 서브유닛과 서열 상동성을 갖는 서브유닛 및 추가 서브유닛을 포함한다. 세균의 시그마 인자 기능은 여러 개의 일반 전사 인자가 함께 수행한다.[6] 고세균에는 TBP, TFB, TFE의 세 가지 일반 전사 인자가 있다. 진핵생물에서 RNA 중합효소 II 의존적 전사에는 TFIIA, TFIIB, TFIID, TFIIE, TFIIF, TFIIH의 여섯 가지 일반 전사 인자가 있다. TFIID는 TBP 결합으로 DNA에 결합하는 첫 번째 구성 요소이고, TFIIH는 가장 마지막으로 모집되는 구성 요소이다. 고세균과 진핵생물에서 RNA 중합효소-프로모터 폐쇄 복합체는 "전사 개시 전 복합체"라고 한다.[36]
전사 개시는 활성제 및 억제제와 같은 추가 단백질에 의해 조절될 수 있다.[6]
RNA 중합효소가 첫 번째 결합을 합성한 후에는 프로모터를 탈출해야 한다. 이 과정에서 RNA 전사체를 방출하고 절단된 전사체를 생성하는 경향이 있는데, 이를 초동적 개시라고 하며, 진핵생물과 원핵생물 모두에서 흔히 발생한다.[37] 초동적 개시는 약 10개 뉴클레오티드 길이의 RNA 생성물이 합성될 때까지 계속되며, 이후 프로모터 탈출이 일어나고 전사 연장 복합체가 형성된다. 프로모터 탈출은 DNA 스크런치를 통해 일어나며, RNA 중합효소 홀로효소와 프로모터 간의 상호작용을 끊는 데 필요한 에너지를 제공한다.[38]
진핵생물에서, RNA 중합효소 II 의존적 프로모터에서 프로모터 해제 시 TFIIH는 RNA 중합효소 II의 카르복시 말단 도메인에서 세린 5를 인산화시켜 캡핑 효소(CE)의 모집을 유도한다.[40][41]
3. 1. 1. 전사 인자
전사 인자는 전사 자체에 관여하는 '''기본 전사 인자'''와 전사를 조절하는 '''전사 조절 인자'''(제어 인자)로 나뉜다. 기본 전사 인자에는 RNA 중합 효소 복합체나 TATA 결합 단백질 등이 포함된다. 전사 개시 후 신장 반응에 기능하는 '''전사 신장 인자'''를 포함하기도 한다. 전사 조절 인자에는 전사 조절 서열의 DNA에 결합하여 기본 전사 인자의 활성을 제어하는 '''특이적 전사 인자'''가 포함된다. 직접 DNA에 결합하지 않고 크로마틴 구조 변환을 수행하는 히스톤 변형 효소나 크로마틴 재구성 인자도 포함된다.[68]
3. 1. 2. 프로모터

포유류에서 전사를 전사 개시 복합체로 설정하는 것은 시스 조절 요소를 포함하여 많은 요소에 의해 조절되며, 여기에는 유전자의 진핵생물 전사 전사 시작 부위 근처에 위치한 코어 프로모터 및 프로모터 근접 요소가 포함된다. 코어 프로모터는 일반 전사 인자와 결합하여 전사 개시를 지시하기에 충분하지만 일반적으로 낮은 기저 활성을 갖는다.[7] 다른 중요한 시스 조절 모듈은 전사 시작 부위에서 멀리 떨어진 DNA 영역에 국한되어 있다. 여기에는 증폭기, 억제 유전자, 절연체 및 고정 요소가 포함된다.[8] 이러한 요소들 중 증폭기와 관련 전사 인자는 유전자 전사의 개시에 주도적인 역할을 한다.[9] 유전자 프로모터에서 멀리 떨어진 DNA 영역에 국한된 증폭기는 유전자 전사에 매우 큰 영향을 미칠 수 있으며, 일부 유전자는 활성화된 증폭기로 인해 최대 100배 증가된 전사를 겪는다.[10]
증폭기는 주요 유전자 조절 요소인 게놈의 영역이다. 증폭기는 세포 유형별 유전자 전사 프로그램을 제어하며, 대부분 먼 거리를 루프하여 대상 유전자의 프로모터와 물리적으로 근접하게 된다.[11] 수십만 개의 증폭기 DNA 영역이 있는 반면,[12] 특정 유형의 조직의 경우 특정 증폭기만 해당 증폭기가 조절하는 프로모터와 근접하게 된다. 뇌 피질 뉴런 연구에서 24,937개의 루프가 발견되어 증폭기가 대상 프로모터로 이동했다.[10] 여러 증폭기는 각각 대상 유전자에서 수만 또는 수십만 개의 뉴클레오티드 떨어진 거리에 있으며, 대상 유전자 프로모터로 루프를 형성하여 공통 대상 유전자의 전사를 제어하기 위해 서로 조정할 수 있다.[11]
전사는 RNA 중합효소와 하나 이상의 전사 인자가 DNA 프로모터 서열에 결합하여 RNA 중합효소-프로모터 폐쇄 복합체를 형성하면서 시작된다. 폐쇄 복합체에서 프로모터 DNA는 여전히 완전히 이중 가닥이다.[6]
RNA 중합효소는 하나 이상의 일반 전사 인자의 도움을 받아 약 14개의 염기쌍의 DNA를 풀어서 RNA 중합효소-프로모터 개방 복합체를 형성한다. 개방 복합체에서 프로모터 DNA는 부분적으로 풀리고 단일 가닥이 된다. 노출된 단일 가닥 DNA는 "전사 버블"이라고 한다.[6]
RNA 중합효소는 하나 이상의 일반 전사 인자의 도움을 받아 전사 버블에서 '''전사 시작 부위'''를 선택하고, 전사 시작 부위 서열에 상보적인 개시 NTP와 연장 NTP(또는 짧은 RNA 프라이머와 연장 NTP)에 결합하며, 결합 형성을 촉매하여 초기 RNA 산물을 생성한다.[6]
세균에서 RNA 중합효소 홀로효소는 2개의 α 서브유닛, 1개의 β 서브유닛, 1개의 β' 서브유닛 및 1개의 ω 서브유닛으로 구성된다. 세균에는 시그마 인자라고 하는 하나의 일반 RNA 전사 인자가 있다. RNA 중합효소 코어 효소는 세균 일반 전사(시그마) 인자에 결합하여 RNA 중합효소 홀로효소를 형성한 다음 프로모터에 결합한다.[6]
고세균과 진핵생물에서 RNA 중합효소는 세균의 5개의 RNA 중합효소 서브유닛 각각과 서열 상동성을 갖는 서브유닛을 포함하며, 또한 추가 서브유닛을 포함한다. 고세균과 진핵생물에서 세균 일반 전사 인자 시그마의 기능은 함께 작용하는 여러 개의 일반 전사 인자에 의해 수행된다.[6] 고세균에는 세 개의 일반 전사 인자가 있다: TBP, TFB, TFE. 진핵생물에서 RNA 중합효소 II 의존적 전사에는 여섯 개의 일반 전사 인자가 있다: TFIIA, TFIIB(고세균 TFB의 상동 유전자), TFIID(핵심 서브유닛인 TBP가 고세균 TBP의 상동 유전자인 다중 서브유닛 인자), TFIIE(고세균 TFE의 상동 유전자), TFIIF, TFIIH. TFIID는 TBP 결합으로 인해 DNA에 결합하는 첫 번째 구성 요소인 반면 TFIIH는 가장 마지막으로 모집되는 구성 요소이다. 고세균과 진핵생물에서 RNA 중합효소-프로모터 폐쇄 복합체는 일반적으로 "전사 개시 전 복합체"라고 한다.[36]
전사 개시는 활성제 및 억제제라고 하는 추가 단백질과 경우에 따라 관련된 공동 활성제 또는 공동 억제제에 의해 조절되며, 이는 전사 개시 복합체의 형성 및 기능을 조절한다.[6]
RNA 중합효소가 첫 번째 결합을 합성한 후에는 프로모터를 탈출해야 한다. 이 과정에서 RNA 전사체를 방출하고 절단된 전사체를 생성하려는 경향이 있다. 이를 초동적 개시라고 하며, 진핵생물과 원핵생물 모두에서 흔히 발생한다.[37] 초동적 개시는 약 10개의 뉴클레오티드 길이의 임계점 이상의 RNA 생성물이 합성될 때까지 계속되며, 이 시점에서 프로모터 탈출이 일어나고 전사 연장 복합체가 형성된다.
기작적으로, 프로모터 탈출은 DNA 스크런치를 통해 일어나며, RNA 중합효소 홀로효소와 프로모터 간의 상호작용을 끊는 데 필요한 에너지를 제공한다.[38]
진핵생물에서, RNA 중합효소 II 의존적 프로모터에서 프로모터 해제 시 TFIIH는 RNA 중합효소 II의 카르복시 말단 도메인에서 세린 5를 인산화시켜 캡핑 효소(CE)의 모집을 유도한다.[40][41]
3. 2. 신장
두 가닥의 DNA 사슬 중 한 가닥을 주형으로 하여, RNA 중합효소가 관여하여 상보적인 염기를 가진 RNA 뉴클레오타이드가 차례로 결합하여 RNA를 합성한다. DNA 복제와 유사하게, 염기 T(티민) 대신 U(유라실)이 A(아데닌)과 결합하는 것을 제외하면, 상보적인 염기를 가진 뉴클레오타이드가 결합한다. 주형 DNA 사슬의 염기가 A이면 U를 갖는 뉴클레오타이드가, C(사이토신)이면 G(구아닌)을 갖는 뉴클레오타이드가 결합한다.[92]
새로운 RNA 뉴클레오타이드가 들어오면 RNA 염기와 주형 DNA 사슬의 염기가 일시적으로 염기쌍을 이루지만, 전사가 끝난 부분의 DNA는 다시 꼬여 원래의 DNA 이중 나선을 형성하고, RNA는 DNA 주형 가닥으로부터 분리된다. RNA 중합효소는 3'-OH에만 뉴클레오타이드의 인산을 결합시키기 때문에 RNA 합성은 5'에서 3' 방향으로 일어난다.[92]
DNA의 한 가닥인 ''주형 가닥''(비코딩 가닥)은 RNA 합성을 위한 주형으로 사용된다. RNA 중합효소는 주형 가닥을 3' → 5' 방향으로 이동하며, DNA 주형과의 염기쌍 상보성을 이용하여 RNA 복사본을 생성한다. 코딩(비주형) 가닥과 새로 형성된 RNA도 참조점으로 사용될 수 있으므로 전사는 5' → 3' 방향으로 발생한다고 할 수 있다. 이는 코딩 가닥과 동일하나 티민은 우라실로 대체되고, 뉴클레오티드는 리보스(5-탄소) 당으로 구성되는 반면 DNA는 당-인산 골격에 데옥시리보스(산소 원자 1개 적음)를 가진다.[3]
mRNA 전사는 단일 DNA 주형에 여러 RNA 중합효소를 포함할 수 있으며, 여러 번의 전사가 가능하므로 단일 유전자 복사본에서 많은 mRNA 분자를 빠르게 생성할 수 있다. 원핵생물과 진핵생물의 전형적인 신장 속도는 약 10~100 nt/초이다.[42] 진핵생물에서는 뉴클레오솜이 전사 신장 동안 전사 중합효소에 대한 주요 장벽 역할을 한다.[43][44] 이러한 유기체에서 뉴클레오솜에 의해 유도된 일시 정지는 TFIIS와 같은 전사 신장 인자에 의해 조절될 수 있다.[44]
신장에는 잘못 삽입된 염기를 교체할 수 있는 교정 메커니즘도 포함된다. 진핵생물에서 이것은 전사 중 짧은 일시 정지와 일치할 수 있다. 이러한 일시 정지는 RNA 중합효소 고유의 특성이거나, 염색질 구조 때문일 수 있다.
신장 단계에서 RNA 중합 효소는 결합된 DNA의 염기를 정확하게 식별하고 4개의 리보뉴클레오사이드 삼인산(ATP, GTP, CTP, UTP) 중에서 상보적인 것을 선택하여, 바로 앞 단계에서 합성된 짧은 RNA 가닥의 5' 말단에 부착한다. RNA 중합 효소와 전사 버블은 다음 염기로 이동하여 RNA 가닥에 리보뉴클레오사이드 삼인산을 부착한다. 이 반응은 RNA의 5' 말단에서 3' 말단으로, 역평행의 (-) 가닥에 대해서는 3' 말단에서 5' 말단으로 향한다. 지나간 DNA는 재회합하며, RNA는 신장하고 전사 산물이 만들어진다.
3. 3. 종결
RNA 중합효소는 DNA 주형 사슬 내의 종결 신호에 도달하면 RNA 합성을 멈추고 DNA로부터 떨어져 나온다. 새로 만들어진 RNA 가닥도 함께 떨어져 나온다.[48]
세균은 전사 종결을 위해 Rho-비의존적 종결과 Rho-의존적 종결, 두 가지 전략을 사용한다. Rho-비의존적 전사 종결에서는 G-C가 풍부한 헤어핀 루프를 형성하고 U가 연속적으로 나타나는 RNA 분자가 나타나면 RNA 전사가 멈춘다. 헤어핀 형성은 약한 rU-dA 결합을 끊는 기계적 스트레스를 유발하고, DNA-RNA 하이브리드를 채운다. 이는 폴리-U 전사체를 RNA 중합효소 활성 부위에서 빼내어 전사를 종결시킨다. Rho-의존적 종결에서는 단백질 인자인 Rho가 주형과 mRNA 간 상호작용을 불안정하게 하여, 새로 합성된 mRNA를 신장 복합체로부터 방출한다.[49]
진핵생물의 전사 종결은 세균보다 덜 알려져 있지만, 새로운 전사체 절단 후 폴리아데닐화 과정에서 주형에 의존하지 않고 새 3' 말단에 아데닌을 추가하는 과정을 포함한다.
DNA 손상이나 활성 복제 분기점과 같은 조건에서는 종결자 서열에 의한 종결 외에도 전사를 종결해야 한다. 세균에서 Mfd ATPase는 죔쇠를 열어 RNA 중합효소를 병변에서 제거하고, 뉴클레오티드 절제 복구 기구를 모집하여 병변을 복구한다. Mfd는 DNA 복제와 전사 간 충돌 해결 역할도 하는 것으로 제안되었다.[50] 진핵생물에서 ATPase TTF2는 세포 분열 동안 RNAP I과 II 작용을 억제하여 염색체 분리 오류를 방지한다.[51] 고세균에서는 Eta ATPase가 유사한 역할을 하는 것으로 제안되었다.[52]
RNA 중합 효소가 유전자 말단의 종결자에 도달하면 '''종결'''이 발생한다. 종결자는 RNA 중합 효소와 협력하여 전사 산물과 DNA 결합을 끊고 RNA 중합 효소와 DNA를 분리시킨다.
이때 터미네이터는 단백질 도움 없이 RNA 폴리머라제에 작용하여 전사 산물을 방출한다. '''내인성 터미네이터'''는 20nt 정도의 역위 반복 영역과 바로 아래 8nt 정도의 A(아데닌) 고함유 영역으로 구성된다.[68] 역위 반복 영역과 상보적인 RNA는 헤어핀 구조를 형성하며, 이는 전사 종결의 원동력이다.
페기 파넘과 테리 플랫은 대장균 트립토판오페론 연구를 통해 ρ 비의존성 종결의 전형적인 예를 보였다. 내인성 터미네이터 요소를 모두 갖춘 '''''trp'' 어테뉴에이터'''의 역위 반복 서열을 전사한 RNA는 아래와 같은 헤어핀 구조를 형성한다.[75] 완전한 역반복은 아니지만, 8bp 염기쌍 형성이 가능하며, 그중 7bp는 강한 G-C 염기쌍이다. 파넘과 플랫은 T 고함유 서열 전사 시 RNA의 U와 DNA의 A 사이 염기쌍(rU-dA)이 형성된다고 생각했다.[76] 이 염기쌍의 융해 온도는 rU-dA 또는 rU-rA보다 20℃ 낮다.[68] T 고함유 서열에서 RNA 폴리머라제가 일시 정지하고, 헤어핀 구조 형성으로 약한 결합이 끊어진다는 결론을 얻을 수 있다. 헤어핀 구조는 RNA 폴리머라제를 밀어 전사 산물에서 떼어내거나, 입체 구조 변화를 일으킨다.[77] W. S. 야넬과 제프리 로버츠는 전사 종결에 두 요소가 반드시 필요하지 않으며, 전사 산물과 극도로 약한 염기쌍을 형성하는 서열과 일시적 전사 정지 요소가 필요함을 밝혔다.[78]
A
/| U
U C G G G C G G A
| | | | | | | |
A G C C C G C C A
U
4. 원핵생물의 전사
원핵생물의 전사는 RNA 중합효소가 DNA에 붙으면서 시작된다. 이 효소는 DNA를 주형으로 하여 상보적인 RNA 분자를 합성하기 때문에 DNA-의존성 RNA 중합효소(DNA-dependent RNA polymerase)라고 부른다.[91]
원핵생물에서의 전사는 RNA 중합효소와 소수의 단백질이 DNA에 결합하며 시작된다. 이때 RNA 중합효소 외에 함께 결합하는 단백질은 전사 과정을 미세하게 조절하는 역할을 한다. 하지만 원핵생물의 전사에서 이러한 조절자의 역할은 진핵생물에 비해 매우 작다.
전사는 RNA 중합효소와 하나 이상의 전사 인자가 DNA 프로모터 서열에 결합하여 RNA 중합효소-프로모터 폐쇄 복합체를 형성하면서 시작된다. 폐쇄 복합체에서 프로모터 DNA는 여전히 이중 가닥이다.[6]
RNA 중합효소는 하나 이상의 일반 전사 인자의 도움을 받아 약 14개의 염기쌍의 DNA를 풀어서 RNA 중합효소-프로모터 개방 복합체를 형성한다. 개방 복합체에서 프로모터 DNA는 부분적으로 풀리고 단일 가닥이 된다. 이 노출된 단일 가닥 DNA를 "전사 버블"이라고 한다.[6]
RNA 중합효소는 전사 버블에서 '''전사 시작 부위'''를 선택하고, 전사 시작 부위 서열에 상보적인 개시 NTP와 연장 NTP(또는 짧은 RNA 프라이머와 연장 NTP)에 결합하며, 결합 형성을 촉매하여 초기 RNA 산물을 생성한다.[6]
세균에서 RNA 중합효소 홀로효소는 2개의 α 서브유닛, 1개의 β 서브유닛, 1개의 β' 서브유닛 및 1개의 ω 서브유닛으로 구성된다. 세균에는 시그마 인자라고 하는 하나의 일반 RNA 전사 인자가 있다. RNA 중합효소 코어 효소는 세균 일반 전사(시그마) 인자에 결합하여 RNA 중합효소를 형성한 다음 프로모터에 결합한다.[6] 진핵생물과 달리, 신생 세균 mRNA의 개시 뉴클레오티드는 변형된 구아닌 뉴클레오티드로 캡핑되지 않는다. 세균 전사물의 개시 뉴클레오티드는 5' 삼인산(5'-PPP)을 가지며, 이는 전사 개시 부위의 게놈 전체 매핑에 사용될 수 있다.[35]
고세균과 진핵생물에서 RNA 중합효소는 세균의 5개 RNA 중합효소 서브유닛 각각과 서열 상동성을 갖는 서브유닛을 포함하며, 추가 서브유닛도 포함한다. 고세균과 진핵생물에서 세균 일반 전사 인자 시그마의 기능은 여러 개의 일반 전사 인자가 함께 수행한다.[6] 고세균에는 TBP, TFB, TFE의 세 가지 일반 전사 인자가 있다.
전사 개시는 활성제 및 억제제라고 하는 추가 단백질과 경우에 따라 관련된 공동 활성제 또는 공동 억제제에 의해 조절되며, 이는 전사 개시 복합체의 형성 및 기능을 조절한다.[6]
전사 억제제는 항생제나 항진균제로 사용될 수 있다. 예를 들어 리팜피신은 DNA 의존성 RNA 중합 효소의 베타-서브유닛에 결합하여 세균의 전사를 억제하고, 8-하이드록시퀴놀린은 항진균 전사 억제제이다.[55]
4. 1. 대장균의 전사
대장균은 약 7000개의 RNA 중합효소를 가지고 있으며, 이 효소는 DNA를 주형으로 하여 상보적인 RNA 분자를 합성하기 때문에 DNA-의존성 RNA 중합효소(DNA-dependent RNA polymerase)라고 불린다.[91] 이 효소는 주형의 염기서열과 정확하게 상보적인 뉴클레오타이드를 선택하여 결합시킬 수 있을 뿐만 아니라, 전사할 유전자가 어디에 있는지도 찾아낼 수 있다.[91]원핵생물의 전사는 RNA 중합효소와 아주 적은 종류의 단백질이 함께 DNA에 붙으면서 시작한다. 이때 RNA 중합 효소 이외에 같이 붙은 단백질들은 전사의 미묘한 부분을 조정하는 조정자 역할을 하게 된다. 그러나 원핵생물의 전사에서 그러한 조정자의 역할은 진핵생물에서의 그것과 비교하면 매우 미미하다.
ρ 인자는 모두 같은 서브 유닛으로 구성된 육량체이다. 전체는 전사 산물과 DNA의 이중 나선을 통과하는 환상 구조를 이루고 있지만, 이 고리는 일부가 끊어져 있다. 게다가, 끊어진 부분을 사이에 둔 두 서브 유닛은 12Å 정도 고리의 축 방향으로 떨어져 있으며, 즉 작은 나선(피치는 45Å)을 이루고 있다.[68] 서브 유닛은 모두 RNA와 결합하는 부위를 가지며, 차례로 RNA에 결합하는 서브 유닛을 지그재그로 바꾼다고 생각된다.[68]
ρ 인자는 DNA의 전사 종결 부위 상류에 있는 '''ρ 인자 결합 부위'''(rho loading site)에서 전사 산물에 결합한다. 이 배열은 시토신을 많이 포함하는 60~100nt로, 비교적 임의적인 이차 구조를 형성하고 있다.[79] ρ 인자가 실제로 결합하기 위해서는 앞선 조건을 만족하는 40nt의 배열이 가장 좋다.[80] 이 ρ 인자 결합 부위를 전사함으로써, 실제로 맞이하는 '''rut 부위'''(Rho utilization sites:rut sites)라는 배열이 RNA에 나타난다. 단, 리보솜과 결합하여 중심 원리에서의 다음 단계인 번역을 받고 있는 것과는 결합할 수 없다.[68] 세균에서는 전사와 번역이 긴밀하게 연계되어, 신장이 아직 끝나지 않았더라도 중합 효소에서 나온 전사 산물은 즉시 번역을 시작할 수 있다.[81] 따라서 ρ가 집결하는 것은 유전자나 오페론의 말단을 지나도 전사가 계속되고 있는 전사 산물뿐이다.
6개의 서브 유닛은 모두 ATP가수분해효소이다. 이 활성에 의해, 필요 이상으로 길어진 전사 산물에 결합했을 때 에너지가 공급되어 회전하면서 5’에서 3’으로 진행한다. 터미네이터의 헤어핀 구조 형성으로 정지된 RNA 중합 효소에 따라잡으면, 전사 산물을 방출시킨다. 구체적인 종결 과정은 아직 단정되지 않았다. 1987년에 테리 프랫은 ρ 인자가 RNA-DNA 헬리케이스 활성을 가진다는 것을 발견했으므로, 한 설에서는 RNA 중합 효소와 접촉했을 때 이중 나선을 풀어서 방출시킨다고 한다.[68] 그 외에는, RNA 중합 효소를 뒤에서 밀어 전사 산물로부터 떼어낸다는 설과, RNA 중합 효소의 입체 구조를 변화시킨다는 설이 있다.[68]
5. 진핵생물의 전사
진핵생물에서의 전사는 원핵생물에서의 전사와 큰 차이를 보인다. 가장 큰 차이점은 RNA 중합효소 외에 전사인자라고 불리는 여러 조절자들이 관여한다는 것이다. 원핵생물과는 달리 진핵생물에서는 전사의 정도, 즉 전사의 양과 속도, 전사되는 단백질의 종류 등을 미묘하게 조절하기 위해 AP-1, NF-κB 등과 같은 수많은 단백질들이 필요하다.[34]
전사 인자들과 RNA 중합효소는 단계적으로 또는 한꺼번에 중합체(complex)를 이루면서 DNA의 특정 영역에 어느 정도의 세기로 붙을 것인지가 결정된다. 원핵생물에서의 전사가 RNA 중합효소와 DNA와의 직접적인 결합에 의해 대부분 조정되는 것에 비해, 진핵생물에서의 전사는 전사인자와 RNA 중합효소의 복합체와 DNA와의 결합에 의해 조절된다. 따라서 진핵생물에서의 전사는 단순히 RNA 중합효소와 DNA와의 결합이 아닌, 전사인자라는 조절자의 역할이 결정적인 역할을 한다.
단백질을 암호화하는 DNA 전사 단위는 단백질로 번역될 "코딩 서열"과 해당 단백질의 합성을 지시하고 조절하는 "조절 서열"을 모두 포함할 수 있다. 코딩 서열 앞(상류)의 조절 서열은 5' 비번역 부위 (5'UTR)라고 하고, 코딩 서열 뒤(하류)의 서열은 3' 비번역 부위 (3'UTR)라고 한다.[2]
DNA 복제와 달리, 전사는 DNA 상보체에서 티민 (T)이 발생했을 모든 경우에 유라실 (U) 뉴클레오티드를 포함하는 RNA 상보체를 생성한다.[3]
전사는 RNA 중합효소와 하나 이상의 전사 인자가 DNA 프로모터 서열에 결합하여 RNA 중합효소-프로모터 폐쇄 복합체를 형성하면서 시작된다. 폐쇄 복합체에서 프로모터 DNA는 여전히 완전히 이중 가닥이다.[6]
RNA 중합효소는 하나 이상의 일반 전사 인자의 도움을 받아 약 14개의 염기쌍의 DNA를 풀어서 RNA 중합효소-프로모터 개방 복합체를 형성한다. 개방 복합체에서 프로모터 DNA는 부분적으로 풀리고 단일 가닥이 된다. 노출된 단일 가닥 DNA는 "전사 버블"이라고 한다.[6]
RNA 중합효소는 하나 이상의 일반 전사 인자의 도움을 받아 전사 버블에서 '''전사 시작 부위'''를 선택하고, 전사 시작 부위 서열에 상보적인 개시 NTP와 연장 NTP(또는 짧은 RNA 프라이머와 연장 NTP)에 결합하며, 결합 형성을 촉매하여 초기 RNA 산물을 생성한다.[6]
고세균과 진핵생물에서 RNA 중합효소는 세균의 5개 RNA 중합효소 서브유닛 각각과 서열 상동성을 갖는 서브유닛을 포함하며, 또한 추가 서브유닛을 포함한다. 고세균과 진핵생물에서 세균 일반 전사 인자 시그마의 기능은 함께 작용하는 여러 개의 일반 전사 인자에 의해 수행된다.[6] 고세균에는 세 개의 일반 전사 인자가 있다: TBP, TFB, TFE. 진핵생물에서 RNA 중합효소 II 의존적 전사에는 여섯 개의 일반 전사 인자가 있다: TFIIA, TFIIB(고세균 TFB의 상동 유전자), TFIID(핵심 서브유닛인 TBP가 고세균 TBP의 상동 유전자인 다중 서브유닛 인자), TFIIE(고세균 TFE의 상동 유전자), TFIIF, TFIIH. TFIID는 TBP 결합으로 인해 DNA에 결합하는 첫 번째 구성 요소인 반면 TFIIH는 가장 마지막으로 모집되는 구성 요소이다. 고세균과 진핵생물에서 RNA 중합효소-프로모터 폐쇄 복합체는 일반적으로 "전사 개시 전 복합체"라고 한다.[36]
전사 개시는 활성제 및 억제제라고 하는 추가 단백질과 경우에 따라 관련된 공동 활성제 또는 공동 억제제에 의해 조절되며, 이는 전사 개시 복합체의 형성 및 기능을 조절한다.[6]
5. 1. RNA 중합효소
진핵생물에서의 전사는 원핵생물에서의 전사와 큰 차이를 보인다. 진핵생물에서는 RNA 중합효소 외에 전사인자라고 불리는 여러 조절 단백질들이 전사 과정에 관여하여 전사의 양과 속도, 전사되는 단백질의 종류 등을 정밀하게 조절한다. AP-1, NF-κB 등이 전사인자의 예시이다.전사 인자들과 RNA 중합효소는 단계적으로 또는 한꺼번에 복합체를 이루어 DNA의 특정 영역에 결합한다. 이 결합의 세기는 전사의 정도를 결정한다. 원핵생물에서는 RNA 중합효소와 DNA의 직접적인 결합으로 전사가 조절되는 반면, 진핵생물에서는 전사인자와 RNA 중합효소의 복합체와 DNA의 결합으로 조절된다. 즉, 진핵생물에서 전사는 전사인자의 역할이 매우 중요하다.[34]
전사는 RNA 중합효소와 하나 이상의 전사인자가 DNA 프로모터 서열에 결합하여 RNA 중합효소-프로모터 폐쇄 복합체를 형성하면서 시작된다. 폐쇄 복합체에서 프로모터 DNA는 이중 가닥 상태를 유지한다.[6]
RNA 중합효소는 전사 인자의 도움을 받아 약 14개 염기쌍의 DNA를 풀어 RNA 중합효소-프로모터 개방 복합체를 형성한다. 개방 복합체에서 프로모터 DNA는 부분적으로 풀려 단일 가닥이 되며, 이 부분을 "전사 버블"이라고 한다.[6]
RNA 중합효소는 전사 버블에서 '''전사 시작 부위'''를 선택하고, 전사 시작 부위 서열에 상보적인 NTP와 연장 NTP (또는 짧은 RNA 프라이머와 연장 NTP)에 결합하여 초기 RNA 산물을 생성한다.[6]
세균에서 RNA 중합효소 홀로효소는 2개의 α 서브유닛, 1개의 β 서브유닛, 1개의 β' 서브유닛 및 1개의 ω 서브유닛으로 구성된다. 세균에는 시그마 인자라고 하는 하나의 일반 RNA 전사 인자가 있다. RNA 중합효소 코어 효소는 시그마 인자에 결합하여 RNA 중합효소 홀로효소를 형성한 다음 프로모터에 결합한다.[6] 진핵생물과 달리, 세균 mRNA의 개시 뉴클레오티드는 변형된 구아닌 뉴클레오티드로 캡핑되지 않는다. 세균 전사물의 개시 뉴클레오티드는 5' 삼인산(5'-PPP)을 가지며, 이는 전사 개시 부위의 게놈 전체 매핑에 사용될 수 있다.[35]
고세균과 진핵생물에서 RNA 중합효소는 세균의 5개 RNA 중합효소 서브유닛 각각과 서열 상동성을 갖는 서브유닛을 포함하며, 추가 서브유닛도 포함한다. 고세균과 진핵생물에서 세균의 시그마 인자 기능은 여러 개의 일반 전사 인자가 함께 수행한다.[6] 고세균에는 TBP, TFB, TFE의 세 가지 일반 전사 인자가 있다. 진핵생물에서 RNA 중합효소 II 의존적 전사에는 TFIIA, TFIIB(고세균 TFB의 상동 유전자), TFIID(핵심 서브유닛인 TBP가 고세균 TBP의 상동 유전자인 다중 서브유닛 인자), TFIIE(고세균 TFE의 상동 유전자), TFIIF, TFIIH의 여섯 가지 일반 전사 인자가 있다. TFIID는 TBP 결합으로 DNA에 결합하는 첫 번째 구성 요소이고, TFIIH는 가장 마지막으로 모집되는 구성 요소이다. 고세균과 진핵생물에서 RNA 중합효소-프로모터 폐쇄 복합체는 "전사 개시 전 복합체"라고 불린다.[36]
전사 개시는 활성제 및 억제제라고 하는 추가 단백질과, 경우에 따라 관련된 공동 활성제 또는 공동 억제제에 의해 조절되며, 이들은 전사 개시 복합체의 형성 및 기능을 조절한다.[6]
RNA 중합효소가 첫 번째 결합을 합성한 후에는 프로모터를 탈출해야 한다. 이 과정에서 RNA 전사체를 방출하고 절단된 전사체를 생성하려는 경향이 있는데, 이를 초동적 개시라고 하며 진핵생물과 원핵생물 모두에서 흔히 발생한다.[37] 초동적 개시는 약 10개 뉴클레오티드 길이의 임계점 이상의 RNA 생성물이 합성될 때까지 계속되며, 이 시점에서 프로모터 탈출이 일어나고 전사 연장 복합체가 형성된다.
기작적으로, 프로모터 탈출은 DNA 스크런치를 통해 일어나며, RNA 중합효소 홀로효소와 프로모터 간의 상호작용을 끊는 데 필요한 에너지를 제공한다.[38]
세균에서, 역사적으로 시그마 인자는 프로모터 해제 후에 확실히 방출된다고 생각되었다. (''의무적 방출 모델'') 그러나 이후의 데이터는 프로모터 해제 시점과 그 이후에 시그마 인자가 확률적 모델에 따라 방출된다는 것을 보여주었다.[39]
진핵생물에서, RNA 중합효소 II 의존적 프로모터에서 프로모터 해제 시 TFIIH는 RNA 중합효소 II의 카르복시 말단 도메인에서 세린 5를 인산화시켜 캡핑 효소(CE)의 모집을 유도한다.[40][41] CE가 진핵생물에서 프로모터 해제를 유도하는 정확한 기전은 아직 알려지지 않았다.
DNA의 한 가닥인 ''주형 가닥''(또는 비코딩 가닥)은 RNA 합성을 위한 주형으로 사용된다. 전사가 진행됨에 따라 RNA 중합효소는 주형 가닥을 통과하며 DNA 주형과의 염기쌍 상보성을 사용하여 RNA 복사본을 생성한다(이는 통과하는 동안 신장됨). RNA 중합효소가 주형 가닥을 3' → 5' 방향으로 통과하지만, 코딩(비주형) 가닥과 새로 형성된 RNA도 참조점으로 사용될 수 있으므로 전사는 5' → 3' 방향으로 발생한다고 설명할 수 있다. 이는 코딩 가닥의 정확한 사본인 5' → 3' 방향의 RNA 분자를 생성한다(단, 티민은 우라실로 대체되고, 뉴클레오티드는 리보스(5-탄소) 당으로 구성되는 반면 DNA는 당-인산 골격에 데옥시리보스(산소 원자 1개 적음)를 가지고 있다).[3]
mRNA 전사는 단일 DNA 주형에 여러 RNA 중합효소를 포함할 수 있으며 여러 번의 전사(특정 mRNA 증폭)가 가능하므로, 단일 유전자 복사본에서 많은 mRNA 분자를 신속하게 생성할 수 있다. 원핵생물과 진핵생물의 전형적인 신장 속도는 약 10~100 nt/초이다.[42] 그러나 진핵생물에서는 뉴클레오솜이 전사 신장 동안 전사 중합효소에 대한 주요 장벽 역할을 한다.[43][44] 이러한 유기체에서 뉴클레오솜에 의해 유도된 일시 정지는 TFIIS와 같은 전사 신장 인자에 의해 조절될 수 있다.[44]
신장에는 잘못 삽입된 염기를 교체할 수 있는 교정 메커니즘도 포함된다. 진핵생물에서 이것은 적절한 RNA 편집 인자가 결합할 수 있도록 전사 중 짧은 일시 정지와 일치할 수 있다. 이러한 일시 정지는 RNA 중합효소 고유의 특성일 수도 있고, 염색질 구조 때문일 수도 있다.
DNA의 활발하게 전사되는 영역에서 이중 가닥 절단은 상동 재조합에 의해 세포 주기의 S기 및 G2기 동안 복구된다.[45][46] 전사는 재조합 유발 병변을 일으킬 수 있는 외부 화학 물질 및 내부 대사 산물에 대한 DNA의 접근성을 향상시키므로, 특정 DNA 염기서열의 상동 재조합은 전사에 의해 강력하게 자극될 수 있다.[47]
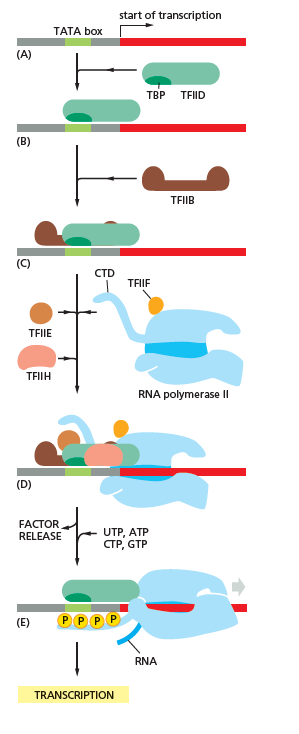
RNA 중합효소는 RNA의 전사 후 변화를 포함한 모든 단계에서 매우 중요한 역할을 한다.
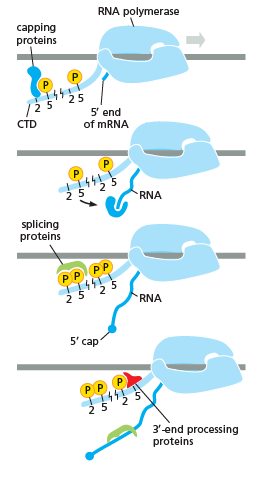
오른쪽 이미지에서 볼 수 있듯이 CTD(C-말단 도메인)는 모양을 변화시키는 꼬리 부분이며, 이 꼬리는 왼쪽 이미지에서 볼 수 있듯이 폴리아데닐화, 스플라이싱 및 캡핑의 운반체로 사용된다.[54]
RNA 중합효소 I에 의한 전사에서 프로모터는 -200~-65 범위에 존재하는 상류 조절 요소(UCE; upstream controling element)라고 불리는 영역과 -45~+20 범위에 존재하는 TATA 박스를 포함하는 코어 프로모터(CPE; core promoter element)라고 불리는 영역의 두 부분으로 구성된다.
전사에 관여하는 인자는 다음과 같다.
SL1은 다음 인자들의 복합체이다.
전사 신장에 관여하는 인자로는 TIF-1C도 알려져 있다.
RNA 중합효소 II에 의한 전사에 필요한 효소 이외의 기본 전사 인자 단백질을 TFII (Transcription Factor for Pol II) 시리즈라고 부르며, 현재 다음과 같은 종류가 알려져 있다.
전사 시작 후 RNA 신장을 촉진하는 인자로는 TFIIS도 존재한다.
전사 시작 흐름은 다음과 같다.
# 프로모터 영역(TATA 박스 근처)에 TFIIA가 결합
# TATA 박스에 TFIID가 결합, TFIIA와 복합체를 형성
# TFIIA-TFIID-DNA 복합체에 TFIIB가 결합
# RNA 중합효소 II(Pol II)에 TFIIF가 결합
# TFIIF-Pol II 복합체에 TFIIE가 결합
# TFIIF-TFIIE-Pol II 복합체에 TFIIH가 결합
# TFIIF-TFIIE-TFIIH-Pol II 복합체가 TFIIA-TFIID-TFIIB-DNA 복합체와 결합
# TFIIH의 작용에 의해 Pol II가 인산화된다
# Pol II 변형
# TFIIH의 작용에 의해, 전사 시작 복합체는 TFIIA-TFIID-TFIIB 복합체와 TFIIF-Pol II 복합체로 분리된다. 이때, TFIIE, TFIIH가 방출된다. ('''프로모터 클리어런스'''라고 한다)
# TFIIF-PolII-DNA 복합체에 TFIIS가 결합한다
# RNA의 전사가 진행된다
TATA 박스는 '''코어 프로모터 요소''' (CPE; core promoter element, 코어 프로모터 요소, 때로는 core promoter motif라고도 불린다) 중 하나이다.
'''코어 프로모터''' (core promoter)는 정확한 전사 시작을 유도하는 기능을 가진 프로모터 영역으로, 일반적으로 전사 시작점을 포함하는 ±35 염기 정도의 길이의 영역을 포함하지만, 많은 예에서 약 40 염기 영역으로 구성되어 있다고 생각된다. 코어 프로모터 안에는 몇 개의 시퀀스 모티프가 존재하며, 이것을 코어 프로모터 요소라고 부른다.
코어 프로모터 요소는 모든 코어 프로모터에 보편적으로 존재하는 것은 아니다. 오히려, 개별 코어 프로모터의 특이성을 부여하는 것이다. 교과서 등을 보면, TATA 박스가 모든 유전자의 코어 프로모터에 존재하고 있는 듯한 인상을 받지만, 실제로는 다르며, 예를 들어 효모에 관한 최근의 연구에서는 TATA-containing core promoter (TATA 박스를 포함하는 코어 프로모터)는 불과 약 19%였다고 보고하고 있다.
- 2~+4의 범위에 존재하는 '''이니시에이터 요소''' (Inr; initiator, PyPyANT/APyPy, A가 전사 시작 위치가 된다)도 코어 프로모터 요소이다. Inr의 인식은 TFIID에 의해 행해진다. 이 외에, DPE (downstream core promoter element), MTE (motif ten element)가 발견되었다.
RNA 중합효소 III에 의한 전사에 필요한 효소 이외의 기본 전사 인자 단백질을 TFIII (Transcription Factor for Pol III) 시리즈라고 부르며, 현재 TFIIIA, TFIIIB, TFIIIC의 세 가지가 알려져 있다. 그 외에도, TBP (TATA 결합 단백질), PTF (PSE 결합 단백질) 등의 인자도 존재한다.
RNA 중합효소 III에 의해 전사되는 유전자의 경우, 전사 개시의 기작이, 그 프로모터 영역의 구조에 따라 다음 세 가지 양식으로 나뉜다.
5. 2. 전사 조절 인자
진핵생물에서의 전사는 원핵생물과 달리 RNA 중합효소 외에 여러 조절 인자들이 관여하여 더 복잡하다. AP-1, NF-κB 등의 전사인자들은 전사의 양과 속도, 전사되는 단백질의 종류 등을 미세하게 조절하는 데 필요한 단백질들이다.전사 인자들과 RNA 중합효소는 단계적으로 또는 한꺼번에 복합체를 이루어 DNA의 특정 영역에 결합한다. 진핵생물에서의 전사는 전사인자와 RNA 중합효소의 복합체와 DNA의 결합에 의해 조절되므로, 전사인자는 RNA 중합효소와 DNA의 결합뿐만 아니라 전사 조절에 결정적인 역할을 한다.
포유류의 전사는 전사 개시 복합체를 포함한 많은 요소에 의해 조절된다. 여기에는 유전자의 진핵생물 전사 전사 시작 부위 근처에 위치한 코어 프로모터 및 프로모터 근접 요소와 같은 시스 조절 요소가 포함된다. 코어 프로모터는 일반 전사 인자와 결합하여 전사 개시를 지시하지만, 일반적으로 낮은 기저 활성을 갖는다.[7] 전사 시작 부위에서 멀리 떨어진 DNA 영역에는 증폭기, 억제 유전자, 절연체 및 고정 요소가 존재한다.[8] 이 중 증폭기와 관련 전사 인자는 유전자 전사 개시에 주도적인 역할을 한다.[9] 유전자 프로모터에서 멀리 떨어진 DNA 영역에 있는 증폭기는 유전자 전사에 큰 영향을 미칠 수 있으며, 일부 유전자는 활성화된 증폭기로 인해 전사가 최대 100배까지 증가한다.[10]
증폭기는 세포 유형별 유전자 전사 프로그램을 제어하며, 대부분 먼 거리를 루프하여 대상 유전자의 프로모터와 물리적으로 근접하게 된다.[11] 수십만 개의 증폭기 DNA 영역이 존재하지만,[12] 특정 유형의 조직에서는 특정 증폭기만 해당 증폭기가 조절하는 프로모터와 근접하게 된다. 뇌 피질 뉴런 연구에서는 24,937개의 루프가 발견되어 증폭기가 대상 프로모터로 이동했다.[10] 여러 증폭기는 각각 대상 유전자에서 수만 또는 수십만 개의 뉴클레오티드 떨어진 거리에 있으며, 대상 유전자 프로모터로 루프를 형성하여 공통 대상 유전자의 전사를 제어하기 위해 서로 조정할 수 있다.[11]
연결 단백질(예: CTCF 또는 YY1)의 이합체는 루프를 안정화하며, 이합체의 한 구성원은 증폭기의 결합 모티프에 고정되고 다른 구성원은 프로모터의 결합 모티프에 고정된다.[13] 여러 세포 기능 특정 전사 인자(인간 세포에는 약 1,600개의 전사 인자가 있다[14])는 일반적으로 증폭기의 특정 모티프에 결합하며,[15] DNA 루프에 의해 프로모터에 근접한 이러한 증폭기 결합 전사 인자의 작은 조합은 대상 유전자의 전사 수준을 제어한다. 중재자 (일반적으로 상호 작용하는 구조로 약 26개의 단백질로 구성된 복합체)는 증폭기 DNA 결합 전사 인자로부터 프로모터에 결합된 RNA 중합 효소 II(pol II) 효소로 직접 조절 신호를 전달한다.[16]
활성 증폭기는 일반적으로 RNA 중합 효소가 두 방향으로 작용하여 DNA의 두 가닥에서 전사되어 두 개의 증폭기 RNA(eRNA)를 생성한다.[17] 비활성 증폭기는 비활성 전사 인자에 결합될 수 있다. 전사 인자의 인산화는 이를 활성화할 수 있으며, 활성화된 전사 인자는 결합된 증폭기를 활성화할 수 있다.[18] 활성화된 증폭기는 대상 유전자에서 메신저 RNA의 전사를 활성화하기 전에 RNA의 전사를 시작한다.[19]
전사는 RNA 중합효소와 하나 이상의 전사 인자가 DNA 프로모터 서열에 결합하여 RNA 중합효소-프로모터 폐쇄 복합체를 형성하면서 시작된다. 폐쇄 복합체에서 프로모터 DNA는 여전히 완전히 이중 가닥이다.[6] RNA 중합효소는 하나 이상의 일반 전사 인자의 도움을 받아 약 14개의 염기쌍의 DNA를 풀어서 RNA 중합효소-프로모터 개방 복합체를 형성한다. 개방 복합체에서 프로모터 DNA는 부분적으로 풀리고 단일 가닥이 된다. 노출된 단일 가닥 DNA는 "전사 버블"이라고 한다.[6] RNA 중합효소는 하나 이상의 일반 전사 인자의 도움을 받아 전사 버블에서 '''전사 시작 부위'''를 선택하고, 전사 시작 부위 서열에 상보적인 개시 NTP와 연장 NTP (또는 짧은 RNA 프라이머와 연장 NTP)에 결합하며, 결합 형성을 촉매하여 초기 RNA 산물을 생성한다.[6]
세균에서 RNA 중합효소 홀로효소는 2개의 α 서브유닛, 1개의 β 서브유닛, 1개의 β' 서브유닛 및 1개의 ω 서브유닛으로 구성된다. 세균에는 시그마 인자라고 하는 하나의 일반 RNA 전사 인자가 있다. RNA 중합효소 코어 효소는 세균 일반 전사(시그마) 인자에 결합하여 RNA 중합효소 홀로효소를 형성한 다음 프로모터에 결합한다.[6] 진핵생물과 달리, 신생 세균 mRNA의 개시 뉴클레오티드는 변형된 구아닌 뉴클레오티드로 캡핑되지 않는다. 세균 전사물의 개시 뉴클레오티드는 5' 삼인산(5'-PPP)을 가지며, 이는 전사 개시 부위의 게놈 전체 매핑에 사용될 수 있다.[35]
고세균과 진핵생물에서 RNA 중합효소는 세균의 5개의 RNA 중합효소 서브유닛 각각과 서열 상동성을 갖는 서브유닛을 포함하며, 또한 추가 서브유닛을 포함한다. 고세균과 진핵생물에서 세균 일반 전사 인자 시그마의 기능은 함께 작용하는 여러 개의 일반 전사 인자에 의해 수행된다.[6] 고세균에는 세 개의 일반 전사 인자가 있다: TBP, TFB, TFE. 진핵생물에서 RNA 중합효소 II 의존적 전사에는 여섯 개의 일반 전사 인자가 있다: TFIIA, TFIIB(고세균 TFB의 상동 유전자), TFIID(핵심 서브유닛인 TBP가 고세균 TBP의 상동 유전자 인 다중 서브유닛 인자), TFIIE(고세균 TFE의 상동 유전자), TFIIF, TFIIH. TFIID는 TBP 결합으로 인해 DNA에 결합하는 첫 번째 구성 요소인 반면 TFIIH는 가장 마지막으로 모집되는 구성 요소이다. 고세균과 진핵생물에서 RNA 중합효소-프로모터 폐쇄 복합체는 일반적으로 "전사 개시 전 복합체"라고 한다.[36]
전사 개시는 활성제 및 억제제라고 하는 추가 단백질과 경우에 따라 관련된 공동 활성제 또는 공동 억제제에 의해 조절되며, 이는 전사 개시 복합체의 형성 및 기능을 조절한다.[6]
5. 2. 1. CpG 섬 메틸화

프로모터의 약 60%에서 전사 조절은 CpG 이중체 내 시토신 메틸화(5' 시토신이 3' 구아닌 또는 CpG 부위가 뒤따르는 경우)에 의해 제어된다. 5-메틸시토신(5-mC)은 메틸화된 형태의 DNA 염기 시토신이다(그림 참조). 5-mC는 주로 CpG 부위에서 발견되는 후성유전학적 표지자이다. 인간 게놈에는 약 2,800만 개의 CpG 이중체가 존재한다.[20] 포유류의 대부분의 조직에서 평균적으로 CpG 시토신의 70%~80%가 메틸화된다(5-메틸CpG 또는 5-mCpG 형성).[21] 그러나 5'시토신-구아닌 3' 서열 내에서 비메틸화된 시토신은 활성 프로모터에서 CpG 섬이라고 불리는 그룹으로 자주 발생한다. 프로모터 서열의 약 60%가 CpG 섬을 갖는 반면, 인핸서 서열의 약 6%만이 CpG 섬을 갖는다.[22] CpG 섬은 조절 서열을 구성하는데, 이는 유전자의 프로모터에서 CpG 섬이 메틸화되면 이로 인해 유전자 전사가 감소하거나 침묵될 수 있기 때문이다.[23]
DNA 메틸화는 MeCP2, MBD1 및 MBD2와 같은 메틸 결합 도메인(MBD) 단백질과의 상호 작용을 통해 유전자 전사를 조절한다. 이러한 메틸-CpG-결합 도메인 단백질은 고도로 메틸화된 CpG 섬에 가장 강력하게 결합한다.[24] 이러한 MBD 단백질은 메틸-CpG-결합 도메인과 전사 억제 도메인을 모두 가지고 있다.[24] 이들은 메틸화된 DNA에 결합하여 염색질 리모델링 및/또는 히스톤 변형 활성을 가진 단백질 복합체를 메틸화된 CpG 섬으로 안내하거나 지시한다. MBD 단백질은 일반적으로 억제성 히스톤 표지의 도입을 촉매하거나, 뉴클레오솜 리모델링 및 염색질 재구성을 통해 전반적인 억제성 염색질 환경을 생성하는 등 국소 염색질을 억제한다.[24]
EGR1 단백질은 CpG 섬의 메틸화를 조절하는 데 중요한 특정 전사 인자이다. EGR1 전사 인자 결합 부위는 인핸서 또는 프로모터 서열에 자주 위치한다.[26] 포유류 게놈에는 약 12,000개의 EGR1 결합 부위가 있으며, EGR1 결합 부위의 약 절반은 프로모터에, 절반은 인핸서에 위치한다.[26] EGR1이 표적 DNA 결합 부위에 결합하는 것은 DNA의 시토신 메틸화에 민감하지 않다.[26]
자극을 받지 않은 세포에서는 소량의 EGR1 전사 인자 단백질만 검출할 수 있지만, 자극 후 1시간에 ''EGR1'' 유전자가 단백질로 번역되는 것은 극적으로 증가한다.[27] 다양한 유형의 세포에서 EGR1 전사 인자 단백질의 생성은 성장 인자, 신경 전달 물질, 호르몬, 스트레스 및 손상에 의해 자극될 수 있다.[27] 뇌에서 뉴런이 활성화되면 EGR1 단백질이 상향 조절되고, 뉴런에서 고농도로 생성되는 기존의 TET1 효소에 결합(모집)한다. TET 효소는 5-메틸시토신의 탈메틸화를 촉매할 수 있다. EGR1 전사 인자가 TET1 효소를 프로모터의 EGR1 결합 부위로 가져오면 TET 효소는 해당 프로모터에서 메틸화된 CpG 섬의 DNA 탈메틸화를 수행할 수 있다. 탈메틸화 후, 이러한 프로모터는 표적 유전자의 전사를 시작할 수 있다. 뉴런에서 수백 개의 유전자는 EGR1이 프로모터에서 메틸화된 조절 서열에 TET1을 모집함으로써 뉴런 활성화 후에 차등적으로 발현된다.[26]
프로모터의 메틸화는 또한 신호에 반응하여 변경된다. 세 개의 포유류 DNA 메틸전달효소(DNMT1, DNMT3A 및 DNMT3B)는 DNA에서 시토신에 메틸기를 추가하는 것을 촉매한다. DNMT1은 유지 메틸전달효소인 반면, DNMT3A 및 DNMT3B는 새로운 메틸화를 수행할 수 있다. 또한 ''DNMT3A'' 유전자에서 생성되는 두 개의 스플라이스 단백질 아이소폼이 있다: DNA 메틸전달효소 단백질 DNMT3A1 및 DNMT3A2.[28]
스플라이스 아이소폼 DNMT3A2는 고전적인 즉시 초기 유전자의 산물처럼 행동하며, 예를 들어 신경 활성화 후에 강력하고 일시적으로 생성된다.[29] DNA 메틸전달효소 아이소폼 DNMT3A2가 결합하여 시토신에 메틸기를 추가하는 위치는 히스톤 번역 후 변형에 의해 결정되는 것으로 보인다.[30][31][32] 반면에, 신경 활성화는 DNMT3A1의 분해를 유발하여 평가된 표적 프로모터의 메틸화가 감소한다.[33]
6. 고세균의 전사
고세균에서의 전사는 기본적으로 진핵생물의 것을 단순화한 것으로 생각하면 된다. 전사 개시 위치 근처에는 진핵생물과 거의 같은 위치에 BRE, TATA 박스, 이니시에이터 엘리먼트 등이 배치되어 있다. TATA 박스에 RNA 중합 효소, TFIIB, TFIID, TFIIE로 구성된 복합체가 형성되고(TFIIA, TFIIF, TFIIH는 발견되지 않음), 이것들을 발판 삼아 RNA 중합 효소가 전사를 시작한다. TFIIS도 존재한다. 자세한 내용은 '''#Pol II계 유전자'''를 참조하면 된다.
7. 전사 조절
전사는 생물이 유전자에 기록된 DNA 염기 서열을 바탕으로 RNA를 합성하는 과정이다. 특히, 단백질 합성에 필요한 전령 RNA(mRNA)를 만드는 과정이 중요하다. 전사는 개시, 신장, 종결의 단계를 거치며, 다양한 전사 조절 인자가 전사의 활성화와 억제에 관여한다.[68]
RNA 중합 효소는 DNA 주형 가닥에 결합하여 상보적인 RNA 가닥을 합성한다.[68] DNA 상의 전사 범위를 지정하는 단위를 '''전사 단위''' 또는 '''오페론'''이라고 한다. 신장 단계에서는 RNA 중합 효소가 DNA 염기를 식별하고, 상보적인 리보뉴클레오사이드 삼인산(ATP, GTP, CTP, UTP)을 선택하여 RNA 가닥을 5'에서 3' 방향으로 신장시킨다.
전사 조절은 크게 크로마틴 구조 조절과 DNA 메틸화 조절로 나눌 수 있다.
7. 1. 크로마틴 구조 조절
DNA는 평상시에는 히스톤이라는 단백질과 함께 매우 촘촘한 구조(뉴클레오솜)를 이루고 있어, 전사 인자와 RNA 중합효소가 DNA에 접근할 수 없다. 즉, 평상시에는 전사가 일어날 수 없는 형태로 DNA는 존재하게 된다. 전사가 일어나기 위해서는 우선 히스톤에 아세틸기(아세트산 CH3COOH에서 하이드록시기 -OH를 떼어 낸 것)가 붙어서 히스톤의 모양이 바뀌고 DNA의 구조가 풀리게 된다. 일반적으로 히스톤이 아세틸화되면 크로마틴 구조가 느슨해지고, 결과적으로 발현이 활성화된다.또한, 초파리 등의 GAGA 인자라고 불리는 단백질은 DNA에 결합하여, 그 주변의 크로마틴 구조를 변화시킨다. NAP-1이 히스톤을 DNA에 부착하고, ACF가 그것을 이동시켜 일정 간격을 유지한다.
포유류의 전사를 전사 개시 복합체로 설정하는 것은 시스 조절 요소를 포함하여 많은 요소에 의해 조절되며, 여기에는 유전자의 진핵생물 전사 전사 시작 부위 근처에 위치한 코어 프로모터 및 프로모터 근접 요소가 포함된다.[7] 코어 프로모터는 일반 전사 인자와 결합하여 전사 개시를 지시하기에 충분하지만 일반적으로 낮은 기저 활성을 갖는다.[7] 다른 중요한 시스 조절 모듈은 전사 시작 부위에서 멀리 떨어진 DNA 영역에 국한되어 있다. 여기에는 증폭기, 억제 유전자, 절연체 및 고정 요소가 포함된다.[8] 이러한 요소들 중 증폭기와 관련 전사 인자는 유전자 전사의 개시에 주도적인 역할을 한다.[9]
증폭기는 주요 유전자 조절 요소인 게놈의 영역이다. 증폭기는 세포 유형별 유전자 전사 프로그램을 제어하며, 대부분 먼 거리를 루프하여 대상 유전자의 프로모터와 물리적으로 근접하게 된다.[11]
활성 증폭기는 일반적으로 그림에 표시된 대로 RNA 중합 효소가 두 방향으로 작용하여 DNA의 두 가닥에서 전사되어 두 개의 증폭기 RNA(eRNA)를 생성한다.[17] 비활성 증폭기는 비활성 전사 인자에 결합될 수 있다. 전사 인자의 인산화는 이를 활성화할 수 있으며, 활성화된 전사 인자는 결합된 증폭기를 활성화할 수 있다(그림에서 증폭기에 결합된 전사 인자의 인산화를 나타내는 작은 빨간 별 참조).[18] 활성화된 증폭기는 대상 유전자에서 메신저 RNA의 전사를 활성화하기 전에 RNA의 전사를 시작한다.[19] 전사 개시를 제어하는 억제자, 활성자가 있다.
세균에서는 HU 단백질이 DNA와 결합하여 핵양체를 형성하지만 크로마틴 구조를 이루지는 않는다.
한편, 많은 고세균은 히스톤을 가지고 있으며, DNA가 감겨 크로마틴 유사 구조를 이룬다.
7. 2. DNA 메틸화 조절
DNA 전사 조절의 약 60%는 CpG 이중체(5' 시토신이 3' 구아닌 또는 CpG 부위가 뒤따르는 경우) 내 시토신 메틸화에 의해 제어된다. 5-메틸시토신(5-mC)은 메틸화된 형태의 DNA 염기 시토신이다.[20] 5-mC는 주로 CpG 부위에서 발견되는 후성유전학적 표지자이다. 인간 게놈에는 약 2,800만 개의 CpG 이중체가 존재하며,[20] 포유류의 대부분의 조직에서 평균적으로 CpG 시토신의 70%~80%가 메틸화된다(5-메틸CpG 또는 5-mCpG 형성).[21] 그러나 5'시토신-구아닌 3' 서열 내에서 비메틸화된 시토신은 활성 프로모터에서 CpG 섬이라고 불리는 그룹으로 자주 발생한다. 프로모터 서열의 약 60%가 CpG 섬을 갖는 반면, 인핸서 서열의 약 6%만이 CpG 섬을 갖는다.[22] CpG 섬은 조절 서열을 구성하는데, 유전자 프로모터의 CpG 섬이 메틸화되면 유전자 전사가 감소하거나 침묵될 수 있기 때문이다.[23]
DNA 메틸화는 MeCP2, MBD1, MBD2와 같은 메틸 결합 도메인(MBD) 단백질과의 상호작용을 통해 유전자 전사를 조절한다. 이러한 메틸-CpG-결합 도메인 단백질은 고도로 메틸화된 CpG 섬에 가장 강력하게 결합하며,[24] 메틸-CpG-결합 도메인과 전사 억제 도메인을 모두 가지고 있다.[24] 이들은 메틸화된 DNA에 결합하여 염색질 리모델링 및/또는 히스톤 변형 활성을 가진 단백질 복합체를 메틸화된 CpG 섬으로 안내하거나 지시한다. MBD 단백질은 일반적으로 억제성 히스톤 표지의 도입을 촉매하거나, 뉴클레오솜 리모델링 및 염색질 재구성을 통해 전반적인 억제성 염색질 환경을 생성하는 등 국소 염색질을 억제한다.[24]
EGR1 단백질은 CpG 섬의 메틸화를 조절하는 데 중요한 전사 인자이다. EGR1 전사 인자 결합 부위는 인핸서 또는 프로모터 서열에 자주 위치한다.[26] 포유류 게놈에는 약 12,000개의 EGR1 결합 부위가 있으며, 이 중 약 절반은 프로모터에, 나머지 절반은 인핸서에 위치한다.[26] EGR1이 표적 DNA 결합 부위에 결합하는 것은 DNA의 시토신 메틸화에 민감하지 않다.[26]
자극을 받지 않은 세포에서는 소량의 EGR1 전사 인자 단백질만 검출되지만, 자극 후 1시간에 ''EGR1'' 유전자가 단백질로 번역되는 것은 극적으로 증가한다.[27] 다양한 유형의 세포에서 EGR1 전사 인자 단백질의 생성은 성장 인자, 신경 전달 물질, 호르몬, 스트레스 및 손상에 의해 자극될 수 있다.[27] 뇌에서 뉴런이 활성화되면 EGR1 단백질이 상향 조절되고, 뉴런에서 고농도로 생성되는 기존의 TET1 효소에 결합(모집)한다. TET 효소는 5-메틸시토신의 탈메틸화를 촉매할 수 있다. EGR1 전사 인자가 TET1 효소를 프로모터의 EGR1 결합 부위로 가져오면 TET 효소는 해당 프로모터에서 메틸화된 CpG 섬의 DNA 탈메틸화를 수행할 수 있다. 탈메틸화 후, 이러한 프로모터는 표적 유전자의 전사를 시작할 수 있다. 뉴런에서 수백 개의 유전자는 EGR1이 프로모터에서 메틸화된 조절 서열에 TET1을 모집함으로써 뉴런 활성화 후에 차등적으로 발현된다.[26]
프로모터의 메틸화는 신호에 반응하여 변경될 수도 있다. 세 개의 포유류 DNA 메틸전달효소(DNMT1, DNMT3A 및 DNMT3B)는 DNA에서 시토신에 메틸기를 추가하는 것을 촉매한다. DNMT1은 유지 메틸전달효소인 반면, DNMT3A 및 DNMT3B는 새로운 메틸화를 수행할 수 있다. 또한 ''DNMT3A'' 유전자에서 생성되는 두 개의 스플라이스 단백질 아이소폼인 DNA 메틸전달효소 단백질 DNMT3A1 및 DNMT3A2가 있다.[28]
스플라이스 아이소폼 DNMT3A2는 고전적인 즉시 초기 유전자의 산물처럼 행동하며, 신경 활성화 후에 강력하고 일시적으로 생성된다.[29] DNA 메틸전달효소 아이소폼 DNMT3A2가 결합하여 시토신에 메틸기를 추가하는 위치는 히스톤 번역 후 변형에 의해 결정되는 것으로 보인다.[30][31][32] 반면에, 신경 활성화는 DNMT3A1의 분해를 유발하여 평가된 표적 프로모터의 메틸화가 감소한다.[33]
8. 전사와 질병
게놈 손상은 자주 발생하며, 각 세포에서 매일 수만에서 수십만 건의 DNA 손상이 발생하는 것으로 추정된다.[53] 전사 과정은 손상에 취약한 단일 가닥 DNA 중간체를 형성하여 DNA 손상의 주요 원인이 된다.[1] 염기 절제 복구 및/또는 게놈을 절단하고 재구성하는 토포아이소머레이스를 사용하는 과정을 통한 전사의 조절 또한 DNA 손상 취약성을 증가시킨다.[1]
8. 1. 암과 전사
프로모터의 약 60%에서 전사 조절은 CpG 이중체 내 시토신 메틸화(5' 시토신 뒤에 3' 구아닌, 즉 CpG 부위가 오는 경우)에 의해 제어된다. 5-메틸시토신(5-mC)은 메틸화된 형태의 DNA 염기 시토신이다(그림 참조). 5-mC는 주로 CpG 부위에서 발견되는 후성유전학적 표지자이다. 인간 게놈에는 약 2,800만 개의 CpG 이중체가 있다.[20] 포유류 대부분의 조직에서 평균적으로 CpG 시토신의 70%~80%가 메틸화되어 5-메틸CpG (5-mCpG)를 형성한다.[21] 그러나 5'시토신-구아닌 3' 서열 내에서 비메틸화된 시토신은 활성 프로모터에서 CpG 섬이라고 불리는 그룹으로 자주 발생한다. 프로모터 서열의 약 60%가 CpG 섬을 갖는 반면, 인핸서 서열의 약 6%만이 CpG 섬을 갖는다.[22] CpG 섬은 조절 서열을 구성하는데, 유전자 프로모터에서 CpG 섬이 메틸화되면 유전자 전사가 감소하거나 침묵될 수 있기 때문이다.[23]
DNA 메틸화는 MeCP2, MBD1, MBD2와 같은 메틸 결합 도메인(MBD) 단백질과의 상호작용을 통해 유전자 전사를 조절한다. 이러한 메틸-CpG-결합 도메인 단백질은 고도로 메틸화된 CpG 섬에 가장 강력하게 결합한다.[24] MBD 단백질은 메틸-CpG-결합 도메인과 전사 억제 도메인을 모두 가지고 있다.[24] 이들은 메틸화된 DNA에 결합하여 염색질 리모델링 및/또는 히스톤 변형 활성을 가진 단백질 복합체를 메틸화된 CpG 섬으로 안내하거나 지시한다. MBD 단백질은 일반적으로 억제성 히스톤 표지의 도입을 촉매하거나, 뉴클레오솜 리모델링 및 염색질 재구성을 통해 억제성 염색질 환경을 생성하여 국소 염색질을 억제한다.[24]
EGR1 단백질은 CpG 섬의 메틸화를 조절하는 데 중요한 전사 인자이다. EGR1 전사 인자 결합 부위는 인핸서 또는 프로모터 서열에 자주 위치한다.[26] 포유류 게놈에는 약 12,000개의 EGR1 결합 부위가 있으며, 이 중 약 절반은 프로모터에, 절반은 인핸서에 위치한다.[26] EGR1이 표적 DNA 결합 부위에 결합하는 것은 DNA의 시토신 메틸화에 민감하지 않다.[26]
자극받지 않은 세포에서는 소량의 EGR1 전사 인자 단백질만 검출되지만, 자극 후 1시간에 ''EGR1'' 유전자가 단백질로 번역되는 것은 극적으로 증가한다.[27] EGR1 전사 인자 단백질 생성은 성장 인자, 신경 전달 물질, 호르몬, 스트레스, 손상 등 다양한 유형의 세포에서 자극될 수 있다.[27] 뇌에서 뉴런이 활성화되면 EGR1 단백질이 상향 조절되고, 뉴런에서 고농도로 생성되는 TET1 효소에 결합(모집)된다. TET 효소는 5-메틸시토신의 탈메틸화를 촉매할 수 있다. EGR1 전사 인자가 TET1 효소를 프로모터의 EGR1 결합 부위로 가져오면 TET 효소는 해당 프로모터에서 메틸화된 CpG 섬의 DNA 탈메틸화를 수행할 수 있다. 탈메틸화 후, 이러한 프로모터는 표적 유전자의 전사를 시작할 수 있다.
프로모터 메틸화는 신호에 반응하여 변경될 수도 있다. 세 개의 포유류 DNA 메틸전달효소(DNMT1, DNMT3A, DNMT3B)는 DNA에서 시토신에 메틸기를 추가하는 것을 촉매한다. DNMT1은 유지 메틸전달효소인 반면, DNMT3A와 DNMT3B는 새로운 메틸화를 수행할 수 있다. ''DNMT3A'' 유전자에서는 두 개의 스플라이스 단백질 아이소폼(DNA 메틸전달효소 단백질 DNMT3A1 및 DNMT3A2)이 생성된다.[28]
스플라이스 아이소폼 DNMT3A2는 고전적인 즉시 초기 유전자의 산물처럼 행동하며, 신경 활성화 후에 강력하고 일시적으로 생성된다.[29] DNA 메틸전달효소 아이소폼 DNMT3A2가 결합하여 시토신에 메틸기를 추가하는 위치는 히스톤 번역 후 변형에 의해 결정되는 것으로 보인다.[30][31][32] 반면, 신경 활성화는 DNMT3A1의 분해를 유발하여 평가된 표적 프로모터의 메틸화를 감소시킨다.[33]
척추동물에서, 대부분의 유전자 프로모터는 수많은 CpG 아일랜드를 포함하고 있다.[57] 유전자 프로모터의 많은 CpG 부위가 DNA 메틸화되면, 해당 유전자는 억제(침묵)된다.[58] 대장암은 일반적으로 3~6개의 드라이버 돌연변이와 33~66개의 히치하이커 또는 승객 돌연변이를 갖는다.[59] 그러나 전사 억제(침묵)는 암으로의 진행을 유발하는 데 있어 돌연변이보다 더 중요할 수 있다. 예를 들어, 대장암에서는 약 600~800개의 유전자가 CpG 아일랜드 메틸화에 의해 전사적으로 억제된다. 암에서 전사 억제는 후성유전학적 기전, 예를 들어 마이크로RNA의 변화된 생산과 같은 다른 기전에 의해서도 발생할 수 있다.[60]
9. 역전사
RNA 바이러스는 DNA로부터 이루어지는 통상의 전사와는 달리 RNA에 유전 정보를 기록하고, RNA로부터 mRNA 합성을 하지만, 이것도 전사의 범주에 포함된다. 다만, 핵산에서 핵산으로의 합성은 DNA 복제 과정에서도 이루어지지만, 이는 생성물이 DNA이며, 유전자의 발현에 직접 관여하지 않기 때문에 전사와는 다르다. mRNA로부터 DNA로의 합성은 역전사라고 부른다.
일부 바이러스는 RNA를 DNA로 전사하는 능력을 가지는데, 이러한 과정을 역전사라고 한다. 대표적인 예로 인간 면역 결핍 바이러스(HIV)가 있으며, 이와 관련된 자세한 내용은 하위 섹션을 참고할 수 있다.
9. 1. 레트로바이러스와 역전사
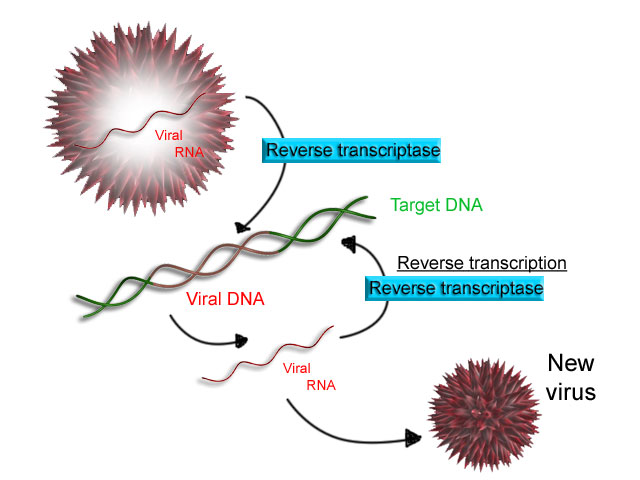
일부 바이러스(예: AIDS의 원인인 HIV)는 RNA를 DNA로 전사하는 능력이 있다. HIV는 RNA 유전체를 가지고 있으며, 이는 DNA로 ''역전사''된다. 결과 DNA는 숙주 세포의 DNA 유전체와 병합될 수 있다. RNA 주형으로부터 DNA를 합성하는 주요 효소는 역전사 효소라고 불린다.[65]
HIV의 경우, 역전사 효소는 바이러스 RNA 유전체에 대한 상보적 DNA 가닥(cDNA)을 합성하는 역할을 한다. 그 다음 효소 리보핵산 분해 효소 H가 RNA 가닥을 분해하고, 역전사 효소는 DNA의 상보적 가닥을 합성하여 이중 나선 DNA 구조(cDNA)를 형성한다. cDNA는 효소 인테그라제에 의해 숙주 세포의 유전체에 통합되어 숙주 세포가 새로운 바이러스 입자로 재조립되는 바이러스 단백질을 생성하게 한다. HIV의 경우, 이후 숙주 세포는 T 세포의 프로그램된 세포 죽음, 즉 세포 자멸사를 겪는다.[66] 그러나 다른 레트로바이러스에서는 바이러스가 세포에서 싹이 터 나올 때 숙주 세포가 손상되지 않은 상태로 유지된다.
일부 진핵세포는 텔로머라아제라고 하는 역전사 활성을 가진 효소를 포함한다. 텔로머라아제는 선형 염색체의 끝에 DNA의 반복 서열인 텔로미어를 합성하는 RNA 주형을 운반한다. 이는 선형 염색체가 복제될 때마다 염색체가 짧아지기 때문에 중요하다. 염색체 끝에 텔로미어가 있으면 단축으로 인해 염색체 끝에서 멀리 떨어진 단백질 암호화 DNA 서열이 아닌 일부 비필수 반복 서열이 제거된다.
텔로머라아제는 종종 암세포가 중요한 단백질 암호화 DNA 서열을 잃지 않고 유전체를 무한정 복제할 수 있도록 활성화된다. 텔로머라아제의 활성화는 암세포가 불멸화되는 과정의 일부일 수 있다. 텔로머라아제에 의한 텔로미어 연장을 통한 암의 불멸화 인자는 모든 발암성 종양의 90%에서 ''생체 내''에서 발생하는 것으로 입증되었으며, 나머지 10%는 ALT 또는 텔로미어의 대체 연장이라는 대체 텔로미어 유지 경로를 사용한다.[67]
참조
[1]
journal
Tentative identification of RNA-dependent RNA polymerases of dsRNA viruses and their relationship to positive strand RNA viral polymerases
1989-07-01
[2]
서적
Biology, 8th Edition, International Student Edition
Thomson Brooks/Cole
[3]
웹사이트
Molecular Biology
https://books.google[...]
Elsevier
2005-06-24
[4]
웹사이트
DNA Strands
http://www.sci.sdsu.[...]
2018-05-01
[5]
서적
Biochemistry
W. H. Freeman
[6]
서적
Molecular Biology of the Gene
Pearson
[7]
journal
Eukaryotic core promoters and the functional basis of transcription initiation
2018-10-01
[8]
journal
The Why of YY1: Mechanisms of Transcriptional Regulation by Yin Yang 1
2020-01-01
[9]
journal
Transcription factors: from enhancer binding to developmental control
2012-09-01
[10]
journal
Three-dimensional genome restructuring across timescales of activity-induced neuronal gene expression
2020-06-01
[11]
journal
Long-range enhancer-promoter contacts in gene expression control
2019-08-01
[12]
journal
Enhancers: five essential questions
2013-04-01
[13]
journal
YY1 Is a Structural Regulator of Enhancer-Promoter Loops
2017-12-01
[14]
journal
The Human Transcription Factors
2018-02-01
[15]
journal
Positional specificity of different transcription factor classes within enhancers
2018-07-01
[16]
journal
The Mediator complex: a central integrator of transcription
2015-03-01
[17]
journal
The degree of enhancer or promoter activity is reflected by the levels and directionality of eRNA transcription
2018-01-01
[18]
journal
MAP kinase phosphorylation-dependent activation of Elk-1 leads to activation of the co-activator p300
2003-01-01
[19]
journal
Enhancer RNAs predict enhancer-gene regulatory links and are critical for enhancer function in neuronal systems
2020-09-01
[20]
journal
DNA methylation in human epigenomes depends on local topology of CpG sites
2016-06-01
[21]
journal
Cytosine methylation and CpG, TpG (CpA) and TpA frequencies
2004-05-01
[22]
journal
Pervasive and CpG-dependent promoter-like characteristics of transcribed enhancers
2020-06-01
[23]
journal
DNA methylation patterns and epigenetic memory
2002-01-01
[24]
journal
Methyl-CpG-binding domain proteins: readers of the epigenome
2015-01-01
[25]
journal
A census of human transcription factors: function, expression and evolution
2009-04-01
[26]
journal
EGR1 recruits TET1 to shape the brain methylome during development and upon neuronal activity
2019-08-01
[27]
journal
Genome-wide investigation of in vivo EGR-1 binding sites in monocytic differentiation
2009-01-01
[28]
journal
Neuronal DNA Methyltransferases: Epigenetic Mediators between Synaptic Activity and Gene Expression?
2018-04-01
[29]
journal
Rescue of aging-associated decline in Dnmt3a2 expression restores cognitive abilities
2012-07-01
[30]
journal
The Dnmt3a PWWP domain reads histone 3 lysine 36 trimethylation and guides DNA methylation
2010-08-01
[31]
journal
Isoform-specific localization of DNMT3A regulates DNA methylation fidelity at bivalent CpG islands
2017-12-01
[32]
journal
H3K36me2/3 Binding and DNA Binding of the DNA Methyltransferase DNMT3A PWWP Domain Both Contribute to its Chromatin Interaction
2019-12-01
[33]
journal
Synaptic control of DNA methylation involves activity-dependent degradation of DNMT3A1 in the nucleus
2020-11-01
[34]
서적
Threshold Concepts in Biochemistry
La Trobe eBureau
[35]
journal
Global repositioning of transcription start sites in a plant-fermenting bacterium
[36]
논문
The complexities of eukaryotic transcription initiation: regulation of preinitiation complex assembly
[37]
논문
Direct detection of abortive RNA transcripts in vivo
2009-05-00
[38]
논문
Abortive initiation and productive initiation by RNA polymerase involve DNA scrunching
2006-11-00
[39]
논문
Holoenzyme switching and stochastic release of sigma factors from RNA polymerase in vivo
2005-11-00
[40]
논문
Functional interactions of RNA-capping enzyme with factors that positively and negatively regulate promoter escape by RNA polymerase II
2004-05-00
[41]
논문
Transcription factors IIE and IIH and ATP hydrolysis direct promoter clearance by RNA polymerase II
1994-04-00
[42]
서적
Cell Biology by the Numbers
CRC Press
2015-00-00
[43]
논문
Nucleosomal fluctuations govern the transcription dynamics of RNA polymerase II
2009-07-00
[44]
논문
Nucleosomal arrangement affects single-molecule transcription dynamics
2016-00-00
[45]
논문
Transcriptionally active chromatin recruits homologous recombination at DNA double-strand breaks
2014-04-00
[46]
논문
RNA transcripts stimulate homologous recombination by forming DR-loops
2021-06-00
[47]
논문
Recombinogenic effects of DNA-damaging agents are synergistically increased by transcription in Saccharomyces cerevisiae. New insights into transcription-associated recombination
2003-10-00
[48]
논문
Rho-dependent termination and ATPases in transcript termination
2002-09-00
[49]
논문
Overlapping pathways dictate termination of RNA polymerase II transcription
2007-10-00
[50]
논문
Structural basis of Mfd-dependent transcription termination.
2020-11-18
[51]
논문
Involvement of transcription termination factor 2 in mitotic repression of transcription elongation.
2004-05-07
[52]
논문
The structure and activities of the archaeal transcription termination factor Eta detail vulnerabilities of the transcription elongation complex.
2022-08-09
[53]
논문
DNA damage and transcription stress
2024-01-00
[54]
논문
Structure of Eukaryotic RNA Polymerases
http://dx.doi.org/10[...]
2008-06-00
[55]
웹사이트
8-Hydroxyquinoline from SIGMA-ALDRICH
http://www.sigmaaldr[...]
2022-02-15
[56]
논문
A Glucose-Triptolide Conjugate Selectively Targets Cancer Cells under Hypoxia
[57]
논문
A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters
2006-01-00
[58]
논문
DNA methylation patterns and epigenetic memory
2002-01-00
[59]
논문
Cancer genome landscapes
2013-03-00
[60]
논문
MicroRNAs in the DNA Damage/Repair Network and Cancer
[61]
논문
TNFα signals through specialized factories where responsive coding and miRNA genes are transcribed
2012-11-00
[62]
웹사이트
Chemistry 2006
http://nobelprize.or[...]
2007-03-29
[63]
논문
Kethoxal-assisted single-stranded DNA sequencing captures global transcription dynamics and enhancer activity in situ
2020-04-00
[64]
논문
Nature, nurture, or chance: stochastic gene expression and its consequences
2008-10-00
[65]
서적
Molecular Biology
https://books.google[...]
Elsevier
2005-06-24
[66]
웹사이트
Some patterns of apoptosis mechanism during HIV-infection
http://www.dissercat[...]
2000-00-00
[67]
논문
Alternative lengthening of telomeres: models, mechanisms and implications
2010-05-00
[68]
서적
遺伝子第8版
[69]
서적
ワトソン 遺伝子の分子生物学
[70]
서적
ワトソン 遺伝子の分子生物学
[71]
서적
ワトソン 遺伝子の分子生物学
[72]
서적
ウィーバー 分子生物学第4版
[73]
서적
ワトソン 遺伝子の分子生物学
[74]
서적
ワトソン 遺伝子の分子生物学
[75]
서적
ウィーバー 分子生物学
[76]
서적
ウィーバー 分子生物学
[77]
서적
ワトソン 遺伝子の分子生物学
[78]
서적
ウィーバー 分子生物学
[79]
서적
ウィーバー 分子生物学
[80]
서적
ワトソン 遺伝子の分子生物学
[81]
서적
ワトソン 遺伝子の分子生物学
[82]
문서
二重らせんを形成しているDNAを二本鎖DNA double-stranded DNA、一本だけの状態で遊離しているものを一本鎖DNA single-stranded DNA と呼ぶ。それぞれdsDNA、ssDNAと略す。
[83]
문서
RNAポリメラーゼが転写を実行するためにDNA上に構築する複合体をRNAポリメラーゼホロ酵素もしくは単にホロ酵素と呼ぶ。α1,α2、β、β’、σ、ωの6つのサブユニットから構成され、全体はカニのはさみに似ている。1つで複合体のほぼ半分を占める2つの爪はそれぞれ巨大なβ、β’サブユニットで、この内部を活性中心溝という。転写の活性部位は活性中心溝の奥、爪の付け根に存在する。
[84]
문서
RNAポリメラーゼにはDNA用の出入り口が5つある。NTP取り込み通路は基質であるリボヌクレオチドを触媒活性中心に迎え入れる。RNA出口通路は後の伸長段階で合成したRNA鎖の部分を出すためにある。下流のdsDNAは下流DNA用通路から活性中心溝に入り、ssDNAに分かれる。非鋳型鎖は非鋳型鎖用通路(NT通路)から、鋳型鎖は活性部位を通って鋳型鎖用通路(T通路)から外に出る。2つの一本鎖はホロ酵素の後方で二重らせんに戻る。
[85]
문서
開放型プロモーター複合体形成に伴う、ssDNAの領域を転写バブルという。その大きさについては様々な方法での測定例がある。1978年にTao-shih HsiehとJames Wangは、大腸菌のRNAポリメラーゼはT7ファージの初期プロモーターを約10bpほど分解すると算定した。1979年にウルリッチ・ジーベンリスト Ulrich Siebenlist が行った実験では、観測されたのは12bpだった。ただし、この結果は実際よりも小さい可能性が指摘されている。これらの実験も、それ以前のほかの実験もRNAポリメラーゼとDNAだけが結びついた複合体を対象としている。すなわち、RNA合成前の転写初期におけるRNAポリメラーゼである。
[86]
문서
σサブユニットは転写開始に最も重要で、ホロ酵素をプロモーターへと誘導するなどの必須な役割を持つ。転写開始が転写において最も遅い段階であるため、σ因子は役割を終えると再利用のため直ちにホロ酵素から分離される。そして、別のコア酵素へと供給される。この循環をσサイクルという。
[87]
문서
TRCFはdsDNAに結合すると、 ATPアーゼ活性を活用して転写と同じ5’から3’の方向へ滑るように移動する。停止したRNAポリメラーゼがあれば衝突し、解離させる。損傷の先へ押されて伸長を再開することもある。
[88]
서적
ウィーバー 分子生物学 第4版
化学同人
2008
[89]
서적
ワトソン 遺伝子の分子生物学 第6版
学校法人東京電機大学出版局
2010
[90]
서적
遺伝子 第8版
東京化学同人
2006
[91]
서적
고급생명과학
서울특별시교육청
[92]
서적
HIGH TOP 생명과학2
두산동아
2016-01-12
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com



